L'ATP synthase
L'ATP synthase (ATP synthétase) est une enzyme mitochondriale localisée dans la membrane interne, où elle catalyse la synthèse d'ATP à partir d'ADP et de phosphate. Elle est entraînée par un flux de protons à travers un gradient généré par transfert d'électrons du proton chimiquement positif vers le côté négatif.
Schéma du processus d'ATP synthase :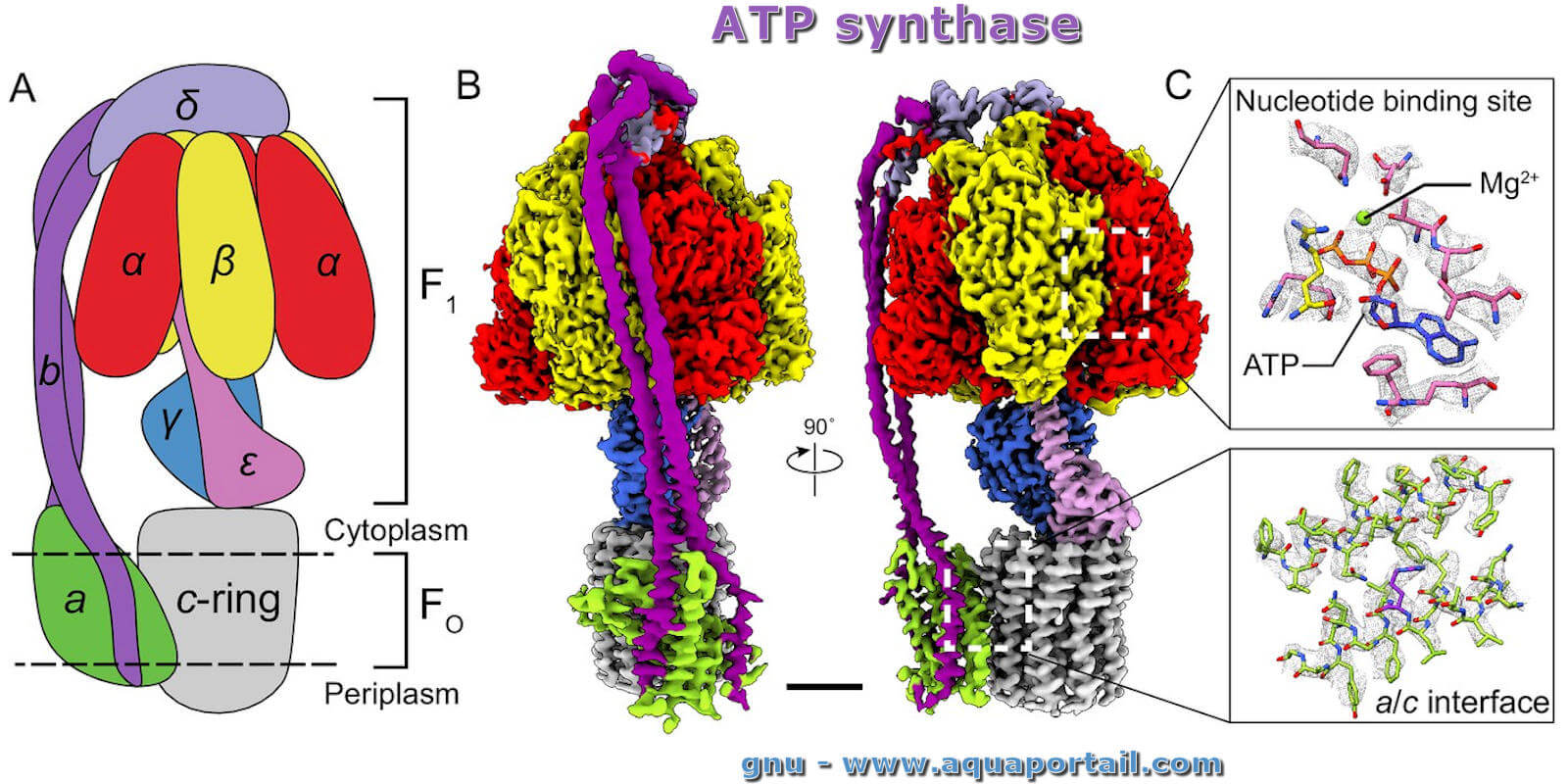
L'ATP synthase produit de l'ATP à partir de l'ADP et du phosphate inorganique avec l'énergie d'une force motrice protonique transmembranaire. Les ATP synthases bactériennes ont été étudiées de manière approfondie parce qu'elles sont la forme la plus simple de l'enzyme et en raison de la facilité relative de manipulation génétique de ces complexes.
Généralités
Une ATP synthase est un complexe enzymatique de 9 chaînes polypeptidiques de la membrane interne des mitochondries et de la membrane des thylakoïdes et des bactéries. C'est un dispositif qui couple la synthèse d'ATP à l'énergie libérée par un flux de protons suivant leur gradient de concentration. L'ATP synthase appartient au groupe EC 7 des translocases.
Le nom synthétase est abandonné au profit de synthase, car il est réservé aux enzymes de la classe 6 (ligases) à laquelle n'appartient par l'ATP synthase. Voir une ATPase et une glutamine (glutamine synthétase).
Le complexe ATP synthase (EC 7.1.2.2) ou complexe V ou F ou F1-ATP synthase (F = facteur de couplage) est une enzyme transmembranaire qui catalyse la synthèse de l'ATP à partir de l'ADP, un groupe phosphate et l'énergie fournie par un flux de protons (H +). Au cours de la respiration cellulaire, la synthèse d'ATP est appelée phosphorylation oxydative et un flux de protons intervient entre l'espace intermembranaire et la matrice mitochondriale. Dans le cas de la photosynthèse, on parle de photophosphorylation et l'enzyme agit sur la lumière et le stroma des chloroplastes.
L'ATP synthase peut être imaginée comme un moteur moléculaire qui produit une grande quantité d'ATP lorsque des protons le traversent. La vitesse de synthèse est élevée, l'organisme humain en phase de repos peut former environ 1021 molécules d'ATP par seconde.
Des expériences in vitro ont montré que l'ATP synthase agit indépendamment de la chaîne de transport d'électrons, l'ajout d'un acide faible (par exemple l'acide acétique) à une suspension de mitochondries isolées est suffisant pour induire la biosynthèse de l'ATP in vitro.
L'ATP synthase a un diamètre de 10 nm et est le plus petit complexe identifié à ce jour. Il fonctionne avec un degré d'efficacité proche de 100 %.
Complexe V
L'ATP synthase (ou F1F0 ATPase et également appelée complexe V) utilise l'énergie libre d'un gradient électrochimique de protons (ou d'ions sodium) généré par la chaîne respiratoire pour synthétiser l'ATP. Les ATP synthases comprennent un très grand groupe d'enzymes hautement conservées que l'on trouve dans les membranes cytoplasmiques bactériennes, les membranes thylacoïdes des chloroplastes et les membranes internes des mitochondries. La plupart des membres du groupe utilisent H+ comme ion de couplage (l'enzyme Propionigenium modestum est un exemple des quelques ATP synthases qui peuvent utiliser Na+ comme ion de couplage physiologique).
Dimère d'ATP synthase
Pendant de nombreuses années, on a supposé que l'ATP synthase et d'autres complexes de conversion d'énergie étaient répartis de manière aléatoire sur la membrane interne. Le premier indice que ce n'est pas le cas est venu de la microscopie électronique à fracture par congélation profonde, qui a révélé des doubles rangées de complexes macromoléculaires dans les crêtes tubulaires de la paramécie ciliée unicellulaire. On pensait que les doubles rangées étaient des réseaux linéaires d'ATP synthase mitochondriale. C'est bien ce qu'ils sont, mais cela n'a pu être démontré sans ambiguïté que plus de 20 ans plus tard par cryo-ET, qui a révélé des rangées de dimères d'ATP synthase dans les mitochondries de toutes les espèces étudiées. Jusque-là, les rangées étaient considérées comme propres à Paramecium.
Les doubles rangées d'ATP synthase dans sept espèces différentes :
Structure et fonction des complexes de protéines membranaires mitochondriales. Doubles rangées d'ATP synthase dans sept espèces différentes. Rangée du haut : tranches de volumes 3D obtenus par cryo-ET avec des rangées de dimères d'ATP synthase. Les encarts montrent des vues latérales des dimères dans la membrane. Les pointes de flèches jaunes indiquent les têtes F1 d'un dimère. Barre d'échelle = 50 nm. Rangée du bas : représentations de surface des moyennes de sous-tomogrammes.
Les réseaux linéaires de dimères d'ATP synthase sont un attribut omniprésent et fondamental de toutes les mitochondries. On les trouve toujours le long des régions les plus étroitement incurvées le long des crêtes de crêtes mitochondriales ou autour de crêtes tubulaires étroites. Les moyennes des sous-tomogrammes indiquent que les dimères des champignons et des mammifères sont indiscernables à basse résolution, alors que ceux des plantes, des algues et des protistes diffèrent par l'angle des dimères ou la position de la tige périphérique par rapport à la tête catalytique F1.
Cependant, l'assemblage de base des complexes d'ATP synthase en dimères et leur association en longues rangées le long des crêtes de la crête sont conservés. Les simulations de dynamique moléculaire à gros grains indiquent que les dimères plient la bicouche lipidique et, par conséquent, s'auto-associent en rangées. En conséquence, la formation de rangées ne nécessite pas d'interactions protéiques spécifiques, mais est entraînée par l'énergie de déformation de la membrane élastique. La cryo-ET de dimères isolés d'ATP synthase solubilisés dans un détergent reconstitués dans des protéoliposomes a fourni des preuves expérimentales que les dimères s'auto-associent en fait en rangées et qu'ils induisent une courbure locale dans la bicouche lipidique. Ce sont donc bien les dimères qui courbent la membrane et non l'inverse.
La structure d'un dimère d'ATP synthase mitochondriale a maintenant été déterminée par cryo-EM à une seule particule à une résolution de 6 à 7 Å. Étonnamment, la structure a révélé une épingle à cheveux de longues hélices α intrinsèques à la membrane dans la sous -unité a à côté de l'anneau du rotor F0, ainsi que des demi-canaux aqueux de chaque côté de la membrane pour la translocation des protons. Les longues hélices semblent jouer un rôle central dans ce processus, car elles sont conservées dans toutes les ATPases rotatives [ 40 ]. Avec les structures à rayons X haute résolution ajustées de la tête catalytique F1 et de l'anneau de rotor dans la membrane, la carte cryo-EM fournit la première image complète de ce complexe membranaire mitochondrial pivot et contribue largement à expliquer son mécanisme
Pompage de protons et synthèse d'ATP
Le complexe I, le complexe III et le complexe IV pompent plusieurs protons dans l'espace intermembranaire pour chaque paire d'électrons qu'ils transportent vers O2. Un nombre suffisant de protons est pompé pour maintenir un gradient de concentration de 10 :1 (une unité de pH) entre l'espace intermembranaire et la matrice.
Le complexe ATP synthase (FoF1-ATP synthase) permet aux protons de refluer dans la matrice et utilise le changement d'énergie libre de ce processus pour synthétiser l'ATP à partir d'ADP et de phosphate inorganique (Pi). Il est situé dans des structures en forme de bouton incrustées dans les crêtes (invaginations de la membrane mitochondriale interne) et s'étendant dans la matrice.
La protéine Fo (le "o" dans Fo fait référence à sa sensibilité à l'oligomycine, un poison qui bloque le flux de protons) s'étend à travers la membrane mitochondriale interne et sert de canal protonique entre l'espace intermembranaire et la matrice.
L'ATP synthase (F1-ATPase) est attachée à la protéine Fo à l'intérieur de la matrice. L'ATP synthase utilise les protons circulant dans la matrice pour lier l'ADP et le Pi et libérer l'ATP. La F1-ATPase est nommée par la réaction inverse qu'elle catalyse lorsqu'elle est isolée des mitochondries et donc découplée du gradient de protons.
Les points-clés sur la chaîne de transport des électrons :
- La chaîne de transport d'électrons est située dans la membrane interne mitochondriale et contient plusieurs types différents de transporteurs d'électrons : mononucléotide de flavine, protéines fer-soufre, coenzyme Q, cytochromes contenant de l'hème et ions cuivre.
- Trois grands complexes multiprotéiques servent de pompes à protons en exploitant l'énergie du flux d'électrons à travers l'ETC vers l'oxygène; à son tour, l'énergie chimiosmotique dans le gradient de protons créé par les pompes est couplée à la synthèse d'ATP par le complexe ATP synthase.
- L'ATP régule sa propre synthèse et le flux d'électrons par le contrôle respiratoire; si la synthèse d'ATP ralentit, le transport d'électrons ralentit et vice versa.
- Le NADH cytosolique ne peut pas traverser la membrane mitochondriale, il transporte donc ses électrons à travers la navette phosphate de glycérol et la navette malate-aspartate.
- L'ATP et l'ADP sont transportés en échange l'un de l'autre par la translocase ATP/ADP.
La protéine inhibitrice de l'ATP synthase IF1
L'ATP synthase est librement réversible, et sa direction dépend de l'équilibre thermodynamique entre Δp et la matrice ΔGp. Des dommages à la chaîne de transport d'électrons, une fuite accrue de protons ou une hypoxie sévère peuvent abaisser Δp de sorte que l'ATP synthase s'inverse dans la cellule et commence à hydrolyser l'ATP cytoplasmique généré par la glycolyse. Expérimentalement, cette inversion peut être détectée comme une diminution de Δψm lors de l'ajout de l'oligomycine, un inhibiteur de l'ATP synthase.
Dans ces conditions, la glycolyse est appelée à desservir non seulement la totalité de la demande d'ATP de la cellule, mais également l'inversion de la synthase. L'un des résultats de ceci est que les cellules peuvent épuiser leur ATP cytoplasmique dans la mesure où la glycolyse et l'oxydation des acides gras, qui nécessitent toutes deux de l'ATP, ne peuvent pas se poursuivre et la cellule meurt. Cette condition est également abordée dans de nombreuses expériences publiées dans lesquelles des protonophores sont ajoutés aux cellules, auquel cas l'hydrolyse mitochondriale de l'ATP peut être extrêmement rapide, n'étant plus limitée par la faible perméabilité aux protons de la membrane interne. Bien que la déplétion en ATP puisse être atténuée in vitro par l'ajout d'oligomycine, un mécanisme physiologique plus subtil existe dans de nombreuses cellules, médié par la protéine inhibitrice de 10 kDa (IF1).
IF1 peut se lier à la F1-ATP synthase dans des conditions de pH acide de la matrice, inhibant partiellement son activité catalytique. Au niveau moléculaire, des études sur l'ATP synthase d'Escherichia coli suggèrent que l'IF1 agit comme un "cliquet" (effet cliquet) empêchant l'inversion de l'enzyme.
Parce qu'une matrice acide n'est normalement observée que dans des conditions hypoxiques, lorsque la voie de transport des électrons est inhibée, ou en présence d'un protonophore, cela signifie essentiellement que IF1 peut inhiber l'inversion de l'ATP synthase mais est sans effet sur la synthèse de l'ATP (lorsque la matrice est alcalin). L'inhibition n'est pas complète mais dépend du rapport entre IF1 et le complexe ATP synthase et peut jouer un rôle important dans la limitation de la déplétion en ATP dans l'hypoxie. Les neurones possèdent généralement des rapports plus élevés de IF1 à F1 que les astrocytes, avec pour résultat que l'inhibition de la chaîne électronique provoque une dépolarisation plus profonde du premier tout en ralentissant la déplétion de l'ATP cytoplasmique.
En rapport avec "ATP synthase"
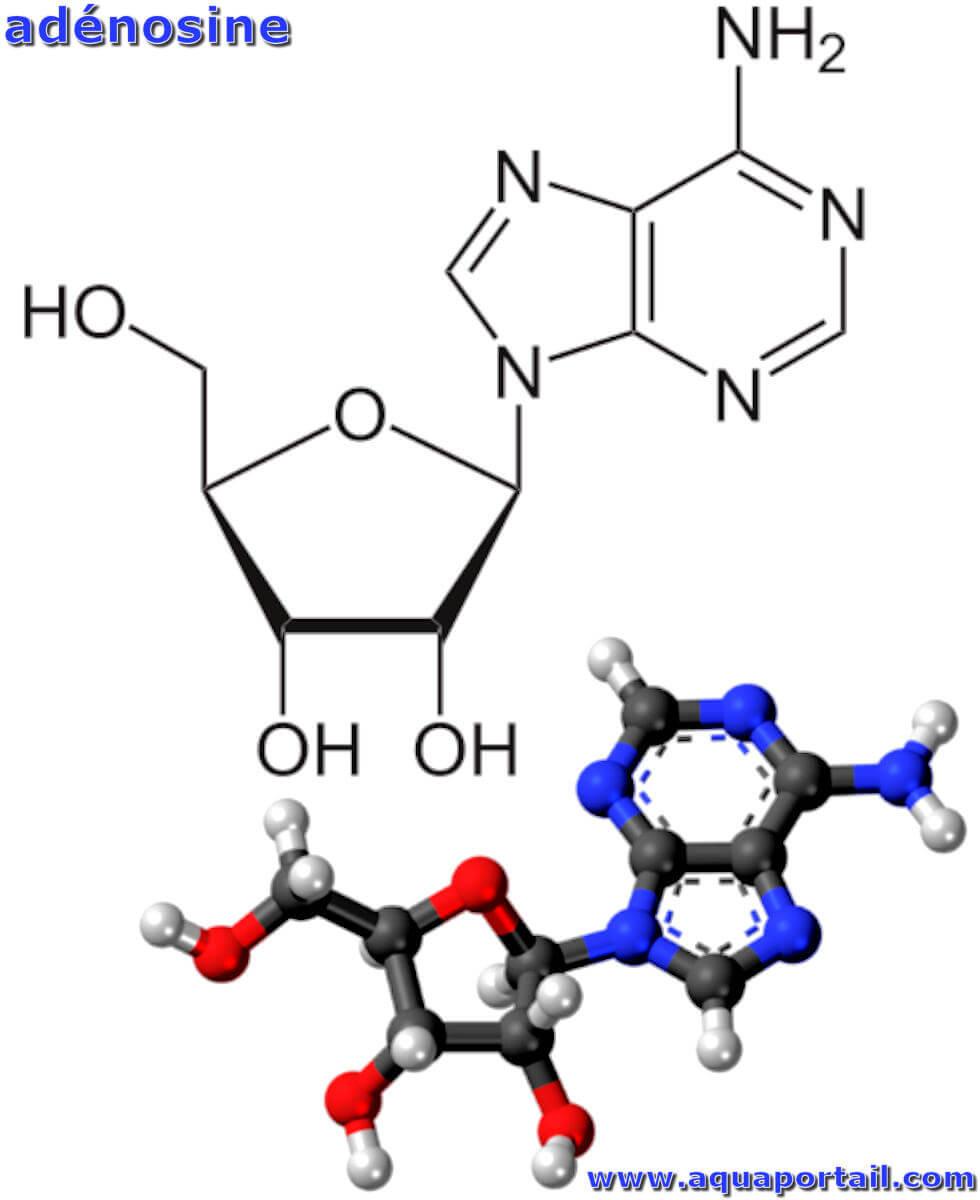
L'adénosine est un nucléoside formé par la liaison de l'adénine à un anneau de ribose (ribofuranose) par une liaison bêta-N₉ glycosidique.

La NO-synthase (oxyde nitrique synthase, NOS) est le système enzymatique qui catalyse la synthèse de NO (oxyde nitrique) à partir d'arginine, de NADPH...

Une synthase est une enzyme qui catalyse une synthèse non couplée à la rupture d'une liaison à haut potentiel énergétique comme le fait une synthétase.

Une translocase est une enzyme qui catalyse le mouvement des molécules ou des ions à travers les membranes cellulaires en général.
