Interactomique
Définition
L'interactomique est l'étude de l'ensemble des interactions entre différentes biomolécules, et notamment les protéines, dans un environnement donné. Elle regroupe la génomique, la transcriptomique, la protéomique et la métabolomique. La discipline, à l'intersection de la bioinformatique et de la biologie, étudie l'interactome protéine-protéine.
L'interactomique :
L'interactomique étudie à la fois les interactions et les conséquences de ces interactions entre et parmi les protéines et d'autres molécules au sein d'une cellule. Au total, il comprend plus d'un quart de million d'interactions dérivées de ressources expérimentales et de prédictions informatiques. Les noeuds et les arêtes du graphique affiché représentent les protéines et leurs interactions.
Explications
L'étude du réseau complexe d'interactions qui relient les molécules biologiques dans une cellule fait l'objet de l'interactomique, actuellement l'un des domaines les plus dynamiques de la biologie moléculaire.
L'interactomique fonctionnelle développe des technologies pour cartographier les réseaux d'interaction protéique, les complexes protéiques et leur régulation post-traductionnelle. Les données sont appliquées pour mieux comprendre la signalisation du carbone et de l'azote en relation (par exemple) avec la croissance des plantes, la résilience au stress et le rendement des cultures, dans le but de s'adapter ou d'atténuer le changement climatique. Les fonctions biologiques sont généralement accomplies par des interactions moléculaires à court terme et à long terme.
Intérêt
La modularité et la dynamique sous-tendent la fonctionnalité des réseaux cellulaires, allant de la régulation transcriptionnelle à la signalisation cellulaire. Les innovations technologiques dans la génération de données et les méthodes de calcul peuvent faire progresser considérablement notre compréhension. De plus, l'intégration des données actuellement disponibles provenant de diverses sources nous aide à acquérir une compréhension plus précise et complète des processus cellulaires. Actuellement, la qualité des données et la couverture des ensembles de données à haut débit imposent des limites à la déduction de réseaux précis. De nombreuses méthodes de calcul utilisées pour analyser les systèmes biologiques n'utilisent pas pleinement les données disponibles et/ou font des hypothèses solides qui pourraient ne pas être réalistes. Avec les progrès vers la résolution de ces problèmes, les phénotypes et les comportements des cellules pourraient potentiellement être prédits avec une plus grande confiance,
L'intégration d'une variété d'ensembles de données, y compris les interactions binaires, les complexes protéiques et les profils d'expression permet l'identification de sous-réseaux qui sont actifs dans certaines conditions.
Les protéines interagissent les unes avec les autres de manière très spécifique et les interactions protéiques jouent un rôle clé dans de nombreux processus cellulaires; en particulier, la distorsion des interfaces protéiques peut conduire au développement de nombreuses maladies. Pour comprendre les mécanismes de reconnaissance des protéines au niveau moléculaire et démêler l'image globale des interactions protéiques dans la cellule, différentes techniques expérimentales ont été développées. Certaines méthodes caractérisent les interactions protéiques individuelles tandis que d'autres sont avancées pour le dépistage des interactions à l'échelle du génome. Différentes techniques expérimentales d'identification des interactions protéiques ainsi que diverses bases de données tentent de classer le large éventail de données expérimentales.
Les progrès récents dans les méthodes expérimentales à haut débit pour l'identification des interactions protéiques ont abouti à une grande quantité de données diverses qui sont quelque peu incomplètes et contradictoires. Aussi précieuses soient-elles, ces approches expérimentales étudiant les interactions protéiques présentent certaines limites qui peuvent être complétées par les méthodes de calcul pour prédire les interactions protéiques. Prédire les partenaires d'interaction protéique et mettre en évidence les réalisations récentes dans la prédiction de domaines spécifiques médiant les interactions protéine-protéine demande des méthodes de calcul à différents types de problèmes de prédiction, avec ses limites.
La représentation en réseau des entités biologiques est très importante pour comprendre les processus biologiques, l'organisation du système et l'interaction entre les entités.
Interactomique comparée
Le comportement, la morphologie et la réponse aux stimuli dans les systèmes biologiques sont dictés par les interactions entre leurs composants. Ces interactions, telles que nous les observons actuellement, sont donc façonnées par des variations génétiques et des pressions sélectives. À l'instar de ce qui a été réalisé en comparant les structures génomiques et les séquences protéiques, des informations précieuses sont obtenues sur l'évolution des systèmes en comparant l'organisation des réseaux d'interaction et en analysant leur variation et leur conservation.
De même, de manière significative, les informations de réseau obtenues expérimentalement dans des systèmes modèles sont bien caractérisées à différents organismes. Malgré l'achèvement récent de plusieurs expériences à haut débit visant à la description d'interactomes complets, les informations d'interaction disponibles ne sont pas encore d'une couverture et d'une qualité suffisantes pour tirer une conclusion biologiquement significative de la comparaison de différents interactomes.
Ainsi, le transfert d'informations de réseau obtenues à partir d'un organisme simple vers des espèces évolutives distantes doit être effectué et considéré avec prudence. En utilisant des ensembles de données plus petits et plus fiables, une plus grande fraction d'interactions est conservée. Cela suggère qu'avec le développement d'approches expérimentales et informatiques plus précises, les chercheurs seront bientôt en mesure d'étudier l'évolution du réseau grâce à l'interactomique.
Identification des interactions protéiques
Pour l'identification en vrac des partenaires d'interaction protéique, la technique la plus couramment utilisée est la purification par co-immunoprécipitation/co-affinité avec la spectrométrie de masse quantitative. Pour cela, la protéine d'intérêt est précipitée des cellules avec ses protéines en interaction, qui sont ensuite identifiées par spectrométrie de masse. Étant donné que ces précipités contiendront également des contaminants (par exemple, des liants non spécifiques aux billes d'affinité, billes d'IgG, anticorps...), un échantillon de contrôle ne contenant pas l'appât est nécessaire.
En fin de compte, les vrais interacteurs peuvent être distingués des contaminants en regardant leurs abondances respectives dans les deux échantillons. Alors que les contaminants sont également présents dans les deux échantillons, des interacteurs spécifiques sont enrichis dans l'échantillon d'appât. Pour déterminer les abondances de protéines, différentes stratégies de quantification sont disponibles.
Pan-interactomique
Les activités cellulaires sont le résultat de la série de communications biochimiques dans le web séquentiel. L'étude sommaire de l'ensemble des réseaux d'interactions moléculaires est appelée interactomique. La pan-interactomique représente l'interaction du réseau de biomolécules et leur ultra-régulation consécutive des voies métaboliques dans la cellule.
L'envie de comprendre l'interaction globale a incité les chercheurs à l'explorer plus en détail. Les progrès de l'instrumentation et des techniques sophistiquées avec la spectroscopie de masse, imagerie par résonance plasmonique de surface (SPR), lactamase fractionnée, protéine fluorescente jaune fractionnée ou YFP fractionnée, variantes de la levure à deux hybrides, ont facilité le développement de différentes bases de données interactives.
Des bases de données interactomiques d'organismes acellulaires à multicellulaires ont été développées et sont placées dans le domaine public. La mise en place de différentes bases de données d'interactomique et les résultats significatifs de projets de séquençage du génome entier ont collectivement contribué au développement le plus récent dans le domaine de l'interactomique, c'est-à-dire l'interactomique prédite. L'interactome prédit repose sur la prédiction d'orthologues en interaction. L'interactomique joue un rôle important dans la découverte de mécanismes moléculaires indéfinis, la prédiction de maladies, la découverte de médicaments et une meilleure compréhension de l'évolution parallèle.
Les "micros" d'interaction ont joué un rôle important dans le dévoilement des mécanismes moléculaires sous-déterminés, la prévision de la pathogénicité, la compréhension de l'état physiologique altéré en réponse à divers stimuli, les découvertes et l'évaluation de l'efficacité et de la précision des nouveaux médicaments. interactomiques a également fonctionné comme un outil pour comprendre le modèle d'évolution conservée. Différentes méthodes de cartographie de l'interactome, des bases de données interactomiques, ainsi que de leur évolution et de leurs applications dans différents mécanismes de régulation au niveau moléculaire sont de plus en plus développées.
Synonymes, antonymes
Voir tous les synonymes pour "interactomique".5 synonymes (sens proche) de "interactomique" :
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à INTERACTOMIQUE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot INTERACTOMIQUE est dans la page 2 des mots en I du lexique du dictionnaire.
Mots en I à proximité
interaction négative interaction protéique interactions de van der Waals interactions trophiques interactome interactomiqueinterambulacre intercalaireintercellulaire intercinèse interculture
En rapport avec "interactomique"
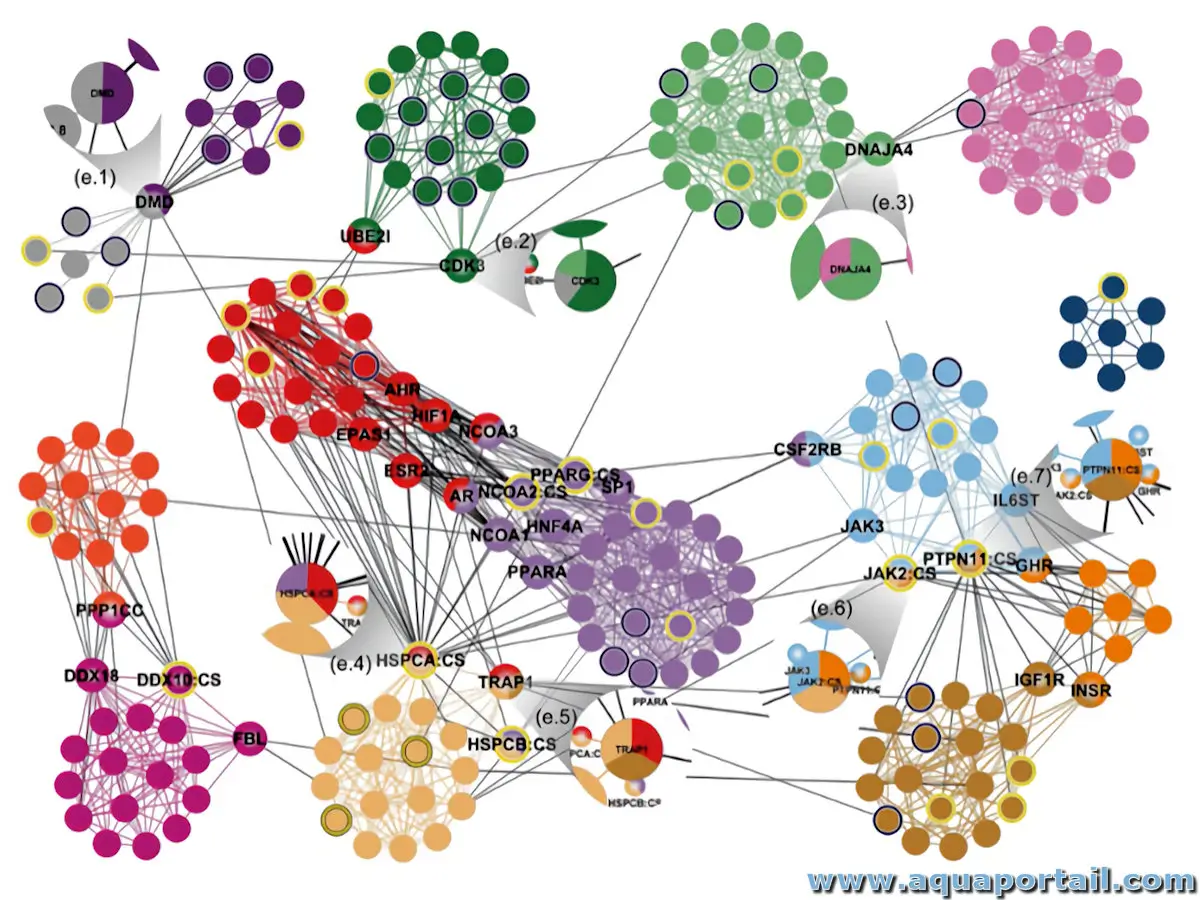
Un interactome est l'ensemble des interactions moléculaires dans une cellule individuelle ou un tissu.
La génomique est l'étude des gènes et de leurs fonctions, une discipline qui étudie le génome et ses applications.
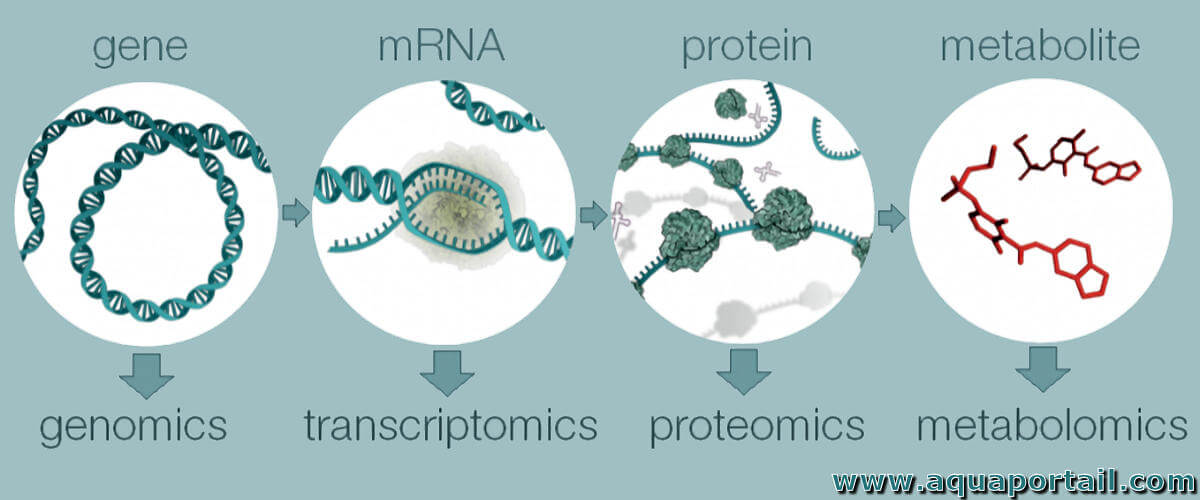
La métabolomique est l'étude à grande échelle de petites molécules, communément appelées métabolites, dans les cellules, les biofluides, les tissus...

La protéomique est l'étude et l'analyse systémique à grande échelle de chaque protéine, en particulier de leurs structures, fonctions, activité, quantité...
