Interactome
Définition
Un interactome est l'ensemble des interactions moléculaires dans une cellule individuelle ou un tissu. Il comprend à la fois les contacts physiques directs entre les protéines (interactions protéine-protéine) et les interactions géniques indirectes (épistase). Les relations d'interactions dans un interactome comprennent celles entre génome, transcriptome, protéome et métabolome. Son étude est faite en biologie moléculaire dans la discipline appelée l'interactomique.
Schéma d'un interactome :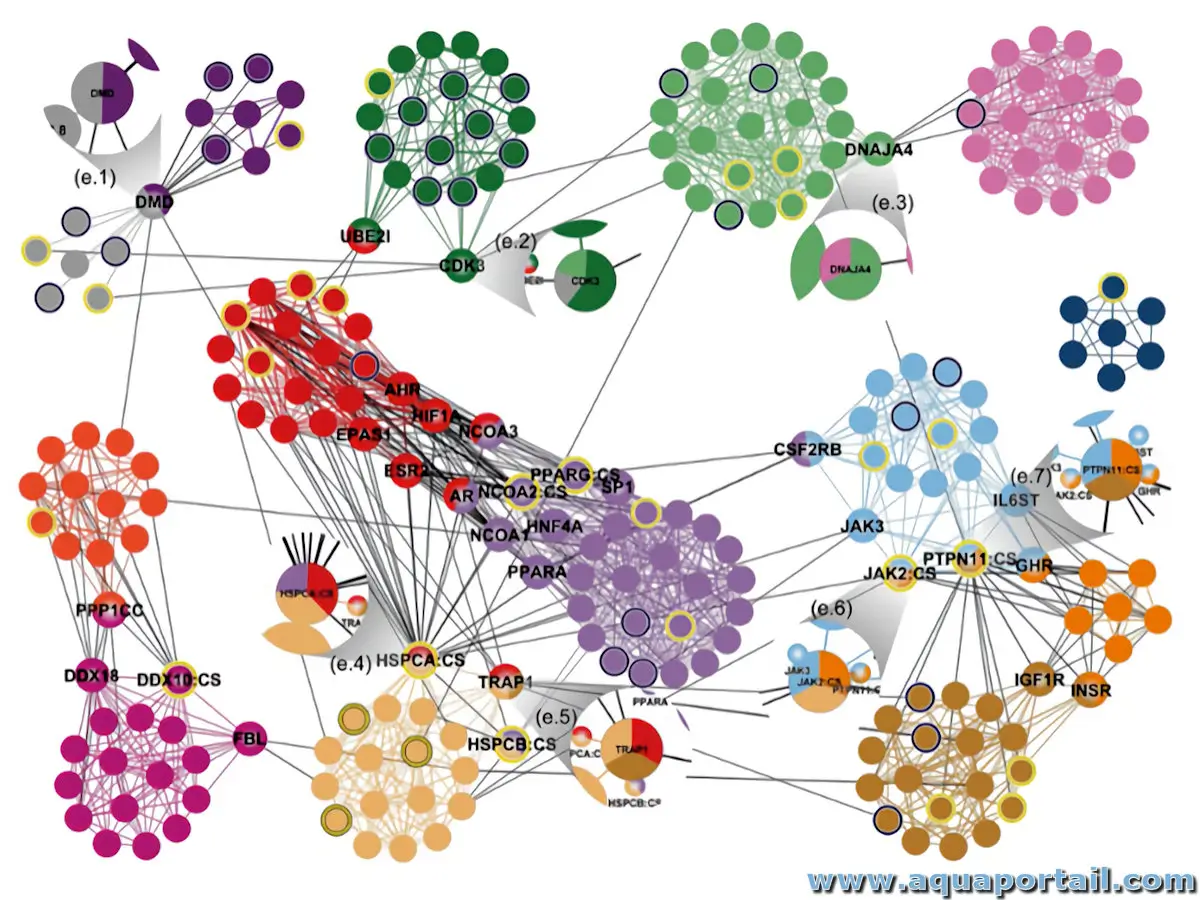
L'interactome est une carte détaillée des interactions moléculaires fonctionnelles au sein d'une cellule ou d'un organisme. Il met souvent en évidence des liens essentiels entre des molécules qui, de prime abord, ne semblent pas liées. Défini comme l'ensemble des interactions physiques et génétiques d'une cellule, il est généralement visualisé sous la forme d'un graphe de réseau biologique.
Explications
Le mot interactome a été inventé à l'origine en 1999 par un groupe de scientifiques Français dirigé par Bernard Jacques. D'un point de vue mathématique, les interactomes sont le plus souvent représentés sous forme de graphiques. Bien que les interactomes soient des réseaux biologiques, ils ne doivent pas être confondus avec d'autres réseaux biologiques, tels que les réseaux neuronaux et trophiques.
Les connexions entre les produits géniques peuvent être cartographiées à l'aide d'enquêtes à l'échelle du génome des partenaires de liaison aux protéines. L'univers des interactions protéine-protéine est connu sous le nom d'interactome. Deux technologies largement utilisées pour cartographier cet espace sont le criblage à deux hybrides et la purification par co-affinité combinées à la détection.
Les interactions protéine-protéine jouent un rôle vital dans les fonctions cellulaires. Les interactions protéine-protéine facilitent la majorité des processus biologiques, y compris la communication de cellule à cellule qui forme un réseau complexe appelé interactome. Environ 650 000 réseaux interactifs protéine-protéine ont été identifiés dans plusieurs organismes (dont environ 250 000 chez l'humain). La modulation de ces interactions ouvrirait la voie à la régulation de ces processus biologiques dans des conditions pathologiques et, par conséquent, leur caractérisation gagne en importance.
Des régions distinctes de l'interactome sont couvertes par différentes méthodes indiquant les avantages potentiels de l'intégration en interactomique. Habituellement, des estimations amplifiées sont obtenues lorsque l'on considère des interactions indirectes ou prédites, souvent en utilisant une combinaison de chromatographie d'affinité et de spectrométrie de masse.
Il a été suggéré que la taille de l'interactome d'un organisme est mieux corrélée avec la complexité biologique d'un organisme que la taille d'un génome. Cependant, les cartes des interactions protéine-protéine, contenant plusieurs milliers d'interactions binaires et disponibles pour plusieurs espèces, sont reconstituées chaque année et, apparemment, ne sont pas finalement terminées, de sorte que cette hypothèse peut encore être réfutée.
Les gènes interagissent de telle sorte qu'ils affectent les fonctions de chacun. Par exemple, une seule mutation peut être inoffensive, mais sa combinaison avec une autre mutation peut être mortelle. On dit que ces gènes sont "génétiquement en interaction". Les gènes ainsi liés forment des réseaux d'interactions génétiques. Les objectifs de l'étude de ces réseaux comprennent le développement de cartes fonctionnelles des processus dans la cellule, l'identification de cibles médicamenteuses et la prédiction des fonctions de gènes non caractérisés.
Exemple et propriétés
Des interactions moléculaires peuvent exister entre des molécules de différents groupes de produits biochimiques (protéines, acides nucléiques, lipides, glucides), ainsi qu'au sein d'un même groupe.
Exemples d'interactomes :
Ces interactomes montrent les interactions moléculaires entre le domaine C-terminal distal du récepteur cannabinoïde CB1 et les protéines interagissant avec le récepteur cannabinoïde (CRIP1a/CRIP1b). Légende : C-terminal CB1R (magenta), CRIP1a (bleu) et CRIP1b (vert). L'approche de l'interactome peut également être appliquée aux interactions miARN-cible. Dans ce cas, les noeuds représentent les miARN et leurs cibles, tandis que les bords représentent les interactions miARN-cible.
Par exemple, l'interactome de la protéine sirt-1 est un réseau qui comprend Sirt-1 et des protéines qui interagissent directement avec lui, et l'interacteur de second ordre de la famille Sirt illustre également les interactions entre voisins. Un autre type d'interactome largement étudié, l'interactome de la protéine ADN (également appelé réseau de régulation des gènes), est un réseau formé par des facteurs de transcription, des protéines régulatrices de la chromatine et leurs gènes cibles. Même les réseaux métaboliques peuvent être considérés comme des réseaux d'interactions moléculaires : les métabolites, c'est-à-dire les composants chimiques d'une cellule, sont convertis les uns dans les autres par des enzymes qui doivent se lier physiquement à leurs substrats.
Tous les types d'interactomes sont interdépendants et se chevauchent. Par exemple, les interactomes protéiques contiennent de nombreuses enzymes, qui à leur tour forment des réseaux biochimiques. De même, les réseaux de régulation des gènes se croisent avec des réseaux d'interactions protéiques et avec des réseaux de signalisation.
Interactomes viraux (viromes)
Les viromes sont les interactomes viraux protéiques qui contiennent des interactions entre les protéines de virus ou de phages. Ces interactomes ont été parmi les premiers à être étudiés, car les génomes des virus et des phages sont petits et toutes les protéines peuvent être analysées à peu de frais. Les interactomes viraux sont liés aux interactions de leurs hôtes, formant des réseaux d'interactions virus-hôte.
Synonymes, antonymes
Voir tous les synonymes pour "interactome".6 synonymes (sens proche) de "interactome" :
- génome
- métabolome
- phylogénome
- Voir la suite...
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à INTERACTOME sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot INTERACTOME est dans la page 2 des mots en I du lexique du dictionnaire.
En rapport avec "interactome"

L'interactomique est l'étude de l'ensemble des interactions entre différentes biomolécules, et notamment les protéines, dans un environnement donné.
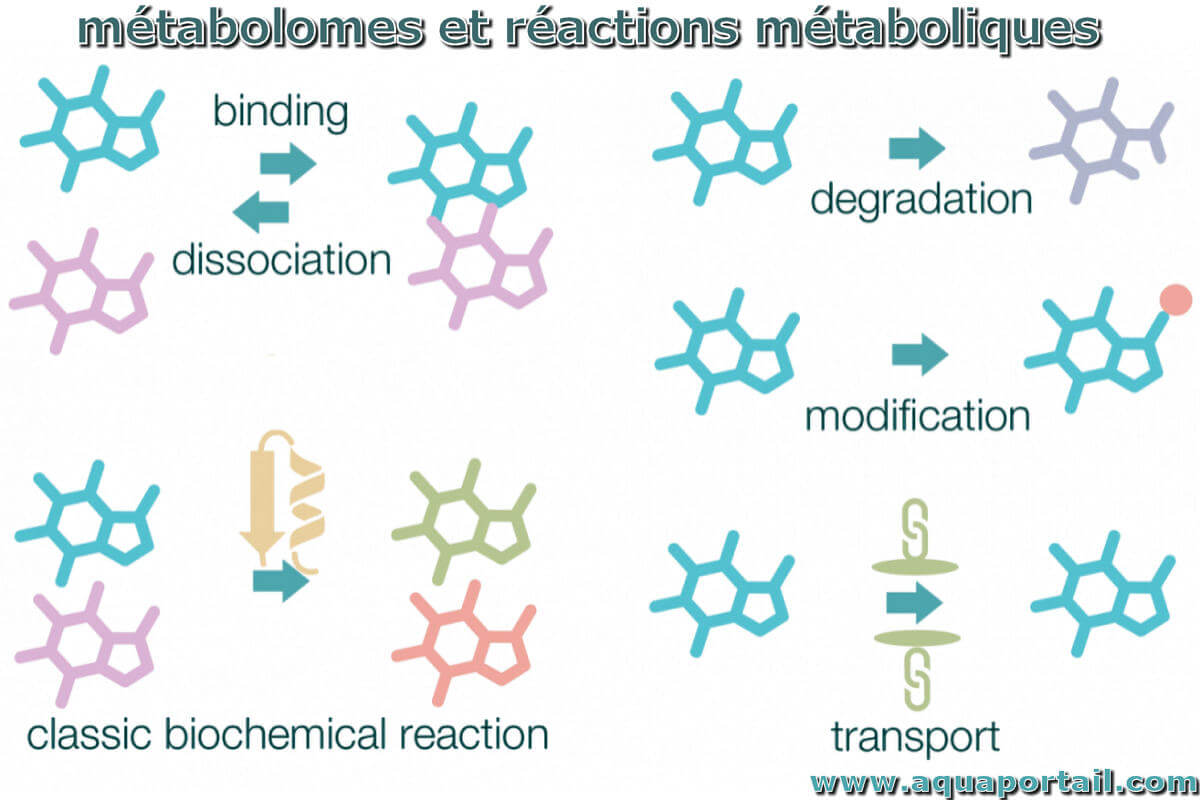
Le métabolome est l'ensemble complet de métabolites dans une cellule, un tissu ou un échantillon biologique à un moment donné.
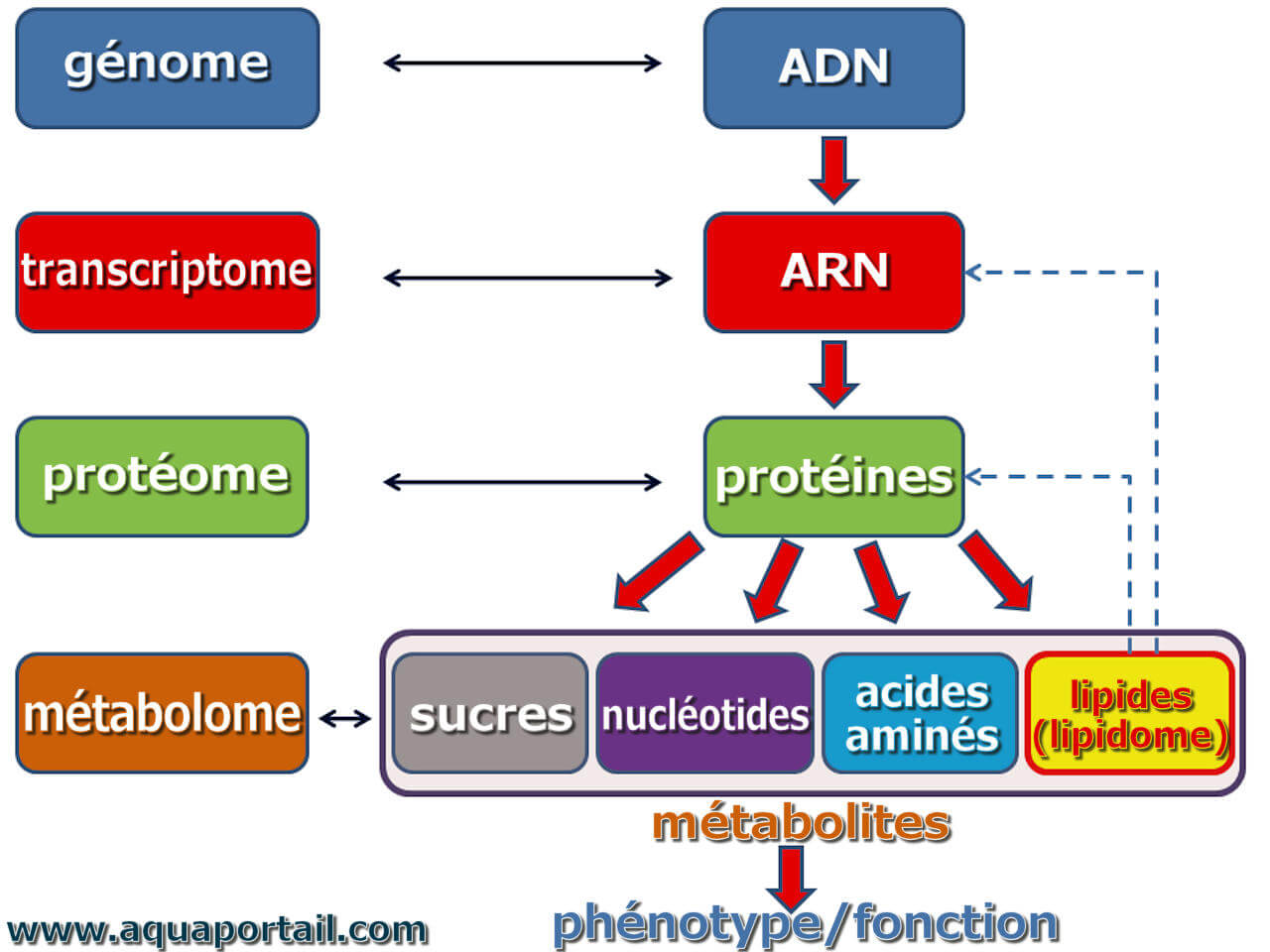
Le protéome cellulaire est l'ensemble de toutes les protéines d'un être vivant, un tissu, une cellule ou un compartiment cellulaire, dans des conditions...
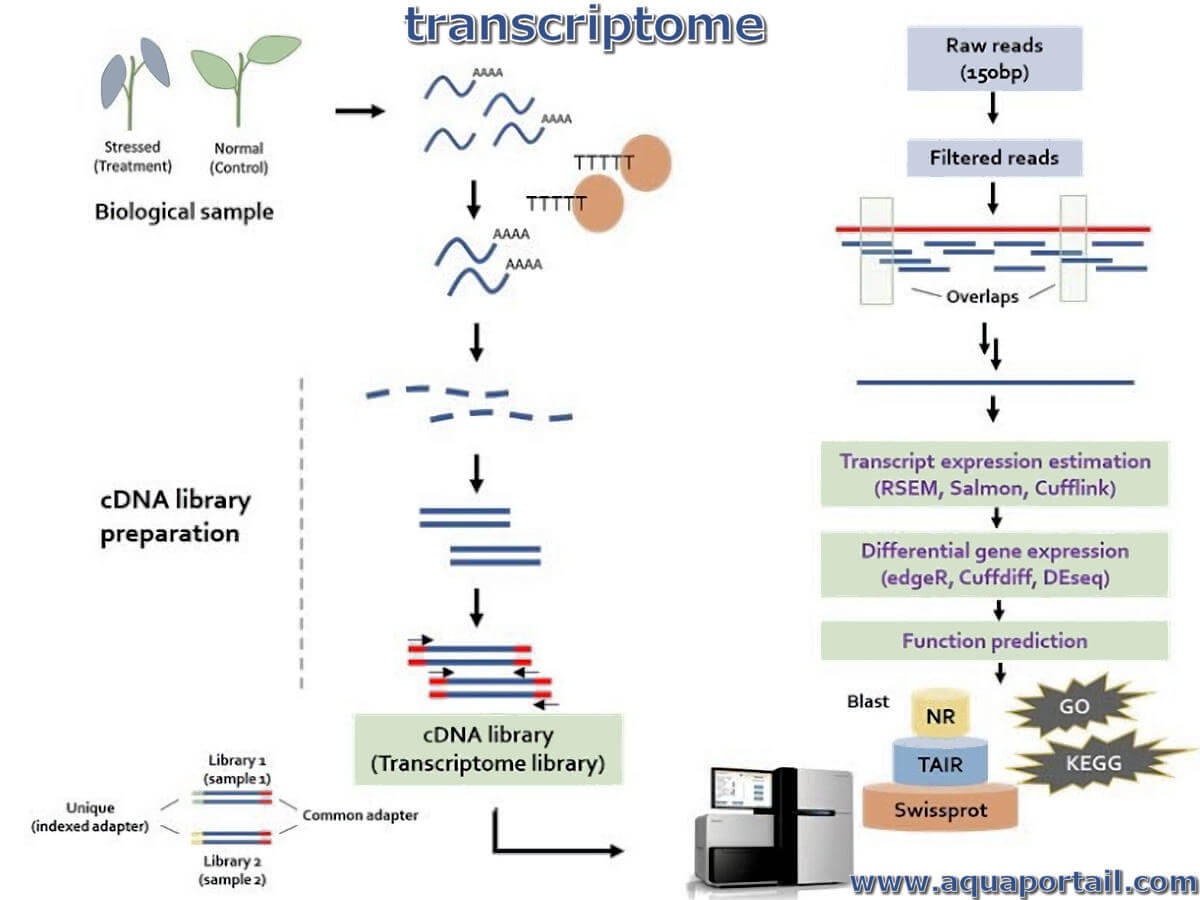
Le transcriptome est l'ensemble des transcrits d'ARN synthétisés par une cellule ou un tissu entier, incluant les ARNm et les ARN non codants.
