Transcriptomique
Définition
La transcriptomique est la discipline de l'analyse du transcriptome, de l'ensemble complet des transcrits d'ARN produits par le génotype à un instant donné qui fait le lien entre le génome, le protéome et le phénotype cellulaire. Intégrée dans l'interactomique, la transcriptomique est une approche globale qui, avec la génomique, la protéomique et la métabolomique, est en constante évolution depuis le début du 21ème siècle.
La transcriptomique (entre la génomique et la protéomique) :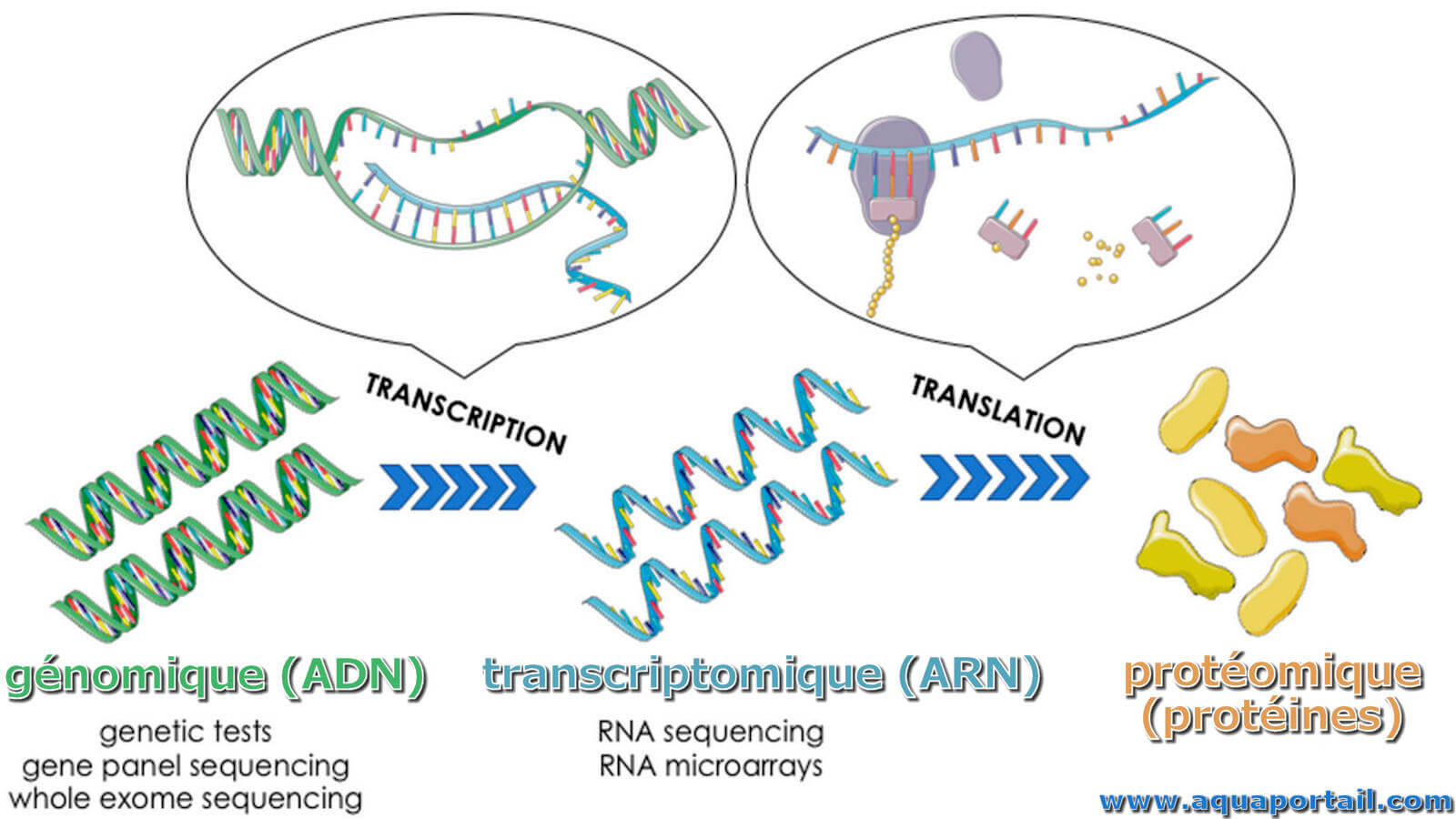
La transcriptomique appartient à différentes approches de l'expression génique, entre la génomique et la protéomique.
Explications
La transcriptomique est également connue sous le nom de profilage d'expression, car il s'agit d'une étude des niveaux d'expression des ARNm dans une population cellulaire donnée. La première utilisation du mot transcriptomique, pour désigner un ensemble complet de transcriptions, a été attribuée à Charles Auffray en 1999. Le terme transcriptome est désormais largement compris comme l'ensemble complet de toutes les molécules d'acide ribonucléique (ARN) exprimées dans une entité donnée, telle qu'une cellule, un tissu ou un organisme.
En microbiologie alimentaire, la transcriptomique a trouvé une application pour comprendre le comportement microbien dans différentes conditions environnementales. Il est également intéressant de comprendre la réponse globale de l'ensemble de la communauté microbienne d'un aliment. Avec l'utilisation de la métatranscriptomique, il est possible d'analyser le transcriptome collectif de la communauté microbienne présente dans un aliment.
La transcriptomique englobe tout ce qui concerne les ARN. Cela inclut leurs niveaux de transcription et d'expression, leurs fonctions, leurs emplacements, leur trafic et leur dégradation. Il comprend également les structures des transcrits et de leurs gènes parents en ce qui concerne les sites de départ, les séquences d'extrémité 5′ et 3′, les modèles d'épissage et les modifications post-transcriptionnelles. La transcriptomique couvre tous les types de transcrits, y compris les ARN messagers (ARNm), les microARN (miARN) et différents types d'ARN longs non codants (ARNlnc).
La transcriptomique moderne utilise des méthodes à haut débit pour analyser l'expression de plusieurs transcrits dans différentes conditions physiologiques ou pathologiques, ce qui élargit rapidement notre compréhension des relations entre le transcriptome et le phénotype dans un large éventail d'entités vivantes.
Les grandes orientations des études transcriptomiques sont :
- caractériser différents états des cellules (c'est-à-dire les stades de développement), des tissus ou des phases du cycle cellulaire par des modèles d'expression;
- explorer les mécanismes moléculaires sous-jacents à un phénotype;
- identifier des biomarqueurs exprimés différemment entre l'état malade et l'état sain;
- distinguer les stades ou les sous-types de la maladie (par exemple, les stades du cancer);
- établir la relation causale entre les variants génétiques et les modèles d'expression génique pour éclairer l'étiologie des maladies.
Métatranscriptomique
La métatranscriptomique est l'analyse du transcriptome microbien cultivable (ou non) par séquençage à grande échelle et à haut débit des transcrits de toutes les communautés microbiennes dans des échantillons environnementaux spécifiques. Le séquençage métatranscriptomique offre la possibilité de séquencer de manière aléatoire les ARNm en tant qu'unité pour comprendre la régulation des processus complexes dans les communautés microbiennes. L'étude du métatranscriptome grâce aux techniques de séquençage de nouvelle génération nous permet d'obtenir des profils d'expression génique à partir de populations microscopiques entières, offrant de nouvelles informations sur des systèmes biologiques mal connus et surmontant les limitations techniques liées à l'isolement des bactéries individuelles.
Bien que les techniques métatranscriptomiques actuelles soient prometteuses, il existe encore plusieurs obstacles qui limitent leur application à grande échelle. Premièrement, une grande partie de l'ARN récolté provient de l'ARN ribosomal (ARNr), et son abondance dominante peut réduire considérablement la couverture de l'ARNm, qui est l'objectif principal des études transcriptomiques. Pour surmonter cela, certains efforts ont été faits pour éliminer efficacement l'ARNr. Deuxièmement, l'ARNm est notoirement instable, compromettant l'intégrité de l'échantillon avant le séquençage. Troisièmement, la différenciation entre l'ARN hôte et microbien peut être difficile, bien que des kits d'enrichissement commerciaux soient disponibles. Cela peut également être fait in silico si un génome de référence est disponible pour l'hôte, comme dans les travaux de Perez-Losada et al. qui ont examiné l'impact des interactions hôte-pathogène sur le microbiome des voies respiratoires humaines. Enfin, les bases de données de référence du transcriptome sont limitées par leur couverture.
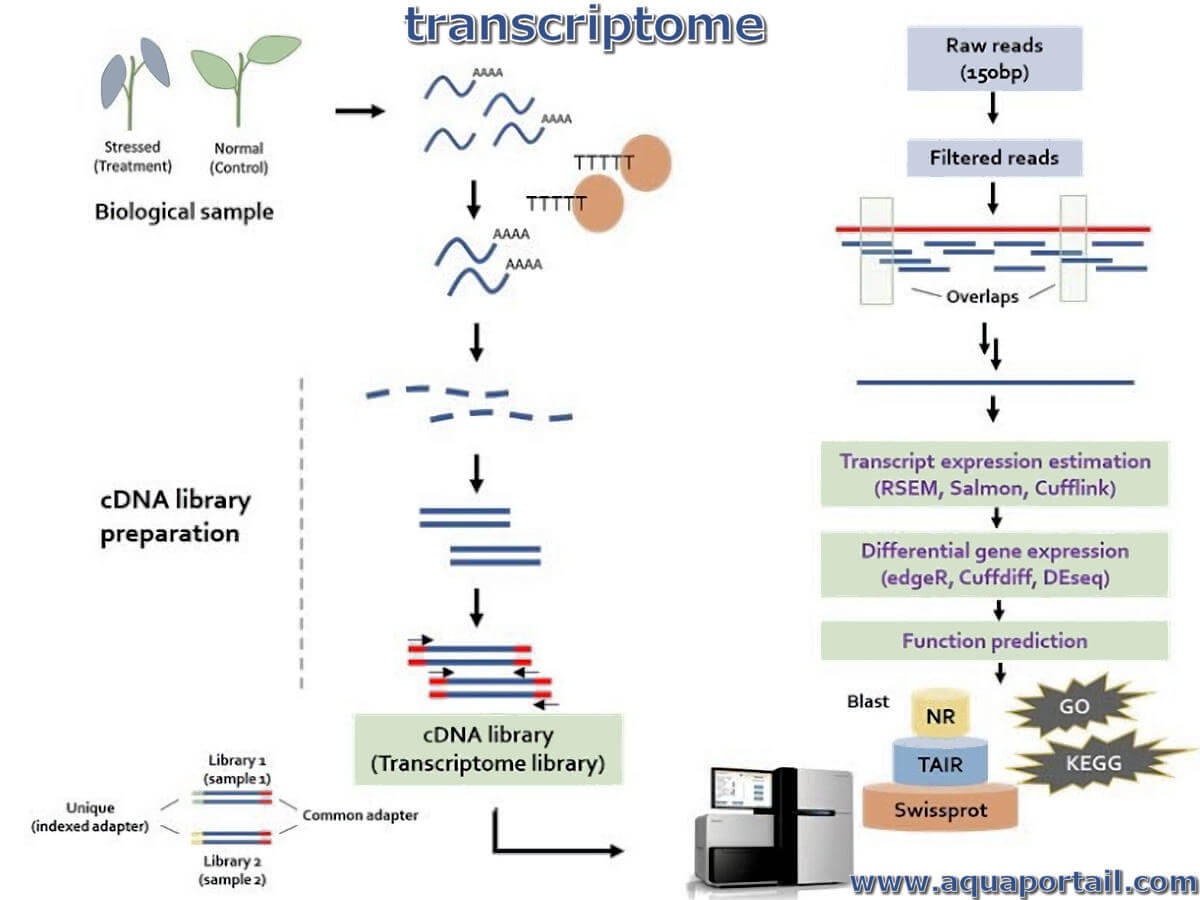
Le flux de travail général pour le séquençage métatranscriptomique est illustré à la figure 1. Pour le dire simplement, la première étape consiste à extraire l'ARN total de l'échantillon, puis à le détecter. L'ARN qualifié est soumis à un criblage de fragments, à la construction d'une base de données et aux tests de qualité correspondants. La bibliothèque qualifiée sera séquencée (principalement à l'aide de la plateforme de séquençage Illumina). Les données brutes obtenues par séquençage seront utilisées pour l'analyse bioinformatique.
Technologies d'analyse
La transcriptomique est une analyse complète d'ensembles entiers de transcrits pour une cellule, un tissu, un organe ou un organisme entier particulier correspondant à un moment ou à des stades de développement particuliers ou pouvant se trouver dans certaines conditions physiologiques spécifiques. La transcriptomique vise à :
- donner une annotation fonctionnelle du génome;
- fournir un aperçu de la structure transcriptionnelle des gènes;
- déchiffrer le site de départ de la transcription;
- élucider l'épissage;
- comprendre les modifications post-traductionnelles;
- cataloguer tous les types de transcrits (ARNm, ARNt, ARNr, ARNsi, ARN non codants, etc.);
- donner un aperçu de la complexité des processus de développement ou pour comprendre les maladies;
- élucider les gènes différentiellement exprimés.
La transcriptomique donne un aperçu du contenu génétique et du système de régulation dans la cellule. Les cellules de type sauvage, qu'il s'agisse de cellule bactérienne ou de lignée cellulaire, possèdent le même contenu génomique. Sur la base de différentes applications et conditions, différents gènes sont exprimés, entraînant différents modèles d'expression génique dans différents organismes. Il existe des mécanismes de régulation cellulaire par lesquels l'expression des gènes peut être activée et désactivée. De nombreux travaux ont été effectués sur les eucaryotes car ils contiennent à la fois des introns et des exons qui, grâce à l'épissage, fournissent un meilleur spectre du transcriptome total.
L'étude du transcriptome des procaryotes est à la traîne en raison de l'absence de la queue poly-A à l'extrémité 3′, qui est considérée comme une signature de l'ARNm mature chez les eucaryotes. Les données relatives au niveau d'expression des gènes dans le génome donné, le profilage du génome, les niveaux d'expression comparatifs entre différents ensembles de données expérimentales et l'effet de différents paramètres sur l'expression des gènes peuvent être évalués à l'aide d'outils de transcriptomique.
Les techniques couramment utilisées pour l'étude du transcriptome sont les méthodes basées sur les marqueurs de séquence exprimée (ou Expressed Sequence Tag, EST), SAGE, les puces à ADN basées sur l'hybridation, la PCR en temps réel, les méthodes de séquençage d'ARN basées sur NGS (ARN-seq), l'interférence ARN et les outils bioinformatiques pour les analyses des transcriptomes.
La méthodologie implique essentiellement l'isolement, la purification, la quantification, la construction d'une bibliothèque d'ADNc et le séquençage à haut débit. Les considérations importantes pour la sélection des outils de transcriptomique sont la rentabilité, la sensibilité, le débit élevé et la concentration minimale d'ARN de départ.
Les techniques couramment utilisées pour l'étude du transcriptome sont les méthodes basées sur les étiquettes de séquence exprimées (EST), SAGE, les puces à ADN basées sur l'hybridation, la PCR en temps réel, les méthodes de séquençage d'ARN basées sur NGS (ARN-seq), l'interférence ARN et les outils bioinformatiques pour les transcriptomes Analyse. La méthodologie implique essentiellement l'isolement, la purification, la quantification, la construction d'une bibliothèque d'ADNc et le séquençage à haut débit. Les considérations importantes pour la sélection des outils de transcriptomique sont la rentabilité, la sensibilité, le débit élevé et la concentration minimale d'ARN de départ.
Le séquençage des marqueurs de séquence exprimée "EST" donne un aperçu du niveau d'expression du gène. Des projets de séquençage des EST à grande échelle ont été appliqués sur des organismes modèles tels que Arabidopsis, Drosophila et l'humain. Des bases de données des EST sont également disponibles et fournissent une référence pour le profil d'expression. Bien qu'il soit utile pour étudier l'abondance des gènes et la découverte de nouveaux gènes, il s'agit d'une technique très coûteuse et longue.
Les microréseaux d'ADNc ou d'EST sont disponibles, les séquences d'ADNc ou les EST étant fixées sur la micropuce. La technique est similaire à la micropuce à ADN, mais l'échantillon ici sont les molécules d'ADNc marquées par fluorescence. Étant une technique rentable et rapide, elle aide à l'étude de l'abondance relative entre les gènes. SAGE est une autre méthode similaire au séquençage EST. Il est avantageux par rapport à l'EST car seules de courts marqueurs ("étiquettes") d'environ 15 bases seulement sont séquencées. Les courts fragments générés sont ensuite réunis et séquencés. Un pool d'ADNc peut être soumis à un NGS à haut débit connu sous le nom de ARN-seq pour la quantification, la découverte de nouveaux EST et le profilage des ARN.
Une puce à ADN, une puce génétique, une biopuce ou un microréseau est une collection de points d'ADN, d'ADNc, d'oligonucléotides attachés à un support solide tel que du verre, une puce de silicium formant un réseau à des fins de profilage d'expression; surveiller les niveaux d'expression de milliers de gènes simultanément et est une méthode basée sur l'hybridation. Plusieurs innovations concernant la fabrication de puces, le contrôle des conditions d'hybridation et l'analyse informatique des données générées ont été réalisées au fil des ans. La puce haute densité permet un profilage de l'expression génique relativement peu coûteux, une fois la fabrication de la puce réalisée. La principale limitation de cette technologie est que l'information sur la séquence du génome est une condition préalable et également un arrière-plan supérieur inhérent à la technique d'hybridation.
La PCR quantitative en temps réel (qRT-PCR) est un type de PCR privilégié pour la quantification fiable d'ARNm à faible abondance ou de transcrits à faible nombre de copies pour les études transcriptomiques. Les principaux avantages de cette technique sont sa haute sensibilité, sa meilleure reproductibilité et sa large plage de quantification dynamique. Il facilite l'expression des gènes et les études de régulation, même dans une seule cellule, grâce à sa capacité d'amplification exponentielle. La disponibilité de divers types de système de surveillance de fluorescence attachés à la PCR a entraîné sa popularité pour les études d'expression génique. Les problèmes d'amplification non spécifique, la formation d'amorces-dimères sont quelques-unes des limites de la qRT-PCR.
RNA-seq est l'une des technologies avancées à haut débit pour la transcriptomique. RNA-seq, également connu sous le nom de séquençage shotgun du transcriptome entier, utilise des outils NGS. Les avantages de l'ARN-seq sont qu'il ne repose pas sur la disponibilité de la séquence du génome, n'a pas de limites de quantification supérieures, présente une reproductibilité élevée et possède une large plage de détection dynamique.
Les transcriptomes des cellules souches et des cellules cancéreuses sont un domaine d'intérêt émergent qui peut fournir un aperçu des processus de différenciation cellulaire et de cancérogenèse. La transcriptomique peut être utilisée dans la fécondation in vitro pour une sélection appropriée des embryons. De même, dans la découverte de biomarqueurs, elle peut être utilisée pour évaluer la sécurité des médicaments ou le risque chimique.
Microdissection laser
La transcriptomique par microdissection laser fait référence aux études des transcrits de gènes, généralement analysés par des microréseaux d'expression d'ADNc. Ces matrices mesurent la quantité de niveaux d'ARNm à l'état d'équilibre dans le tissu étudié, qui bien sûr peut parfois différer considérablement de la quantité de protéine fonctionnelle dans ce tissu.
De telles études d'expression d'ADNc ont conduit à un certain nombre de percées passionnantes en science fondamentale, ainsi qu'en biologie du cancer, dans lesquelles le pronostic et le traitement peuvent parfois être dirigés avec plus de précision en observant un profil d'expression d'une tumeur.
Synonymes, antonymes
Voir tous les synonymes pour "transcriptomique".2 synonymes (sens proche) de "transcriptomique" :
- métatranscriptomique
- profilage d'expression
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à TRANSCRIPTOMIQUE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot TRANSCRIPTOMIQUE est dans la page 3 des mots en T du lexique du dictionnaire.
Mots en T à proximité
Transcaucasie transcriptase transcriptase inverse transcription transcriptome transcriptomiquetranscytose transdifférenciation transducine transducteur transduction
En rapport avec "transcriptomique"

L'interactomique est l'étude de l'ensemble des interactions entre différentes biomolécules, et notamment les protéines, dans un environnement donné.
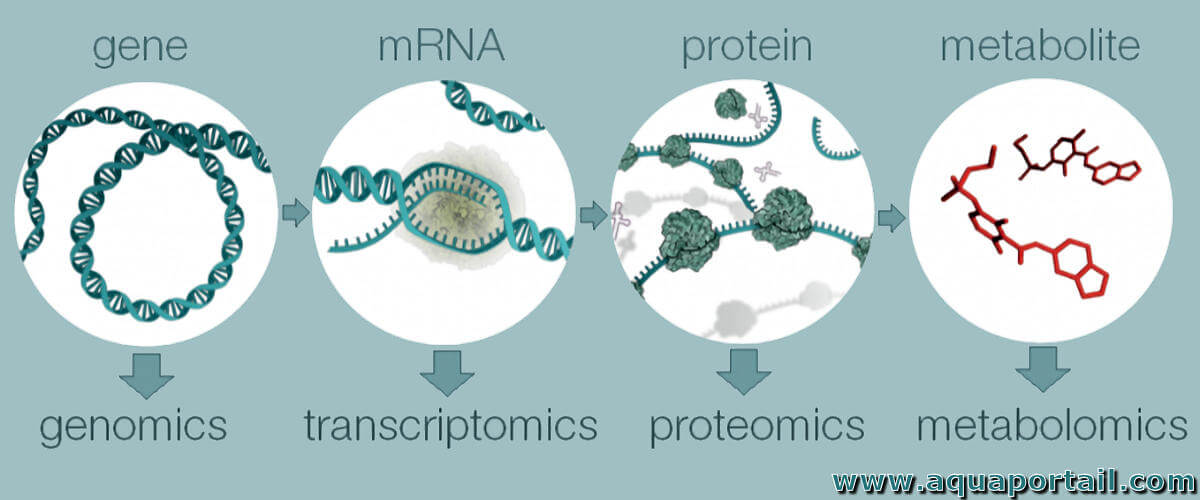
La métabolomique est l'étude à grande échelle de petites molécules, communément appelées métabolites, dans les cellules, les biofluides, les tissus...

La protéomique est l'étude et l'analyse systémique à grande échelle de chaque protéine, en particulier de leurs structures, fonctions, activité, quantité...
Le transcriptome est l'ensemble des transcrits d'ARN synthétisés par une cellule ou un tissu entier, incluant les ARNm et les ARN non codants.
