Transcriptase
Définition
L'activité transcriptase au sein d'un virion assemblé catalyse la synthèse de l'ARN à brin positif descendant à partir de la matrice d'ARN double brin (bicaténaire) parental, accompagnée du déplacement du brin positif parental et de sa libération du virion. Son rôle est l'opposé de la transcriptase inverse.
L'action d'une transcriptase :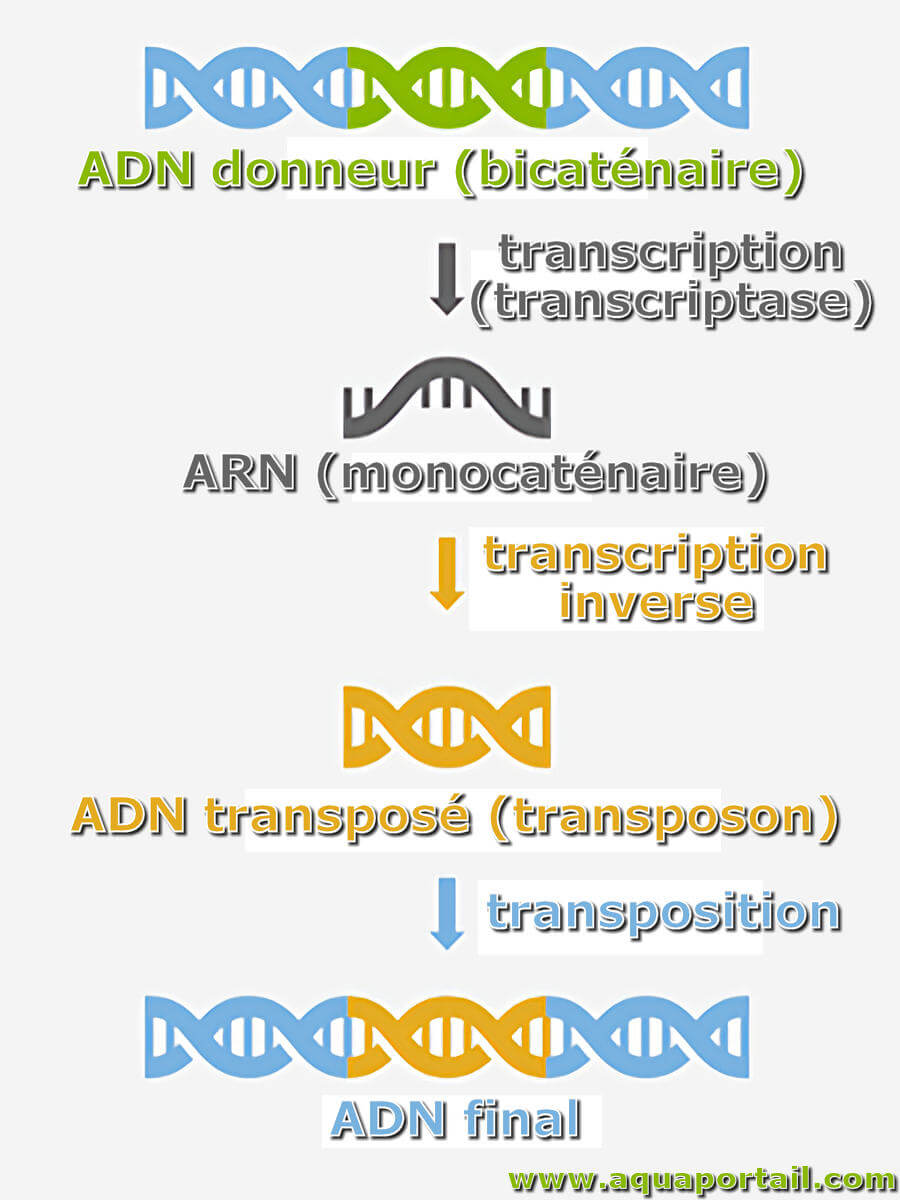
La régulation des fonctions de transcriptase et de réplicase par des ligands d'ARN structurés en 5′ émerge comme un thème commun pour les polymérases sNSV.
Explications
Le passage de la transcriptase à la réplicase est médié par des facteurs intrinsèques et extrinsèques. La question de savoir si la liaison de la polymérase-N affecte la conformation des domaines cap-snatching et/ou le résultat fonctionnel de l'initiation reste une question ouverte.
Cependant, N n'est pas nécessaire pour la synthèse d'ARN amorcée et non amorcée sur des modèles courts, ce qui suggère que la liaison à la polymérase-N ne fonctionne pas dans la licence initiale de réplicase ou de transcriptase et sert probablement de facteur d'allongement et de processivité. Il est également possible que l'architecture et la fonction de la polymérase dépendent de la phosphorylation de N, qui sert de commutateur pour l'oligomérisation de N sur l'ARN encapsidé.
Action
Au cours de la transcription, chaque segment du génome d'ARN double brin doit traverser le site actif de l'enzyme polymérase. La nature entièrement conservatrice de la transcriptase impose certaines exigences topologiques au processus. Les deux brins des segments d'ARN double brin parentaux doivent se dérouler avant la transcription pour permettre aux brins de la matrice –ve-sens d'entrer dans le site actif de la polymérase.
Cependant, les duplex de brins parent-fille résultants doivent également être séparés afin que les chaînes d'ARN naissantes puissent être exportées de la particule centrale probablement à travers les pores de la coque du sous-noyau VP3 (T2) aux cinq axes, permettant aux brins parentaux de le duplex d'ARNdb à reanneler. VP6 (Hel) est une hélicase et possède une activité ATPase utilisant l'énergie libérée par l'hydrolyse de l'ATP en ADP et Pi pour séparer l'ARN double brin en ses brins composants. Cette activité peut faciliter la séparation des brins d'ARN avant ou après la transcription ou les deux.
Cas des virus (rotavirus)
La synthèse des transcrits viraux est médiée par une ARN polymérase virale dépendante de l'ARN (transcriptase) qui possède un certain nombre d'activités enzymatiques. La transcriptase est un composant du virion et les propriétés de cette enzyme (ou complexe enzymatique) ont été déduites en étudiant les caractéristiques des produits issus de réactions de transcription in vitro.
Les particules de rotavirus contiennent vraisemblablement les mêmes activités enzymatiques que celles trouvées dans les réovirus, y compris la transcriptase, la nucléotide phosphohydrolase, la guanyltransférase et deux méthylases. Ces activités sont déduites car les transcrits de rotavirus produits in vitro en présence de S-adénosyl méthionine possèdent une structure de coiffe 5′-terminale méthylée, m7GpppGm, et la transcription est inhibée par le pyrophosphate. Les particules contiennent également une activité poly(A) polymérase dont la fonction précise reste inconnue; il a été postulé qu'il était responsable de la synthèse des molécules oligo (A).
La transcriptase associée au virus est latente dans les particules à trois couches et peut être activée in vitro par traitement avec un agent chélateur ou par traitement par choc thermique. De tels traitements entraînent l'élimination des protéines de la capside externe avec conversion des particules à triple couche en particules à double couche. Dans les cellules infectées, il a été démontré que les particules à triple couche ne sont pas recouvertes de particules à double couche, et la transcription dans les cellules intervient à partir de ces particules. La transcription est asymétrique et tous les transcrits sont des brins (+) pleine longueur fabriqués à partir du brin d'ARNdb (-). Le site intracellulaire de la transcription est inconnu.
L'activation de l'activité transcriptase est un processus qui n'est pas bien compris. "Activation" peut être un terme impropre, car dans les réovirus, il a été suggéré que ce processus ne modifie pas réellement le complexe enzymatique, mais libère plutôt les matrices des contraintes structurelles, leur permettant de dépasser le site catalytique de la transcriptase. La transcription du rotavirus nécessite une forme hydrolysable d'ATP et des études avec des analogues qui inhibent la transcription suggèrent que l'ATP est nécessaire dans des réactions autres que la polymérisation.
L'ATP peut être utilisé pour l'initiation ou l'allongement de molécules d'ARN, comme cela a été décrit pour les ARN polymérases du virus de la stomatite vésiculeuse ou du virus de la vaccine. On ne sait toujours pas si des polypeptides distincts dans les particules actives sur le plan de la transcription remplissent des fonctions distinctes ou si les polypeptides du noyau interne fonctionnent comme un complexe enzymatique, mais l'emplacement de VP1 et VP3 en tant que complexe structurel aux cinq axes favorise cette dernière hypothèse.
VP1 a été réticulé avec un analogue de nucléotide photoréactif, indiquant que VP1 est un composant de la transcriptase. VP3 dans les particules virales et exprimé seul dans les cellules d'insectes peut se lier au GTP, suggérant que cette protéine est la guanyltransférase. Que VP3 possède également une activité transcriptase seule ou en association avec VP1 reste incertain. De même, le rôle de VP2 dans le processus de transcription n'est pas clair.
La synthèse d'ARN à brin positif et négatif a été étudiée dans des cellules infectées par SA11 et dans un système acellulaire. L'optimisation d'un système électrophorétique qui permet la séparation des brins positifs et négatifs des ARN de rotavirus sur la base des brins complémentaires migrant à des vitesses différentes dans des gels d'urée acide et d'agarose a facilité ces études.
L'analyse de la cinétique de synthèse de l'ARN dans les cellules infectées a montré que les ARN à brin positif et négatif sont détectés initialement à 3 heures après l'infection, en accord avec d'autres études qui ont examiné le moment de l'incorporation de [3H] uridine dans l'ARN du rotavirus. Après 3 heures, le niveau de transcription augmente jusqu'à 9 à 12 heures, moment auquel les niveaux d'ARN à brin plus sont maximaux. Le rapport entre la synthèse d'ARN à brin plus et à brin moins change pendant l'infection et le niveau maximal de synthèse d'ARN à brin moins est observé plusieurs heures avant le pic de synthèse d'ARN à brin plus.
On a émis l'hypothèse que le retard dans l'obtention de la synthèse maximale d'ARN à brin plus était dû à une exigence d'accumulation de quantités stoechiométriques d'une protéine (VP6) nécessaire à l'assemblage des particules de transcriptase. L'ARN à brin positif nouvellement synthétisé et préexistant peut servir de modèle pour la synthèse d'ARN à brin négatif tout au long de l'infection, un résultat inattendu basé sur des études antérieures avec des réovirus. L'observation que le niveau de réplication de l'ARN n'augmente pas continuellement en conjonction avec les niveaux croissants d'ARN à brin plus suggère que la réplication de l'ARN est régulée par des facteurs autres que le niveau d'ARN à brin plus dans la cellule infectée.
La synthèse de l'ARNdb a également été analysée à l'aide d'un système sans cellule pour étudier la réplication de l'ARN du rotavirus. Les composants de ce système comprennent : (1) des particules à noyau ouvert préparées à partir de particules purifiées à double couche ou de particules de type virus composées de VP1/2/3/6; (2) un ARNm viral ajouté de manière exogène ou un transcrit synthétique; et (3) des sels et des nucléosides triphosphates. Le système de réplication in vitro ne nécessite pas les protéines non structurelles et réplique spécifiquement les matrices de rotavirus.
La synthèse d'ARNdb in vitro est un processus asymétrique dans lequel un ARN à brin positif sensible à la nucléase agit comme matrice pour la synthèse d'ARN à brin négatif. Après sa synthèse, l'ARNdb reste associé aux particules sous-virales, ce qui suggère que l'ARNdb libre ne se trouve pas dans les cellules.
Ce système in vitro prend en charge l'initiation de l'ARN à brin négatif en utilisant un ARN à brin positif viral exogène comme matrice. La conversion d'ARNm exogène en ARNdb par des particules sous-virales fournit une méthode pour étudier (1) la spécificité des protéines virales dans la reconnaissance et la réplication des ARNm de rotavirus, et (2) l'effet de l'ajout d'ARN synthétiques exogènes contenant des mutations spécifiques sur la réplication.
Enfin, il est possible que l'ARNdb (double brin) répliqué naissant puisse être assemblé dans ces particules virales dans ce système de réplication in vitro. Malheureusement, à ce jour, l'efficacité du système n'a pas été suffisante pour atteindre cet objectif. Ensemble, ces résultats suggèrent qu'un système acellulaire pour soutenir la réplication de l'ARN du rotavirus, la transcription et l'assemblage de particules sous-virales peut être établi.
Ce système devrait être utile pour aider à définir les défauts des mutants de rotavirus et pour étudier les séquences d'ARN et les protéines impliquées dans la réplication et l'assemblage du virus. Le rôle des protéines individuelles et des complexes protéiques spécifiques dans la réplication de l'ARN et la morphogenèse virale ne sera probablement pas résolu tant qu'ils ne seront pas étudiés in vitro avec des espèces pures de protéines natives de rotavirus et d'ARN viraux.
Synonymes, antonymes
Voir tous les synonymes pour "transcriptase".0 synonyme (sens proche) pour "transcriptase".
2 antonymes (sens contraire) :
Les mots ou les expressions apparentés à TRANSCRIPTASE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot TRANSCRIPTASE est dans la page 3 des mots en T du lexique du dictionnaire.
Mots en T à proximité
tranquilliseur transaminase transamination transcapsidation Transcaucasie transcriptasetranscriptase inverse transcription transcriptome transcriptomique transcytose
En rapport avec "transcriptase"
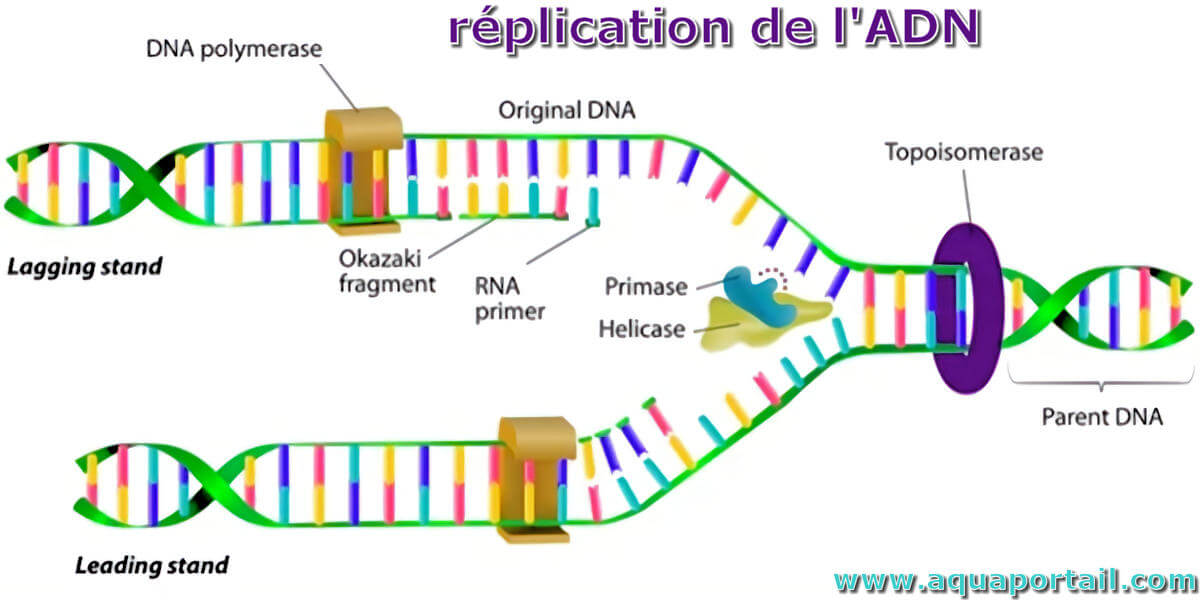
Une réplication est l'ensemble des processus qui permettent de reproduire à l'identique un ADN.
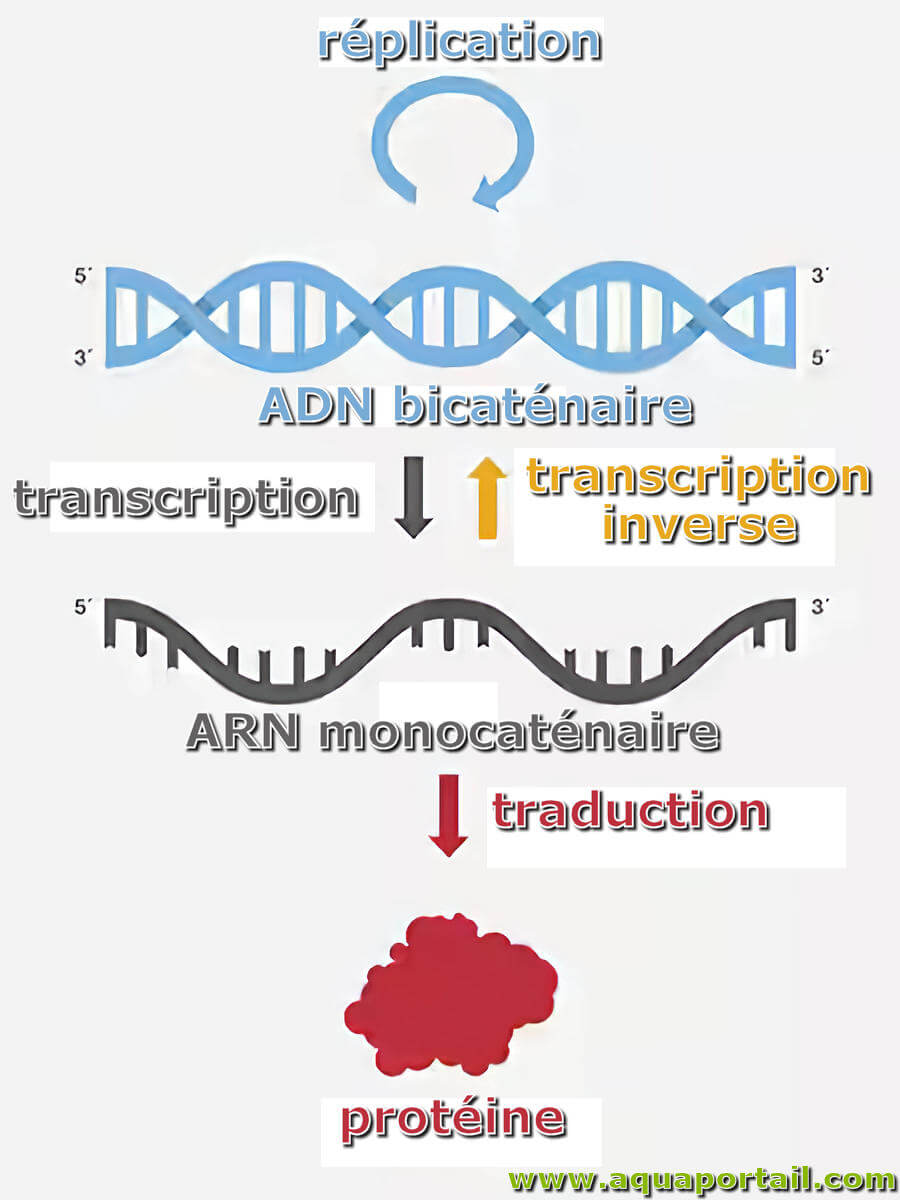
La transcriptase inverse, également appelée rétrotranscriptase et ADN polymérase ARN-dépendante, est une ADN polymérase qui transcrit l'ARN simple brin en ADN.

La transcription est un processus de synthèse de l'ARN complémentaire à partir d'une matrice d'ADN.
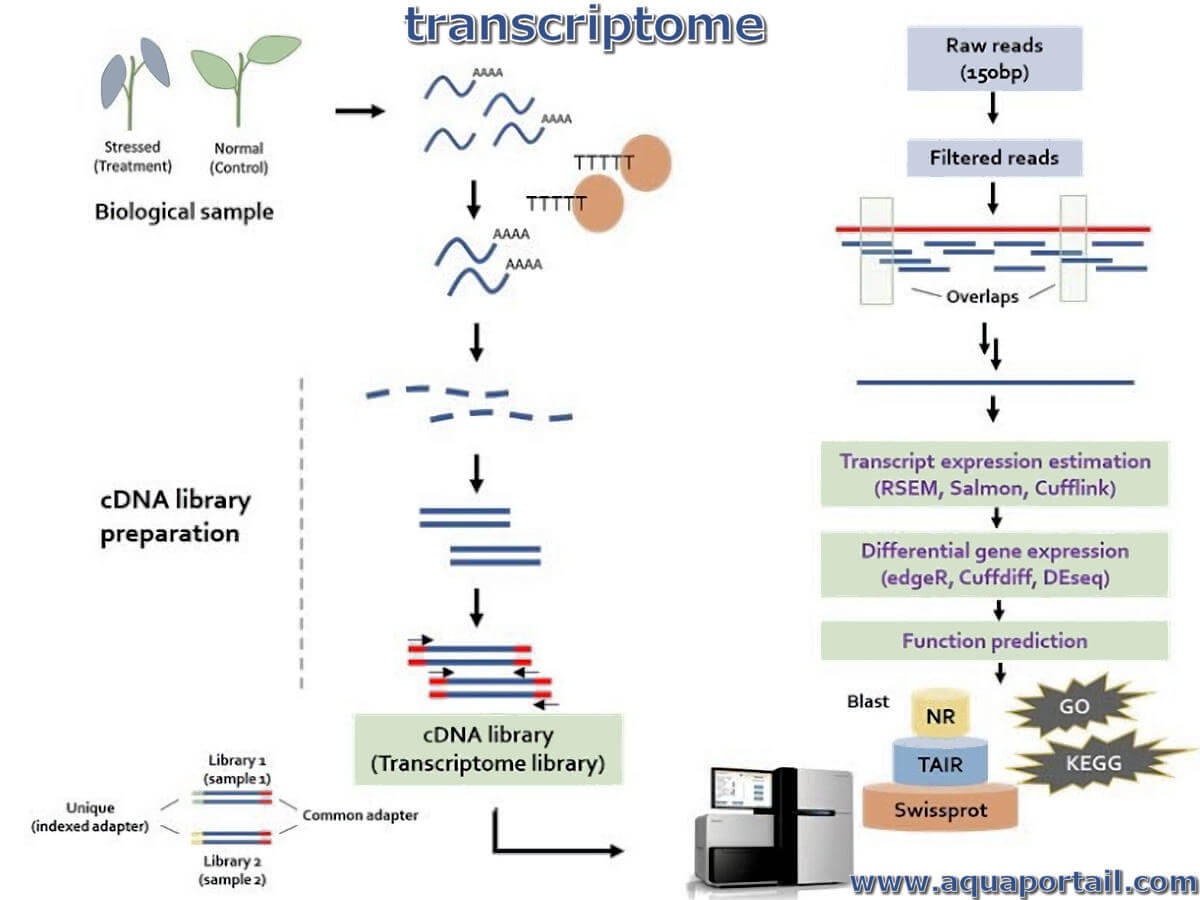
Le transcriptome est l'ensemble des transcrits d'ARN synthétisés par une cellule ou un tissu entier, incluant les ARNm et les ARN non codants.
