Marqueur de séquence exprimée
Définition
Un marqueur de séquence exprimée (EST ou Expressed Sequence Tag) est un morceau d'ADNc (ARNm retranscrit), long de plusieurs centaines de nucléotides. Un seul marqueur a généralement 200 à 500 nucléotides. Les marqueurs de séquence exprimées (EST) fournissent une fenêtre directe sur la relation entre l'activité et le comportement des gènes.
Des marqueurs de séquence exprimées (EST) :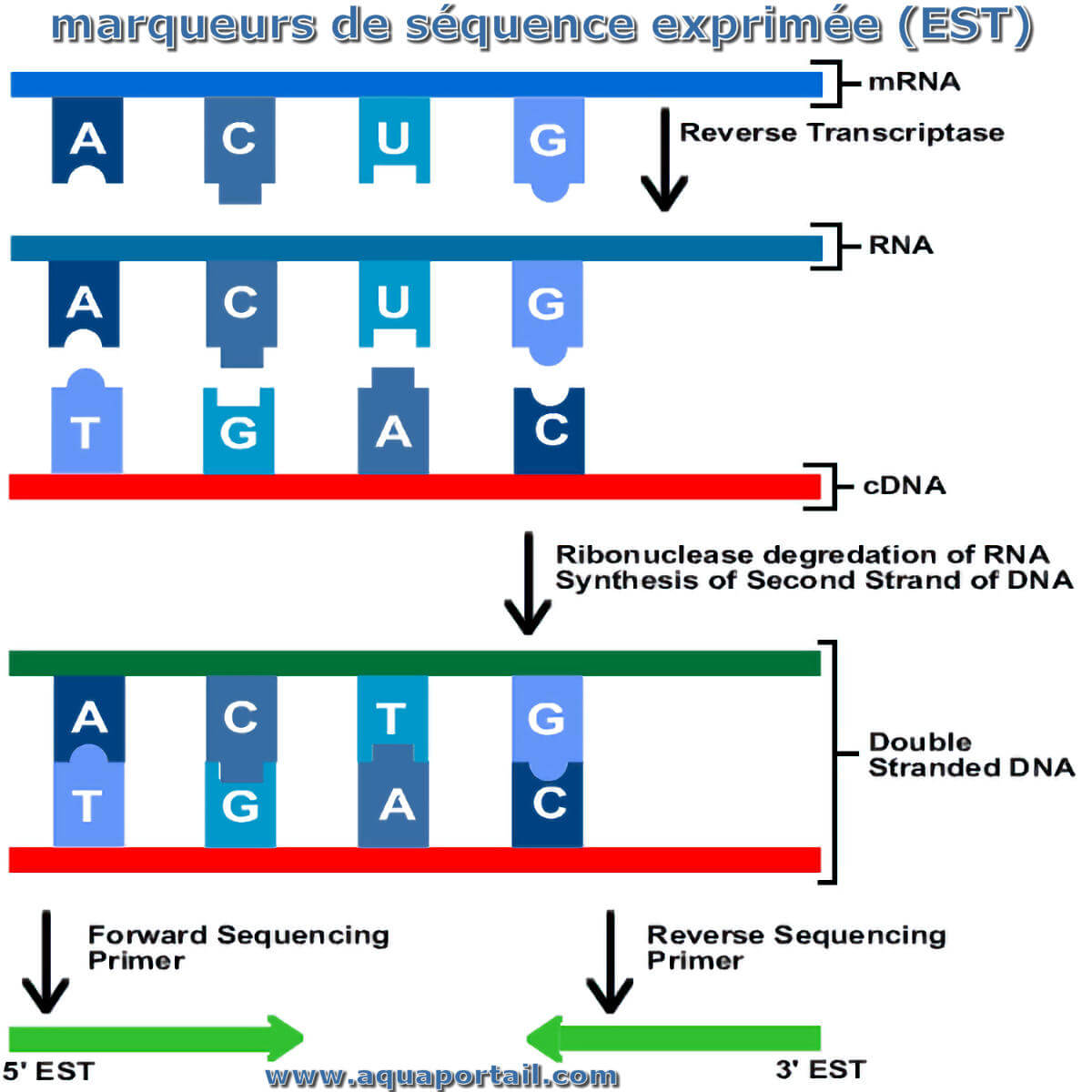
Les marqueurs de séquence exprimées (EST) sont générés par séquençage d'ADNc, qui est lui-même synthétisé à partir des molécules d'ARNm dans une cellule. Les ARNm dans une cellule sont des copies des gènes qui sont exprimés. L'ARNm ne contient pas de séquences des régions entre les gènes, ni des introns non codants qui sont présents dans de nombreuses parties intéressantes du génome.
Explications
Les marqueurs de séquence exprimée ont été conçus à l'origine dans le but d'initier la transcription des gènes, mais sont devenus extrêmement important dans l'identification des gènes et dans la détermination de la structure des chromosomes. Sans les EST, la transcriptomique (et les disciplines de l'interactomique) n'existerait pas.
Les marqueurs de séquence génétique radiomarqués ou fluorescents peuvent être utilisés pour indiquer les gènes et leur emplacement sur un chromosome. Un marqueur de séquence exprimée peut également montrer de grandes mutations sur les chromosomes (échanges de bras chromosomiques), ce qui peut être utile dans la recherche sur le cancer, par exemple.
Un EST est produit à partir d'ADNc qui est coupé en petits morceaux à l'aide d'enzymes de restriction. Les molécules d'ANDc sont obtenues en rétrotranscriptant la population d'ARNm contenue dans les cellules d'un tissu.
Les marqueurs de séquence exprimée peuvent être utilisés pour identifier les transcriptions génétiques et sont cruciaux dans le processus de découverte de gènes et dans la détermination des séquences génétiques. L'identification de l'EST dans l'expression génétique s'est produite à un rythme accéléré, avec environ 66 millions d'EST en 2008, puis 75 millions en 2013, disponibles dans des bases de données publiques telles que GenBank. Plus de 8 millions de marqueurs de séquence exprimée proviennent de séquences humaines. Il s'agit notamment de nombreuses séquences redondantes ou cartographiques en différents points d'un même ARNm, ce qui explique l'énorme abondance d'EST par rapport au nombre de gènes.
La fiabilité du système est bonne, car la probabilité statistique qu'une partie se reproduise de manière aléatoire est très faible. Pour une séquence de 20 nucléotides, il existe déjà une très grande spécificité, à savoir A, C, T (ou U) et G (en raison des quatre bases possibles). C'est 1 sur près de 1,1 billion. Il s'agit d'une limite théorique qui ne s'appliquerait que si le génome et l'EST étaient constitués de séquences complètement aléatoires. En réalité, la spécificité sera beaucoup plus faible, bien qu'elle soit encore si élevée qu'elle est négligeable comme source d'erreur.
Les EST ne correspondent pas nécessairement à des fragments d'ARNm, mais elles peuvent également appartenir à d'autres ARN ne codant pour aucune protéine (ARNnc), tels que les miARN, bien que présents en très faibles quantités. EST Sont des séquences courtes (généralement entre 300 pb et 500 pb) et plutôt inexactes (environ 2 % d'erreur); cela est dû au fait que la réaction de séquençage est généralement effectuée automatiquement dans une seule direction ou sans chevauchement. Les EST sont donc généralement plus courtes que les transcriptions complètes, car le séquençage d'un ARNm entier est plus coûteux, mais cela peut produire des résultats incorrects s'il existe des formes inconnues d'épissage alternatif.
Le système immunitaire des plantes utilise également le fait que de courtes séquences (ARN) sont déjà spécifiques à une séquence (voir ARNi).
L'EST a été introduit en 1991 par Craig Venter (1946-), un biologiste américain connu pour avoir défié le Human Genome Project dans la course au séquençage du génome.
Synonymes, antonymes
Voir tous les synonymes pour "marqueur de séquence exprimée".2 synonymes (sens proche) de "marqueur de séquence exprimée" :
- EST
- marqueur de séquence génétique
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à MARQUEUR DE SÉQUENCE EXPRIMÉE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
L'expression MARQUEUR DE SEQUENCE EXPRIMEE est dans la page 2 des mots en M du lexique du dictionnaire.
Mots en M à proximité
marlin noir marmite de géant marnage marnemarqueur biologique marqueur de séquence expriméemarqueur génétique marronnage marsouin marsupium martin-pêcheur
En rapport avec "marqueur de séquence exprimée"

Le marqueur biologique est une molécule biologique présente dans le sang, d'autres fluides corporels ou des tissus et qui est le signe d'un processus normal...
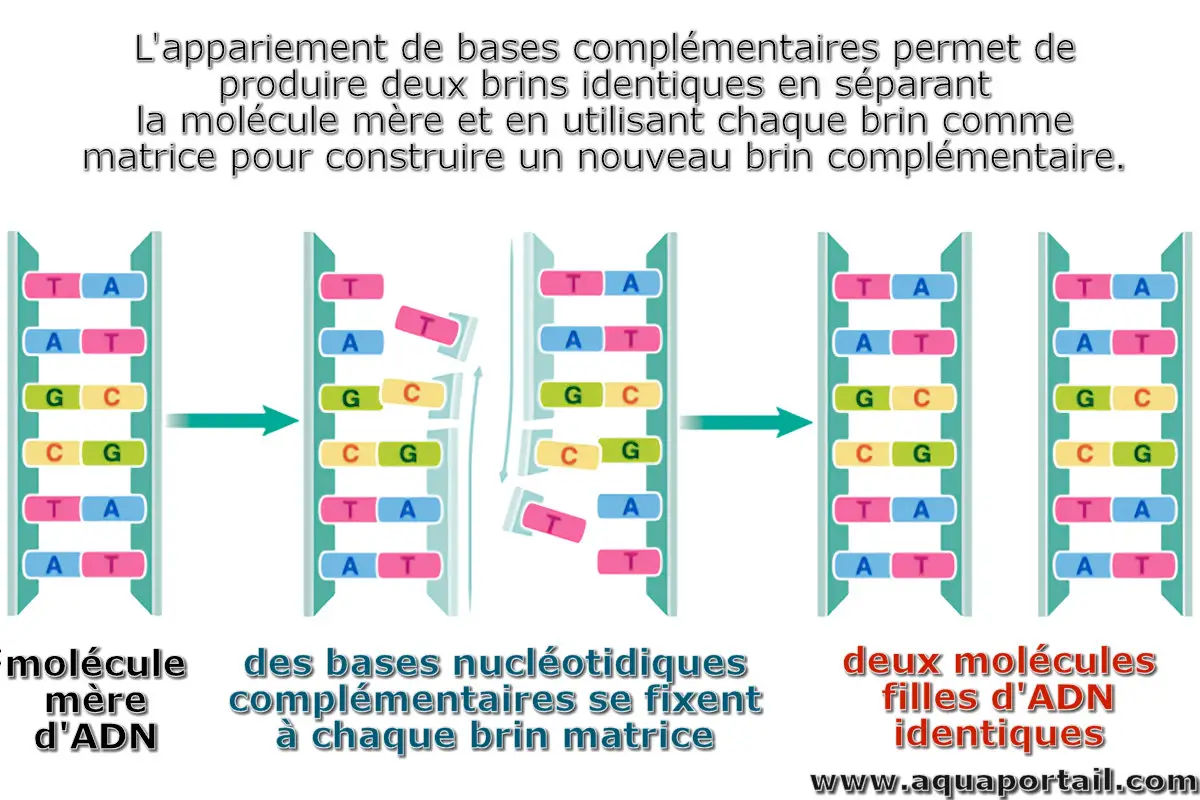
L'ADN complémentaire, l'ADNc, est une copie d'ADN d'un ARN messager (ARNm) produite par la transcriptase inverse, car une ADN polymérase peut utiliser l'ADN...
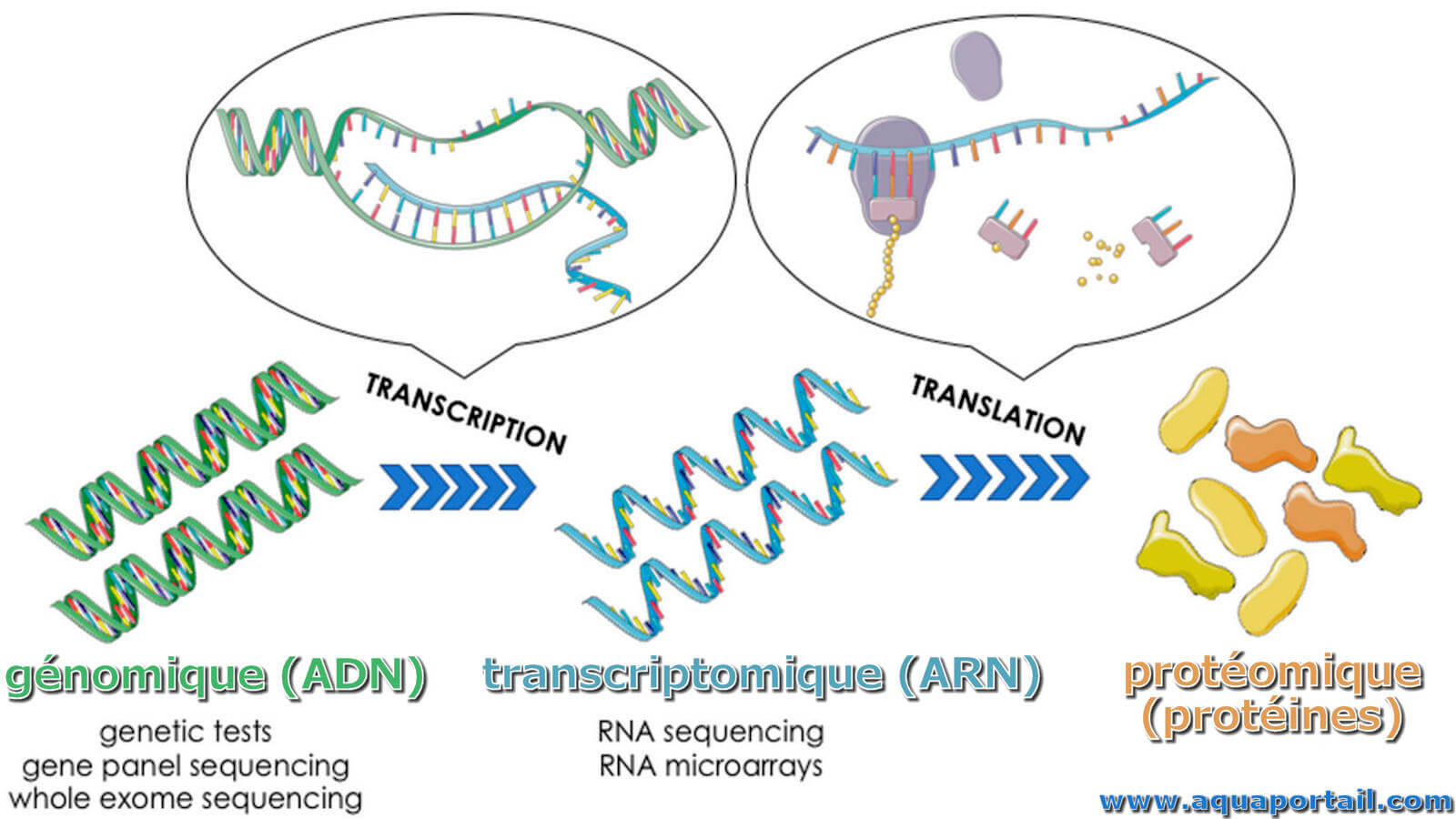
La transcriptomique est la discipline de l'analyse du transcriptome, de l'ensemble complet des transcrits d'ARN produits par le génotype à un instant donné...
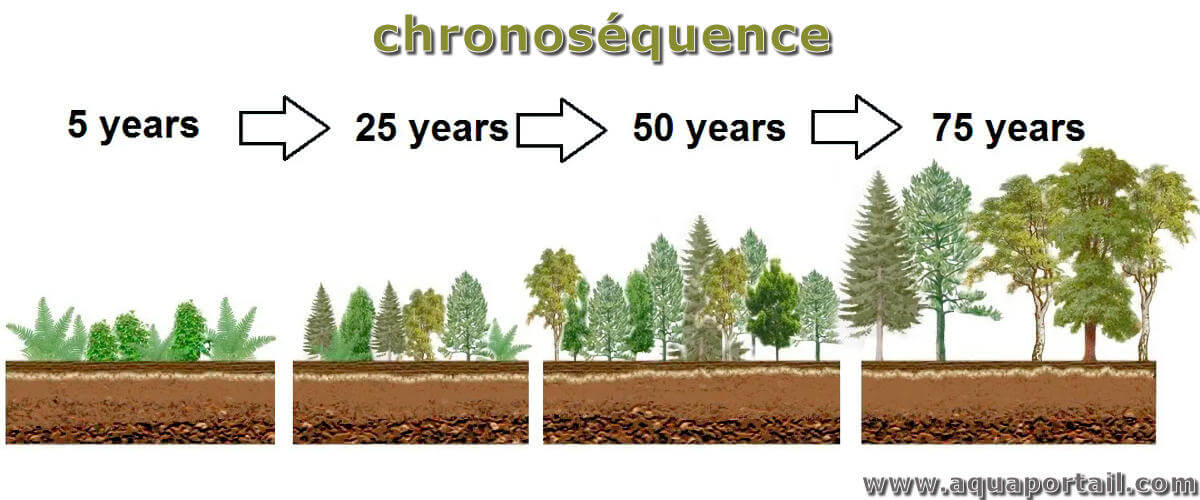
Une chronoséquence est une suite de sols génétiquement liés dans lesquels la végétation, la topographie et le climat sont similaires.
