Séquence génétique
Définition
L'analyse d'une séquence génétique est une technique consistant à obtenir la séquence nucléotidique de zones homologues d'ADN, codantes ou non, et à les comparer entre elles pour en déduire des liens de parentés. Le séquençage génétique de l'ADN détermine l'ordre des quatre éléments constitutifs chimiques (paires de bases) qui composent la molécule d'ADN.
Extraction d'une séquence génétique :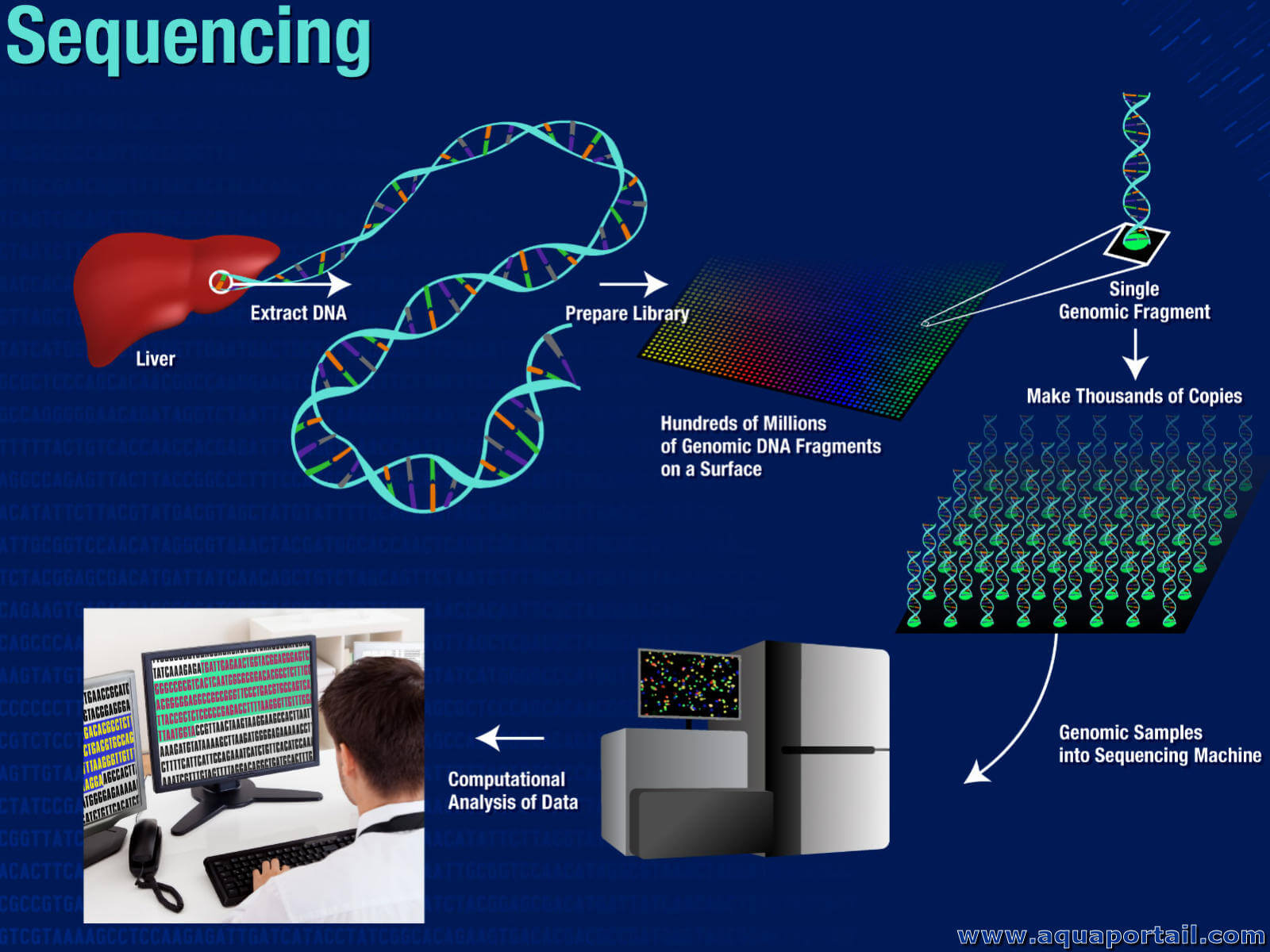
Le coût de l'obtention d'une séquence génétique de qualité est encore très élevé aujourd'hui.
Explications
Les séquences génétiques d'un génome sont déterminées par séquençage (séquençage du génome), une signature ADN, un profil génétique, une empreinte génétique, et une analyse génétique. La séquence indique aux scientifiques le type d'information génétique contenue dans un segment d'ADN particulier.
La détermination de l'ordre des éléments constitutifs de l'ADN (nucléotides) dans le code génétique d'un individu, appelé séquençage de l'ADN, a fait progresser l'étude de la génétique et est une technique utilisée pour tester les troubles génétiques. Deux méthodes, le séquençage de l'exome entier et le séquençage du génome entier, sont de plus en plus utilisées en santé et en recherche pour identifier les variations génétiques; les deux méthodes reposent sur de nouvelles technologies qui permettent un séquençage rapide de grandes quantités d'ADN. Ces approches sont connues sous le nom de séquençage de nouvelle génération (ou séquençage de nouvelle génération).
La technologie de séquençage d'origine, appelée séquençage Sanger (du nom du scientifique qui l'a développée, Frederick Sanger), était une percée qui a aidé les scientifiques à déterminer le code génétique humain, mais elle prend du temps et coûte cher. La méthode Sanger a été automatisée pour la rendre plus rapide et est encore utilisée dans les laboratoires aujourd'hui pour séquencer de courts morceaux d'ADN, mais il faudrait des années pour séquencer tout l'ADN d'une personne (connu sous le nom de génome de la personne). Le séquençage de nouvelle génération a accéléré le processus (ne prenant que quelques jours à quelques semaines pour séquencer un génome humain) tout en réduisant le coût.
Avec le séquençage de nouvelle génération, comme le séquençage shotgun, il est désormais possible de séquencer de grandes quantités d'ADN, par exemple tous les morceaux d'ADN d'un individu qui fournissent des instructions pour fabriquer des protéines. On pense que ces morceaux, appelés exons, représentent 1 % du génome d'une personne. Ensemble, tous les exons d'un génome sont connus sous le nom d'exome, et la méthode de séquençage est connue sous le nom de séquençage de l'exome entier. Cette méthode permet d'identifier des variations dans la région codant pour la protéine de n'importe quel gène, plutôt que dans seulement quelques gènes sélectionnés. Étant donné que la plupart des mutations connues qui causent la maladie interviennent dans les exons, le séquençage de l'exome entier est considéré comme une méthode efficace pour identifier les mutations pouvant causer la maladie.
Cependant, les chercheurs ont découvert que les variations d'ADN en dehors des exons peuvent affecter l'activité des gènes et la production de protéines et conduire à des troubles génétiques - des variations que le séquençage complet de l'exome manquerait. Une autre méthode, appelée séquençage du génome entier, détermine l'ordre de tous les nucléotides dans l'ADN d'un individu et peut déterminer les variations dans n'importe quelle partie du génome.
Alors que beaucoup plus de changements génétiques peuvent être identifiés avec le séquençage de l'exome entier et du génome entier qu'avec le séquençage de gènes sélectionnés, la signification d'une grande partie de ces informations est inconnue. Parce que tous les changements génétiques n'affectent pas la santé, il est difficile de savoir si les variantes identifiées sont impliquées dans la condition d'intérêt. Parfois, une variante identifiée est associée à un trouble génétique différent qui n'a pas encore été diagnostiqué (on les appelle des découvertes fortuites ou secondaires).
En plus d'être utilisé en clinique, le séquençage de l'exome entier et du génome entier sont des méthodes précieuses pour les chercheurs. Une étude continue des séquences d'exome et du génome peut aider à déterminer si de nouvelles variations génétiques sont associées à des problèmes de santé, ce qui facilitera le diagnostic de la maladie à l'avenir.
Séquence nucléotidique
La séquence nucléotidique est la manière typique dont la structure d'un gène est définie. L'analyse de la séquence nucléotidique du gène E a révélé que le virus de la fièvre jaune a évolué tôt dans la lignée des flavivirus transmis par les moustiques, il y a environ 3 000 ans.
Le virus de la fièvre jaune est apparenté de loin à d'autres flavivirus par des tests sérologiques et en termes d'homologie dans les séquences d'ARN. Les souches du virus de la fièvre jaune de différentes régions géographiques peuvent être distinguées par l'empreinte ARN et sont actuellement classées en trois génotypes, un d'Afrique de l'Ouest, un d'Afrique centrale et orientale et un d'Amérique du Sud. Un haut degré d'homologie a été noté entre les souches appartenant à un même topotype.
Les séquences de nucléotides ou les séquences d'acides aminés prédites des membres du genre forment une lignée distincte au sein de la sous-famille. Les membres du genre n'ont été trouvés que chez les oiseaux et sont les seuls membres de la sous-famille associés à la malignité. La production de virus extracellulaire infectieux semble être limitée à l'épithélium plume-follicule. Tous les membres actuels présentent une réaction sérologique croisée.
Séquence d'acides nucléiques
Une séquence d'acides nucléiques, l'ARN messager ou ARNm, est traduite en la protéine qu'elle code au moyen d'ARN de transfert interagissant avec l'appareil ribosomal. Les ARN de transfert se lient à trois nucléotides à la fois et divisent ainsi la séquence d'acide nucléique en codons triplets, chacun spécifiant un acide aminé.
Cependant, selon le point auquel la division en codons commence, l'acide nucléique peut être lu en trois phases ou cadres de lecture distincts, et la séquence ne contient pas de signaux de ponctuation pour indiquer quel cadre doit être utilisé. Une mutation de décalage de cadre est une insertion ou une délétion dans la séquence d'acide nucléique qui déplace le mécanisme de traduction d'un cadre de lecture à un autre.
Synonymes, antonymes
Voir tous les synonymes pour "séquence génétique".2 synonymes (sens proche) de "séquence génétique" :
- séquence d'acides nucléiques
- séquence nucléotidique
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à SÉQUENCE GÉNÉTIQUE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
L'expression SEQUENCE GENETIQUE est dans la page 2 des mots en S du lexique du dictionnaire.
Mots en S à proximité
séquençage du génome séquençage génomique séquençage shotgun séquence codante séquence d'ADN séquence génétiqueséquence nucléotidique séquence signal séquences moléculairesséquestration du carbone sère
En rapport avec "séquence génétique"
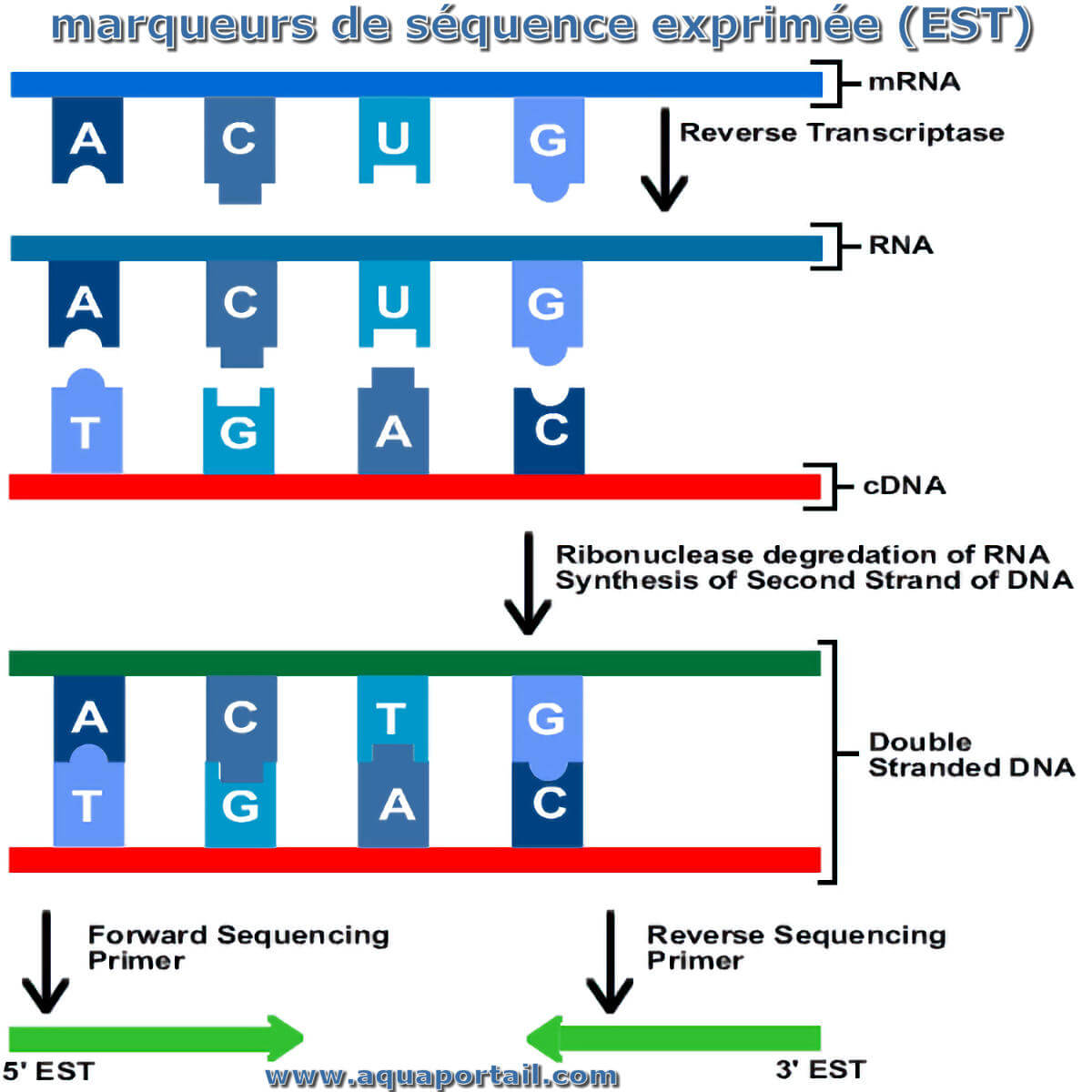
Un marqueur de séquence exprimée (EST ou Expressed Sequence Tag) est un morceau d'ADNc (ARNm retranscrit), long de plusieurs centaines de nucléotides.
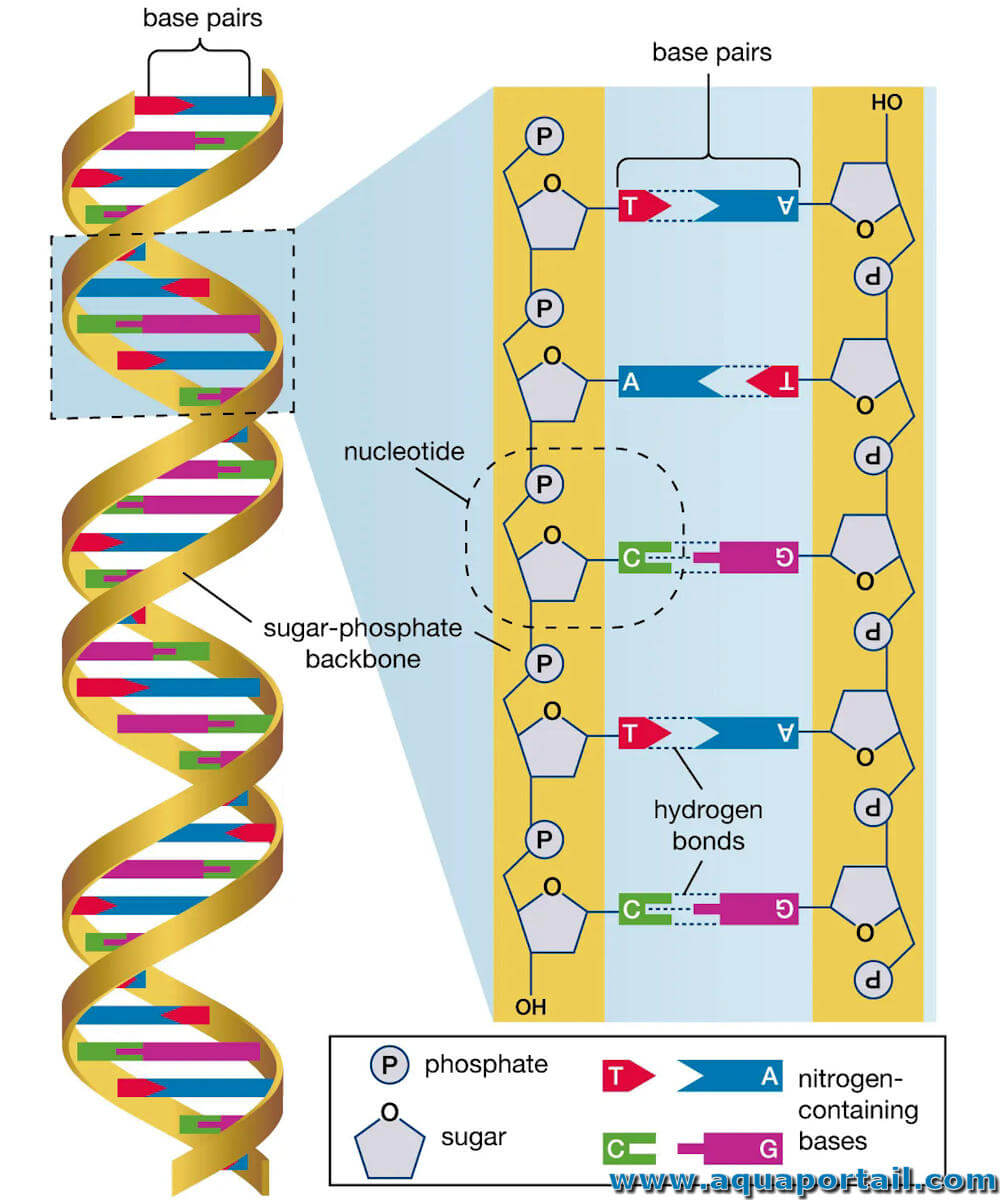
En biologie, le séquençage est la détermination de l'ordre linéaire des composants d'une macromolécule, les séquences moléculaires.
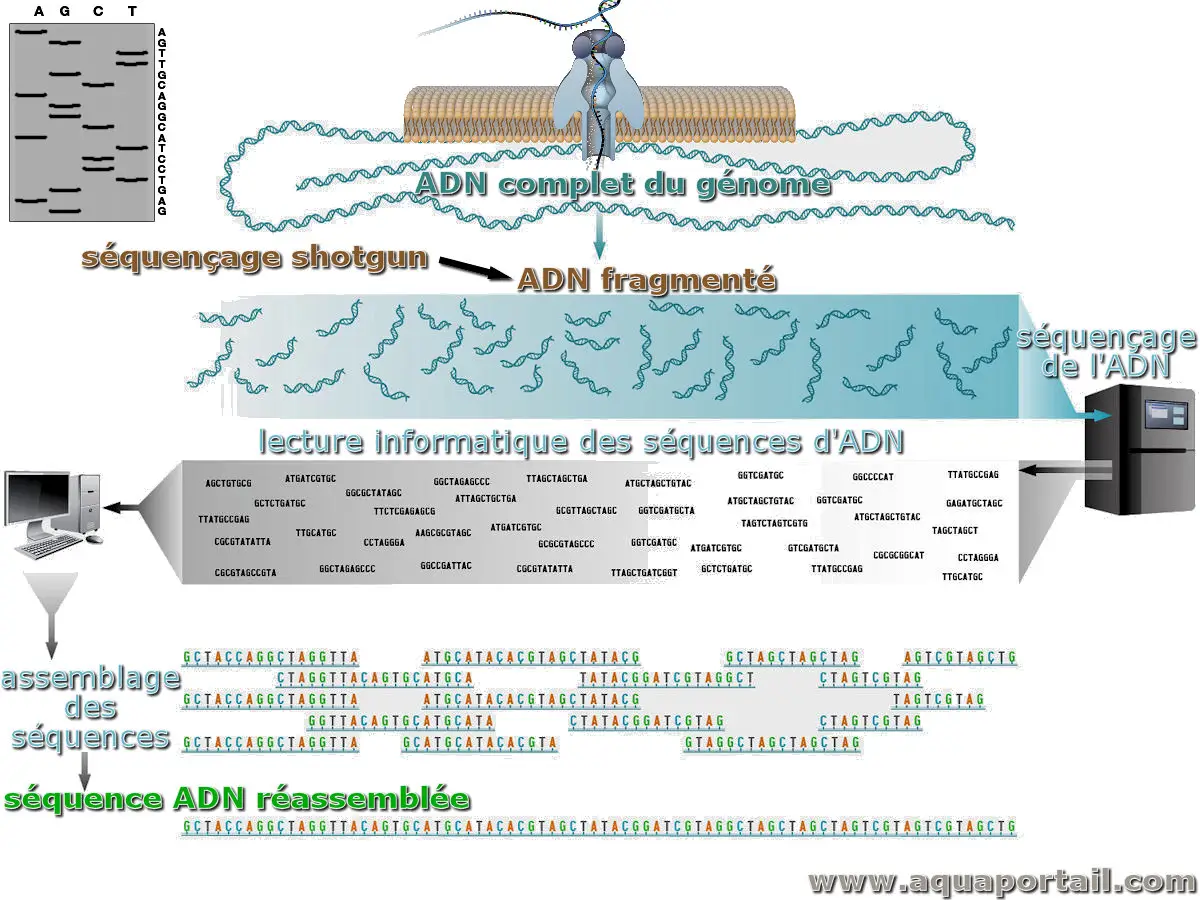
Le séquençage shotgun est une technique de métagénomique utilisée pour déterminer la séquence d'ADN du génome d'un organisme.

Une action génétique en dominance est un type d'action génétique où un allèle l'emporte sur l'autre, parfois de façon complète mais d'autres fois, de façon...
