Séquençage de l'ADN
Définition
Le séquençage de l'ADN consiste à déterminer l'ordre des quatre blocs de construction chimiques (appelés "bases") qui composent la molécule d'ADN. La séquence indique le type d'information génétique (signature ADN) contenue dans un segment d'ADN particulier.
Exemple de séquençage de l'ADN :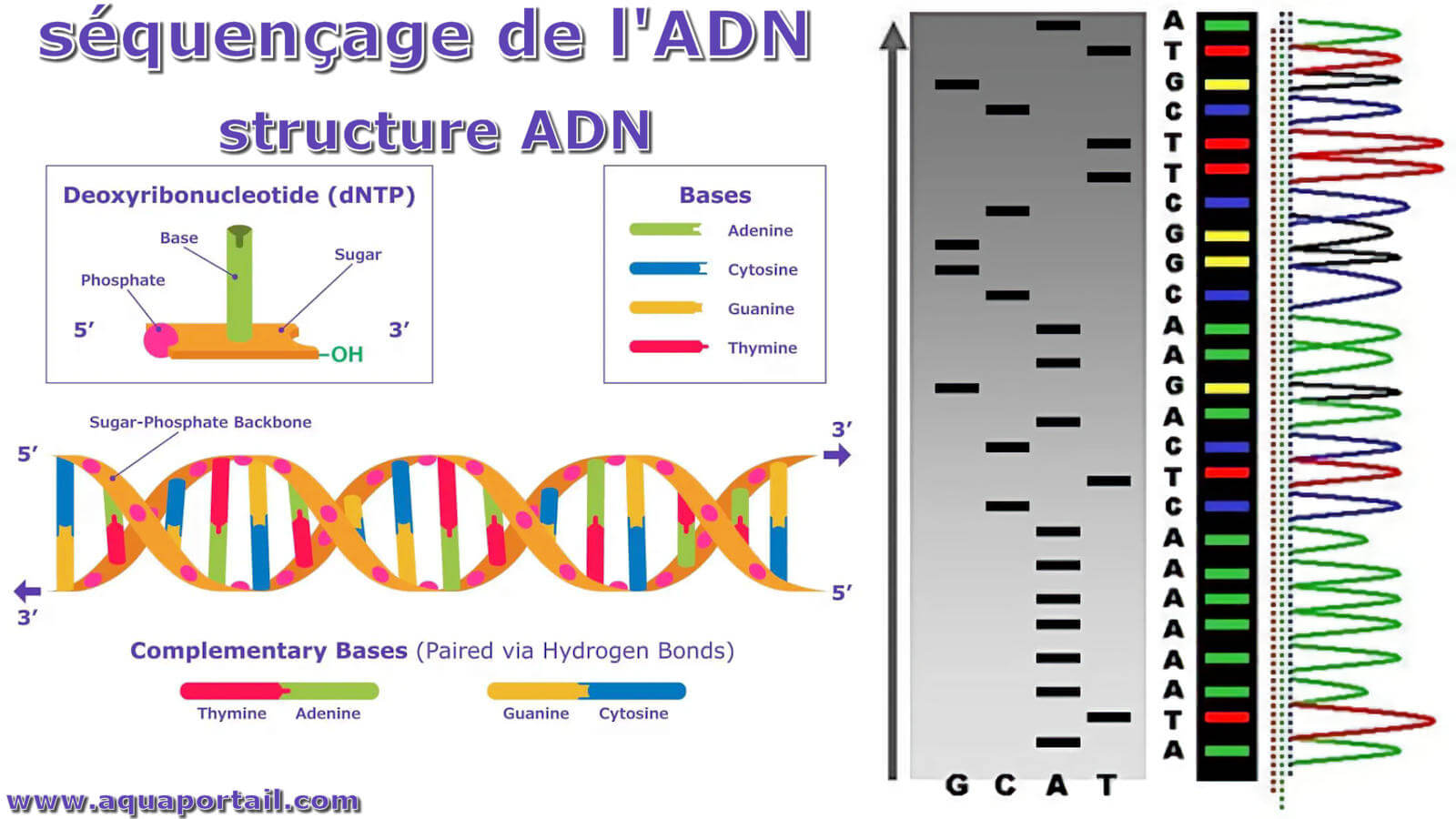
Le séquençage de l'ADN est la technique utilisée pour déterminer la séquence nucléotidique de l'ADN (séquence génétique de l'acide désoxyribonucléique). La séquence nucléotidique est le niveau de connaissance le plus fondamental d'un gène ou d'un génome.
Explications
Le séquençage de l'ADN est une méthode utilisée pour déterminer l'ordre précis des quatre bases nucléotidiques - adénine, guanine, cytosine et thymine - qui composent un brin d'ADN. Ces bases fournissent la base génétique sous-jacente (le génotype) pour dire à une cellule quoi faire, où aller et quel type de cellule devenir (le phénotype).
L'analyse de séquences d'ADN devient de plus en plus importante dans les sciences de la vie et de la santé. La méthode de séquençage enzymatique développée par Sanger et ses collègues et la méthode chimique développée par Maxam et Gilbert sont couramment utilisées pour séquencer l'ADN.
Le séquençage Sanger, également connu sous le nom de "méthode de terminaison de chaîne", est une méthode permettant de déterminer la séquence nucléotidique de l'ADN. La méthode a été développée par le double lauréat du prix Nobel Frederick Sanger et ses collègues en 1977, d'où le nom de séquence de Sanger.
Le séquençage Sanger peut être effectué manuellement ou, plus communément, de manière automatisée via une machine de séquençage. Chaque méthode suit trois étapes de base, décrites ci-dessous.
La séquence d'ADN d'intérêt (gène d'intérêt) est utilisée comme matrice pour un type spécial de PCR appelée PCR de terminaison de chaîne. La PCR de terminaison de chaîne fonctionne exactement comme la PCR standard, mais avec une différence majeure : l'ajout de nucléotides modifiés (dNTP) appelés didésoxyribonucléotides (ddNTP). Dans l'étape d'extension de la PCR standard, l'ADN polymérase ajoute des dNTP à un brin d'ADN en croissance en catalysant la formation d'une liaison phosphodiester entre le groupe 3′-OH libre du dernier nucléotide et le 5′-phosphate du suivant.
Dans la PCR de terminaison de chaîne, l'utilisateur mélange un faible rapport de ddNTP de terminaison de chaîne avec les dNTP normaux dans la réaction de PCR. les ddNTP n'ont pas le groupe 3′-OH requis pour la formation de la liaison phosphodiester; par conséquent, lorsque l'ADN polymérase incorpore un ddNTP au hasard, l'extension cesse. Le résultat de la PCR de terminaison de chaîne est des millions à des milliards de copies d'oligonucléotides de la séquence d'ADN d'intérêt, terminées à des longueurs aléatoires (n) par des 5′-ddNTP.
Dans le séquençage Sanger manuel, quatre réactions PCR sont configurées, chacune avec un seul type de ddNTP (ddATP, ddTTP, ddGTP et ddCTP) mélangés. Dans le séquençage Sanger automatisé, tous les ddNTP sont mélangés dans une seule réaction, et chacun des les quatre dNTP ont un marqueur fluorescent unique.
Dans la deuxième étape, les oligonucléotides à terminaison de chaîne sont séparés par taille par électrophorèse sur gel. Dans l'électrophorèse sur gel, des échantillons d'ADN sont chargés dans une extrémité d'une matrice de gel et un courant électrique est appliqué. L'ADN est chargé négativement, de sorte que les oligonucléotides seront tirés vers l'électrode positive du côté opposé du gel. Étant donné que tous les fragments d'ADN ont la même charge par unité de masse, la vitesse à laquelle les oligonucléotides se déplacent ne sera déterminée que par la taille. Plus un fragment est petit, moins il subira de friction lorsqu'il se déplacera dans le gel et plus il se déplacera rapidement. En conséquence, les oligonucléotides seront disposés du plus petit au plus grand, lisant le gel de bas en haut.
Dans le séquençage Sanger manuel, les oligonucléotides de chacune des quatre réactions PCR sont exécutés dans quatre voies distinctes d'un gel. Cela permet à l'utilisateur de savoir quels oligonucléotides correspondent à chaque ddNTP.
Dans le séquençage Sanger automatisé, tous les oligonucléotides sont exécutés dans une seule électrophorèse sur gel capillaire dans la machine de séquençage.
La dernière étape consiste simplement à lire le gel pour déterminer la séquence de l'ADN d'entrée. Étant donné que l'ADN polymérase ne synthétise l'ADN que dans la direction 5′ vers 3′ à partir d'une amorce fournie, chaque ddNTP terminal correspondra à un nucléotide spécifique dans la séquence d'origine (par exemple, le fragment le plus court doit se terminer au premier nucléotide à partir de l'extrémité 5′, le deuxième fragment le plus court doit se terminer au deuxième nucléotide à partir de l'extrémité 5′, etc.) Par conséquent, en lisant les bandes de gel de la plus petite à la plus grande, nous pouvons déterminer la séquence 5′ à 3′ du brin d'ADN d'origine.
Dans le séquençage manuel de Sanger, l'utilisateur lit les quatre voies du gel à la fois, en se déplaçant de bas en haut, en utilisant la voie pour déterminer l'identité du terminal ddNTP pour chaque bande. Par exemple, si la bande inférieure se trouve dans la colonne correspondant à ddGTP, alors le plus petit fragment de PCR se termine par ddGTP, et le premier nucléotide à partir de l'extrémité 5′ de la séquence d'origine a une base guanine (G).
Dans le séquençage automatisé de Sanger, un ordinateur lit chaque bande du gel capillaire, dans l'ordre, en utilisant la fluorescence pour appeler l'identité de chaque terminal ddNTP. En bref, un laser excite les étiquettes fluorescentes dans chaque bande et un ordinateur détecte la lumière émise résultante. Étant donné que chacun des quatre ddNTP est étiqueté avec une étiquette fluorescente différente, la lumière émise peut être directement liée à l'identité du ddNTP terminal. La sortie est appelée un chromatogramme, qui montre le pic fluorescent de chaque nucléotide le long de la longueur de l'ADN matrice.
Synonymes, antonymes
Voir tous les synonymes pour "séquençage de l'ADN".0 synonyme (sens proche) pour "séquençage de l'ADN".
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à SÉQUENÇAGE DE L'ADN sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
L'expression SEQUENCAGE DE L'ADN est dans la page 2 des mots en S du lexique du dictionnaire.
Mots en S à proximité
septifrageseptomycèteseptum séqué séquençage séquençage de l'ADNséquençage du génome séquençage génomique séquençage shotgun séquence codante séquence d'ADN
En rapport avec "séquençage de l'ADN"
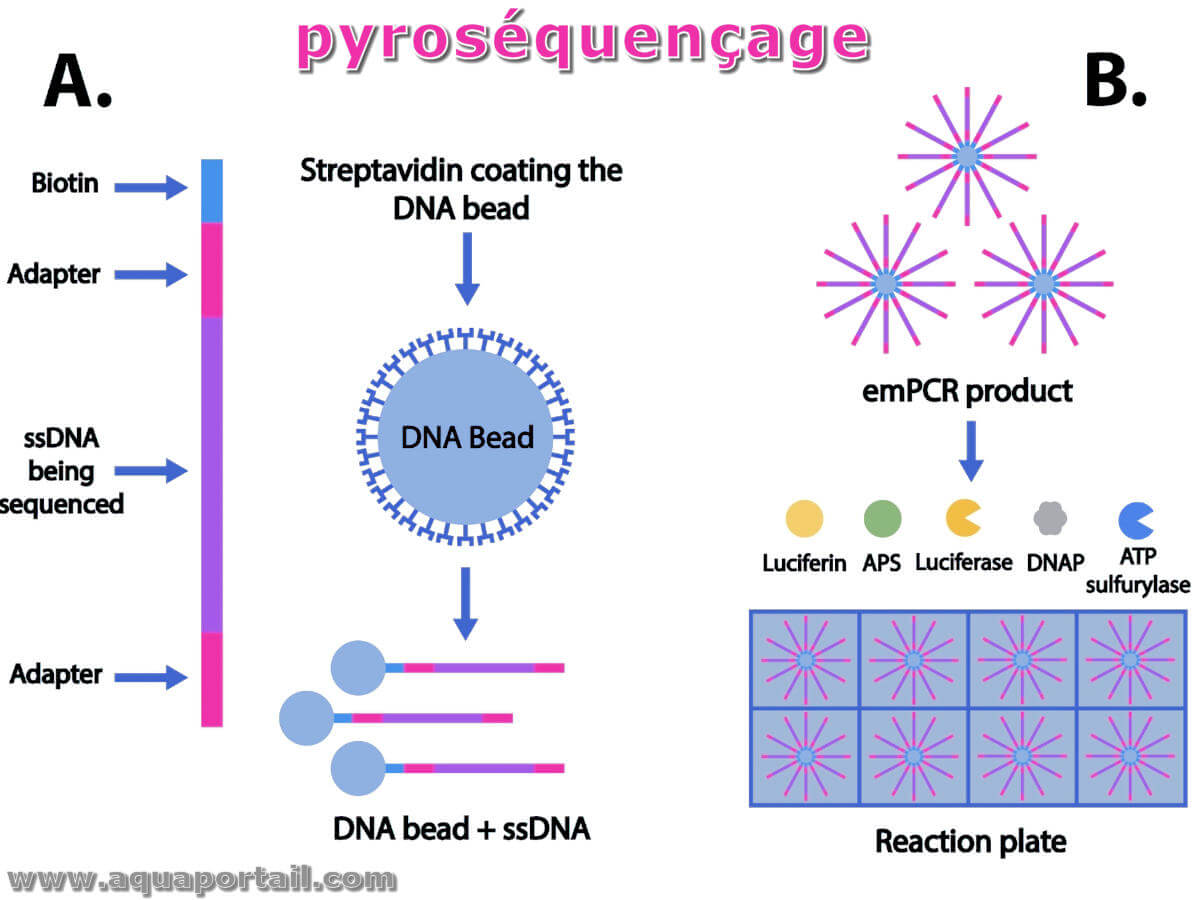
Le pyroséquençage est une technologie de séquençage de l'ADN sans gel et par synthèse, qui consiste à obtenir des informations génétiques en synthétisant...
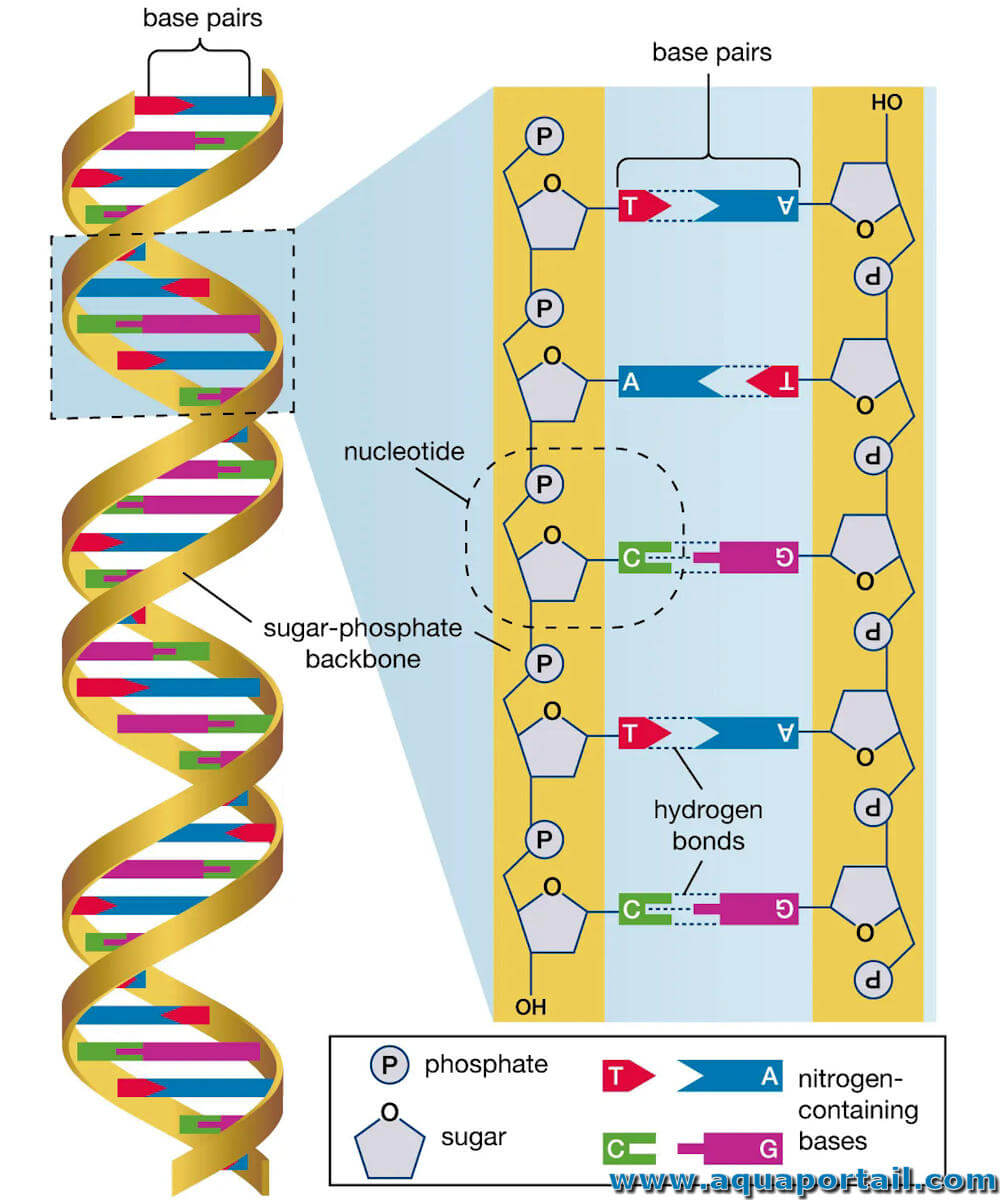
En biologie, le séquençage est la détermination de l'ordre linéaire des composants d'une macromolécule, les séquences moléculaires.

Le séquençage du génome est l'analyse du génome qui détermine la succession de toutes les bases qui composent l'ADN d'un organisme.
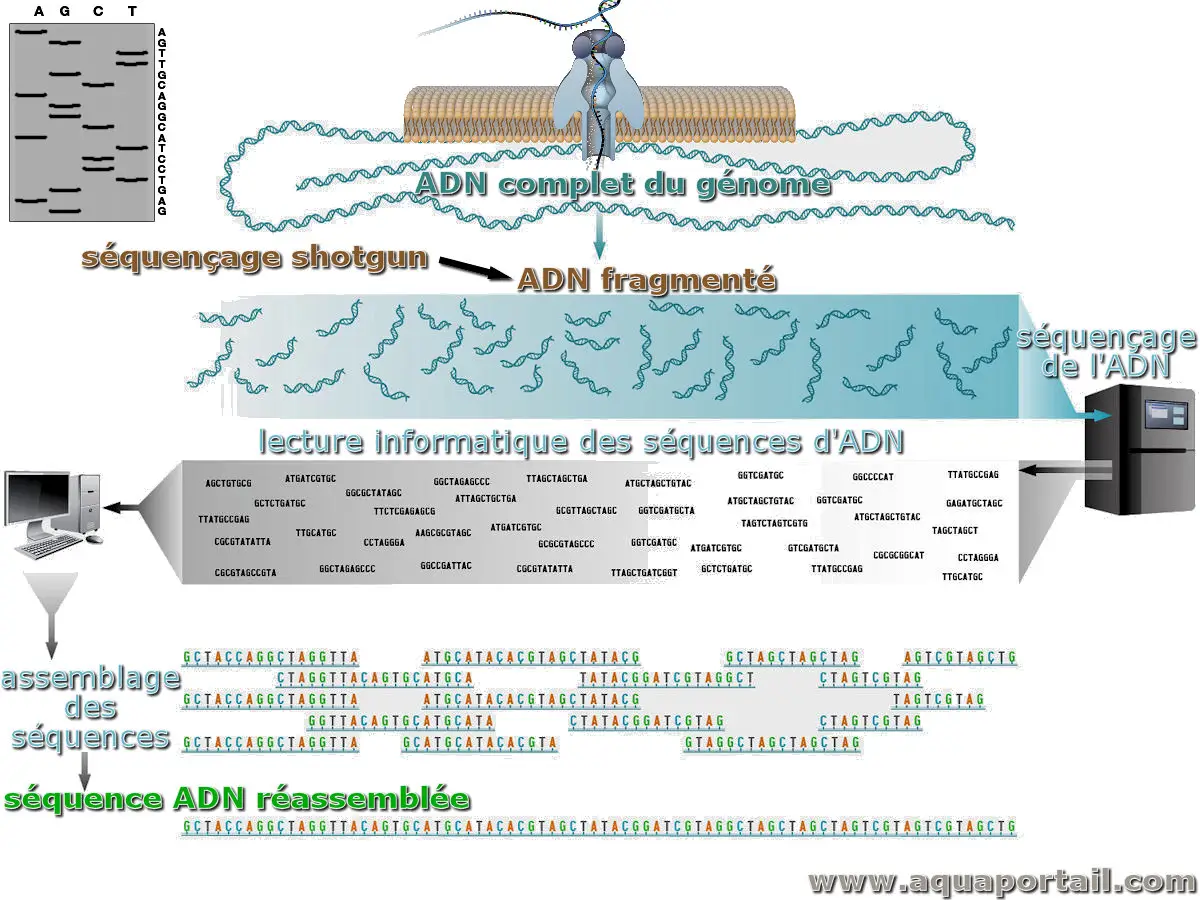
Le séquençage shotgun est une technique de métagénomique utilisée pour déterminer la séquence d'ADN du génome d'un organisme.
