ADN polymérase
Définition
L'ADN polymérase est une enzyme cellulaire ou virale qui lie les nouveaux nucléotides au brin d'ADN naissant lors du processus de la réplication de l'ADN. Elle catalyse la polymérisation d'une molécule d'ADN en ajoutant des désoxyribonucléotides à l'extrémité 3′.
Molécule d'ADN polymérase et réplication :
La molécule d'ADN polymérase humaine (structure 3D) accompagne une réplication d'ADN (processus schématique).
Explications
Les ADN polymérases effectuent la synthèse du nouveau brin d'ADN (synthèse d'ADN) en faisant correspondre les désoxyribonucléotides triphosphates (dNTP) aux désoxyribonucléotides complémentaires correspondants de l'ADN matrice. Les dNTP utilisés dans la réplication de l'ADN contiennent trois phosphates de base liés au groupe hydroxyle en 5′ du désoxyribose et, en fonction de la base azotée, ils seront dATP, dTTP, dCTP ou dGTP.
Processus et réactions
La réaction fondamentale est un transfert d'un groupe phosphate dans lequel le groupe 3′-OH joue le rôle d'un nucléophile à l'extrémité 3′ de la chaîne en croissance. L'attaque nucléophile intervient sur le phosphate α (le plus proche du désoxyribose) du désoxyribonucléoside 5′ triphosphate entrant, libérant du pyrophosphate inorganique et allongeant l'ADN (lorsqu'une nouvelle liaison phosphodiester est formée). Contrairement à la majorité des processus biologiques intervenant dans la cellule dans laquelle un seul groupe phosphate est séparé (Pi), lors de la réplication, les deux derniers groupes phosphate sont séparés, sous la forme d'un groupe pyrophosphate (PPi)...
Ce processus peut être résumé en une équation chimique : (ADN)n + dNTP (ADN)n+1 + PPi.
Bien que l'ADN polymérase ne possède qu'un seul site actif pour coupler les quatre dNTP différents, l'union correcte des paires de bases A : T, C : G est possible en fonction de la géométrie de celles-ci : si l'union est incorrecte, elle produit un déplacement de l'α-phosphate rendant plus difficile la liaison avec l'extrémité 3′-OH et ralentissant ainsi la vitesse de catalyse, de sorte que l'ADN polymérase ajoute de préférence les bases correctes.
Les ADN polymérases peuvent ajouter jusqu'à 1 000 nucléotides par seconde. Cela est dû à sa nature, c'est-à-dire au nombre de nucléotides capables d'ajouter chaque fois qu'ils sont associés à la matrice d'ADN qu'ils vont copier. Étant donné que l'addition des nucléotides est un processus qui dure quelques millisecondes, la vitesse de la catalyse dépendra du temps pendant lequel l'ADN polymérase reste attachée à l'ADN, c'est-à-dire de sa processivité.
La croissance de la chaîne intervient dans la direction 5′→3′, puisqu'un groupe 3′-OH libre est requis pour le début de la synthèse puisqu'il s'agit de celui qui effectue l'attaque nucléophile de l'α-phosphate du dNTP, de sorte que les ADN polymérases nécessitent un initiateur 3′-OH (qui peut être un ADN ou un ARN) appelé amorce synthétisée par l'ARN primase. L'extrémité 3′ de l'amorce s'appelle l'extrémité amorce.
Les ADN polymérases remplissent également d'autres fonctions au cours du processus de réplication. En plus de participer à l'élongation, ils remplissent une fonction correctrice et restauratrice grâce à leur activité 3′ exonucléase, ce qui leur permet de dégrader l'ADN d'un de ses côtés. Il est important que ces mécanismes de correction existent car sinon, les erreurs générées lors de la copie de l'ADN entraîneraient des mutations.
Dans le cas d'une ligase au lieu d'une polymérase, voir aussi l'ADN ligase.
Activité polymérase
La polymérase permet la liaison chimique de molécules individuelles (monomères) à une chaîne (polymère). Dans le cas de l'ADN polymérase, le polymère formé est l'acide désoxyribonucléique (ADN), les monomères servant de désoxyribonucléotides, plus spécifiquement de désoxynucléoside triphosphates (dNTP). L'ADN polymérase utilise toujours un simple brin d'ADN existant comme matrice pour la synthèse d'un nouveau brin complémentaire dont la séquence de nucléotides est ainsi déterminée par la matrice. Cette préservation de la séquence d'ADN est essentielle à la capacité de l'ADN polymérase de copier l'information génétique codée dans l'ADN. La copie correcte de la matrice est obtenue par appariement complémentaire des bases nucléotidiques incorporées avec les bases de la matrice ADN, à médiation par liaison hydrogène. La synthèse du nouveau brin d'ADN se fait de l'extrémité 5′ à l'extrémité 3′. D'un point de vue chimique, une attaque nucléophile du groupe 3′-hydroxyle terminal du brin d'ADN survient sur l'α-phosphate du dNTP, ce qui libère du pyrophosphate. Cette étape est catalysée par la polymérase.
Contrairement aux ARN polymérases, la synthèse du brin d'ADN complémentaire dans les ADN polymérases ne peut avoir lieu que si la polymérase a une extrémité 3′-hydroxyle libre. Le premier nucléotide est ensuite ajouté à cela. Dans le cas de la réaction en chaîne de la polymérase (PCR), on utilise un simple brin d'ADN (amorce) d'une longueur d'environ 15 à 20 nucléotides, qui sert de point de départ à la réaction. Les ADN polymérases nécessitent généralement des ions magnésium en tant que cofacteur.
Voir aussi l'ADN polymérase ARN-dépendante.
La catalyse de la formation de la liaison diester est fonctionnellement analogue à la réaction correspondante des ARN polymérases. Le dernier nucléotide de la partie déjà synthétisée et le nucléotide à attacher sont coordonnés à chacun de deux ions magnésium dans le centre catalytique du domaine de la polymérase. Le premier groupe phosphate du nucléotide à attacher est coordonné aux deux ions magnésium. La localisation spatiale permet l'attaque du groupe hydroxyle du nucléotide précédent sur le groupe phosphate de l'attaché. Dans le processus, un résidu de pyrophosphate est séparé.
Activité exonucléase
De nombreuses polymérases ont également des fonctions enzymatiques supplémentaires. En présence de faibles concentrations de dNTP, l'activité exonucléase 3′→5′ est prédominante. Certaines polymérases ont également une activité exonucléase 5′→3′. Afin de s'assurer qu'il n'y a pas d'erreur dans la lecture de la matrice d'ADN, ils ont cette fonction de vérification, c'est-à-dire qu'ils sont capables de détecter l'incorporation d'un nucléotide inapproprié, puis à nouveau. enlever de l'ADN. Cela permet la dégradation d'un brin existant d'ADN ou d'ARN déjà apparié avec le brin matrice pendant la formation d'un nouveau brin. Il en résulte un échange de l'ancien brin contre un nouveau brin. Cette activité exonucléase est exploitée dans la méthode de "traduction de Nick" (technique de marquage en biologie moléculaire).
ADN polymérases eucaryotes
Il existe 13 types bien que les plus importants soient quatre :
- ADN polymérase α (alpha) : se trouve dans le noyau de la cellule, est inhibée par l'aphidicoline et compte 4 sous-unités chez l'homme, bien que les plus importantes soient deux :
- Sous-unité principale (130 kDa) : possède une activité polymérase.
- Sous-unité mineure (48 kDa) : possède un centre de primase actif pour synthétiser l'amorce nécessaire à la réplication. Comme toutes les polymérases possèdent un domaine commun, représenté par une main, dans lequel un domaine d'exonucléase est inclus. Par conséquent, il a une faculté de correction.
- ADN polymérase σ (sigma) : se trouve dans le noyau de la cellule et est inhibée par aphidicoline. Il a une ou deux sous-unités en fonction de l'organisme. Il a une grande capacité de polymérisation et une activité de correction d'erreur (exo 3′→ 5′). Il manque d'activité primase.
La primase (qui fait partie de la molécule d'α-ADN polymérase) synthétise les amorces d'ARN et commence également l'élongation avec l'ADN des deux brins. Ensuite, un changement de polymérase intervient et l'ADN polymérase σ entre, ce qui poursuit la synthèse.
ADN polymérase-α
L'ADN polymérase-α est un complexe enzymatique impliqué dans l'initiation de la réplication de l'ADN et lors de la synthèse de fragments d'Okazaki sur le brin retardé. Le complexe ADN polymérase-α est constitué de 4 sous-unités : POLA1, POLA2, PRIM1 et PRIM2. POLA1 est la sous-unité catalytique. POLA2 est la sous-unité de régulation. PRIM1 et PRIM2 sont les petites et les grandes sous-unités de primase.
La structure tétracyclique de l'aphidicoline :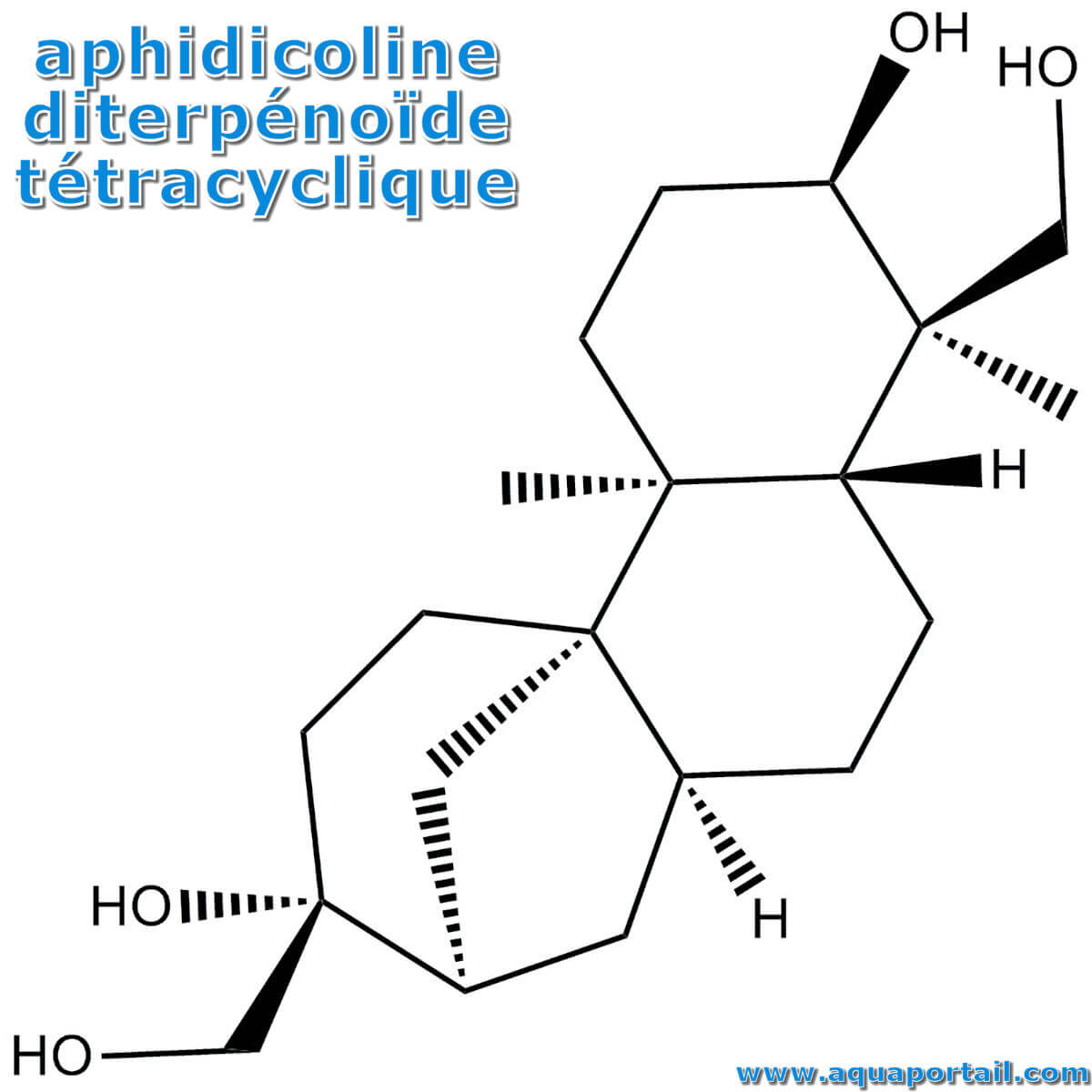
L'aphidicoline (diterpénoïde tétracyclique) est un inhibiteur de l'ADN polymérase-α avec un rôle d'antibiotique, produit par la moisissure Cephalosporium aphidicola.
L'aphidicoline a inhibé sélectivement l'activité de l'ADN polymérase-α sans activité pour l'ADN polymérase-β et l'ADN polymérase mitochondriale. L'aphidicoline a empêché la division cellulaire chez les embryons d'oursins. L'aphidicoline était un puissant inhibiteur de la synthèse cellulaire de l'acide désoxyribonucléique. L'aphidicoline a fortement inhibé la croissance du virus de l'herpès simplex à la fois dans la culture tissulaire et dans l'oeil du lapin. L'aphidicoline était active contre le virus de l'herpès résistant à l'iododésoxyuridine. L'aphidicoline a montré une activité antivirale et a inhibé l'incorporation de thymidine dans l'ADN de cellules pulmonaires embryonnaires humaines en culture. L'aphidicoline (15 μg/ml) a réduit l'activité des ADN polymérases brutes et partiellement purifiées du cytosol.
Réaction en chaîne de la polymérase
La propriété des ADN polymérases de répliquer des brins d'ADN est utilisée pour la réaction en chaîne de la polymérase, connue sous le nom de PCR pour son acronyme en anglais, afin d'obtenir un grand nombre de copies d'un fragment d'ADN particulier, en l'amplifiant à des fins de recherche. Dans les processus de PCR, l'utilisation de polymérases thermostables, telles que la Taq polymérase, est nécessaire car il est nécessaire d'appliquer des températures élevées pour dénaturer la molécule d'ADN.
Phage ADN polymérase Φ29
Le phage Φ29 synthétise sa propre ADN polymérase. Cette enzyme est largement utilisée en biologie moléculaire pour de nombreuses procédures de déplacement par amplification d'ADN et possède une série de caractéristiques qui la rendent particulièrement adaptée à cette application.
ADN polymérases de E. coli (procaryotes)
L'ADN polymérase dans les cellules procaryotes est mis en exemple avec la bactérie Escherichia coli. Les principales ADN polymérases chez E. coli sont l'ADN Pol I, l'ADN Pol II, l'ADN Pol III et chacune d'elles est spécialisée dans une ou plusieurs de ces fonctions en fonction de son rôle dans la réplication.
La processivité est liée à la durée pendant laquelle la DNApol reste attachée à l'ADN lorsqu'elle ajoute des nucléotides. Ainsi, l'ADN Pol I non transformable (ajoute entre 20 et 100 nucléotides par événement de liaison), est responsable de l'élimination des amorces et du "remplissage" de l'espace restant avec de l'ADN (activité exonucléase 5′→3′ et activité de la polymérase 5′→3′). Ce processus s'appelle la traduction du pseudo. DNA Pol II est responsable de la réparation des ruptures produites dans l'ADN (activités de la polymérase 5′→3′ et de l'exonucléase 3′→5′), l'ADN Pol III très processif est le principal responsable de l'élongation de l'ADN (Activité 5′→3′ polymérase) au cours desquelles il effectue également des tâches de correction (activité exonucléase 3′→5′). Il convient de noter que les activités de correction et de réparation ne sont pas les mêmes. Seuls les ADN pol I et bêta ont une activité de réparation, tandis que toutes les polymérases ont une activité corrective (elles ont 2 sites actifs : un site de polymérisation et un site de correction).
Pourquoi ça se passe ? Comment DNApol réalise-t-il qu'il y a eu une erreur dans la réplication de l'ADN ? Cela est dû au phénomène de tautomérisation. Les nucléotides changent de forme chimique alternative, le DNApol les confond avec un autre nucléotide et l'allonge. Lorsque cela revient à sa conformation plus stable, il en résulte une modification de la largeur du brin (20A) et une ouverture finale. Ceci est détecté par la polymérase, qui change de conformation et laisse le brin positionné dans le site de correction.
Les ADN Pol IV et V, identifiés en 1999, sont impliqués dans une forme peu commune de réparation de l'ADN.
Contrairement à ce qui se passe avec l'ADN Pol I, qui ne devrait ajouter que 5 à 10 nucléotides une fois que l'amorce a été retirée, il est important que l'ADN Pol III soit très processif et, pour cette raison, il fait généralement partie d'un complexe appelé holoenzyme Pol III ADN qui lui donne une plus grande processivité. Ce complexe est constitué de plusieurs sous-unités polypeptidiques responsables de chaque fonction et qui, ensemble, constituent un dimère asymétrique : une moitié est responsable de la synthèse du brin direct et l'autre moitié est en retard. Le noyau catalytique est composé des sous-unités α qui correspondent à deux copies de l'ADN Pol III, de la sous-unité ε avec l'activité de correction exonucléase 3′→5′ et de la sous-unité θ dont la fonction pourrait être d'assembler les deux autres; les sous-unités τ maintiennent la structure dimérique; le complexe γ est constitué des sous-unités γ et δ dont la fonction est d'augmenter la processivité de l'ADN de Pol III; la sous-unité β lie l'ADN de Pol III à l'ADN.
Importance biologique
Les ADN polymérases jouent un rôle central dans la réplication de l'ADN. Ils permettent la copie fidèle d'informations génétiques sous forme d'ADN, ce qui constitue une étape décisive dans la propagation et la reproduction des êtres vivants. De plus, les ADN polymérases jouent un rôle important dans les processus impliqués dans la réparation de l'ADN.
Synonymes, antonymes
Voir tous les synonymes pour "ADN polymérase".1 synonyme (sens proche) de "aDN polymérase" :
2 antonymes (sens contraire) :
Les mots ou les expressions apparentés à ADN POLYMÉRASE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
L'expression ADN POLYMERASE est dans la page 3 des mots en A du lexique du dictionnaire.
Mots en A à proximité
ADN de transfert ADN environnemental ADN génomique ADN ligase ADN mitochondrial ADN polyméraseADN polymérase ARN-dépendante ADN recombinant ADN topoisomérase ADN-T adnation
En rapport avec "ADN polymérase"
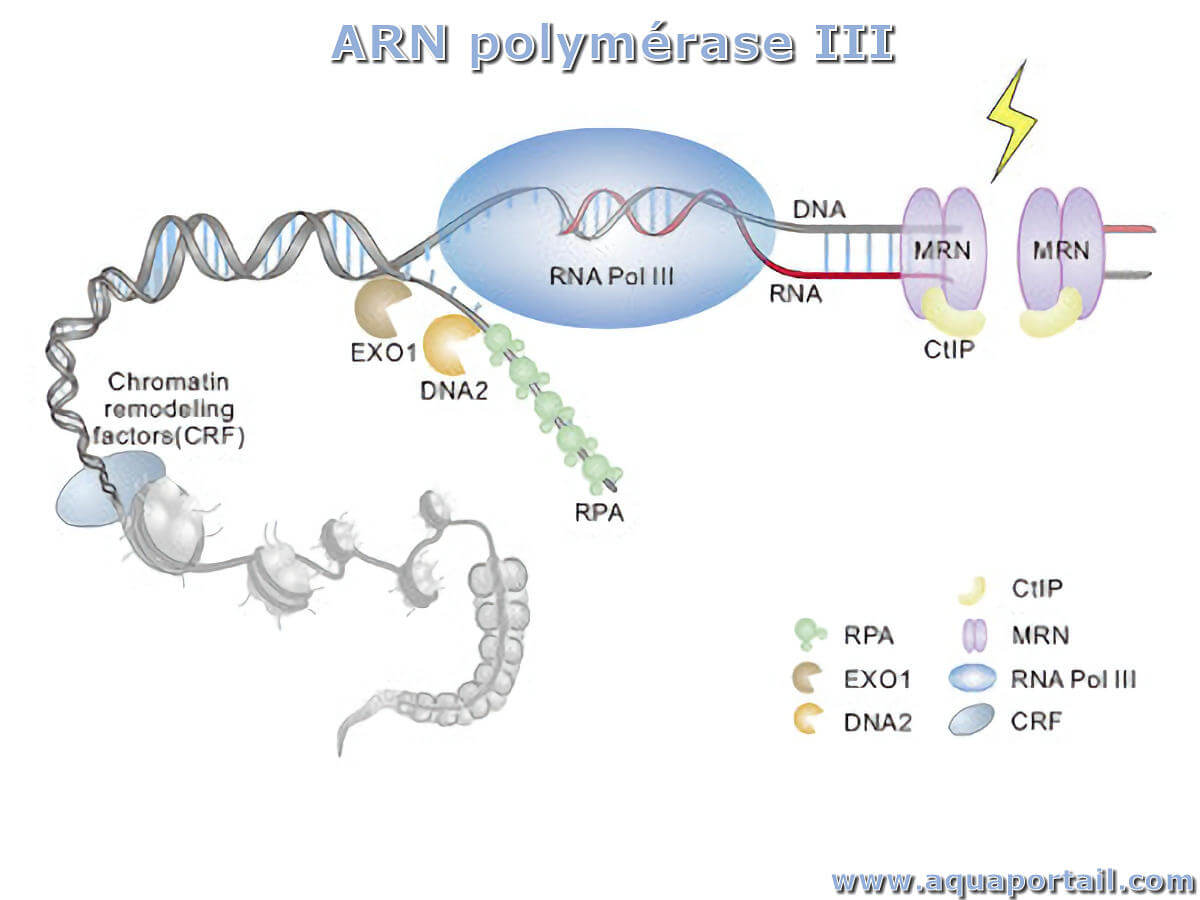
Une ARN polymérase (ARNP) est une enzyme qui se lie aux promoteurs de l'ADN puis catalyse la synthèse des ARN lors de la transcription.
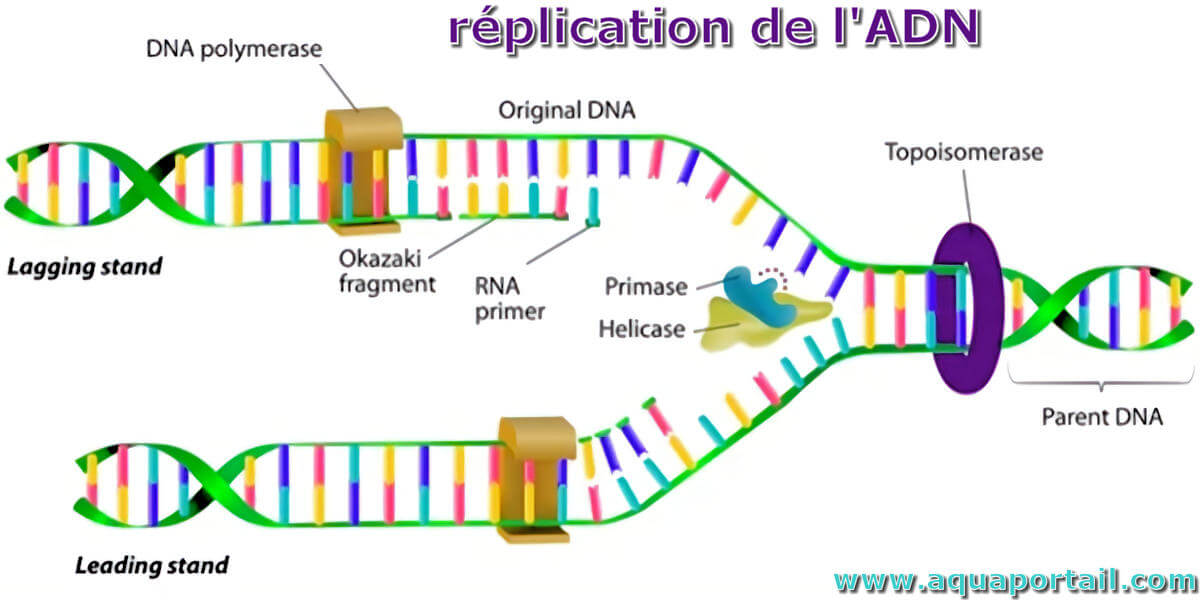
Une réplication est l'ensemble des processus qui permettent de reproduire à l'identique un ADN.
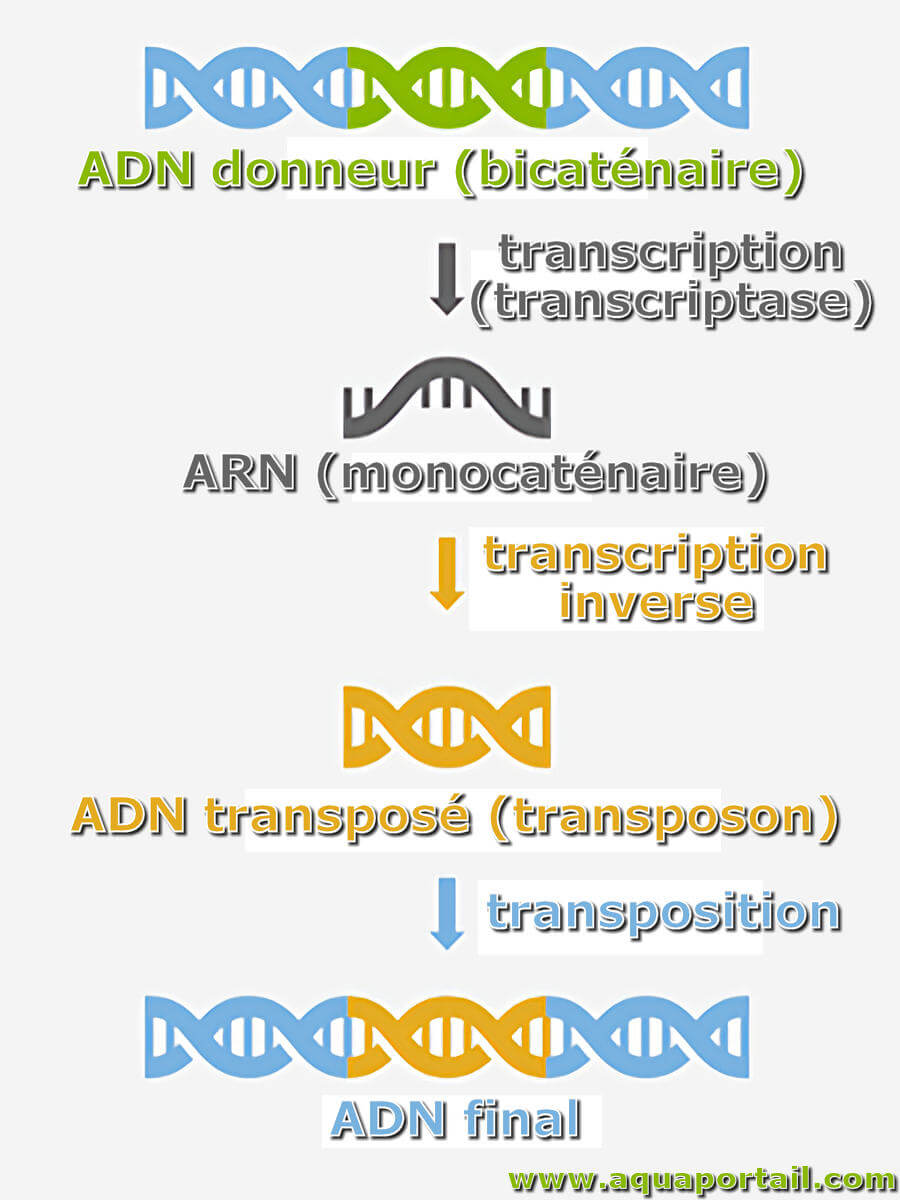
L'activité transcriptase au sein d'un virion assemblé catalyse la synthèse de l'ARN à brin positif descendant à partir de la matrice d'ARN double brin...
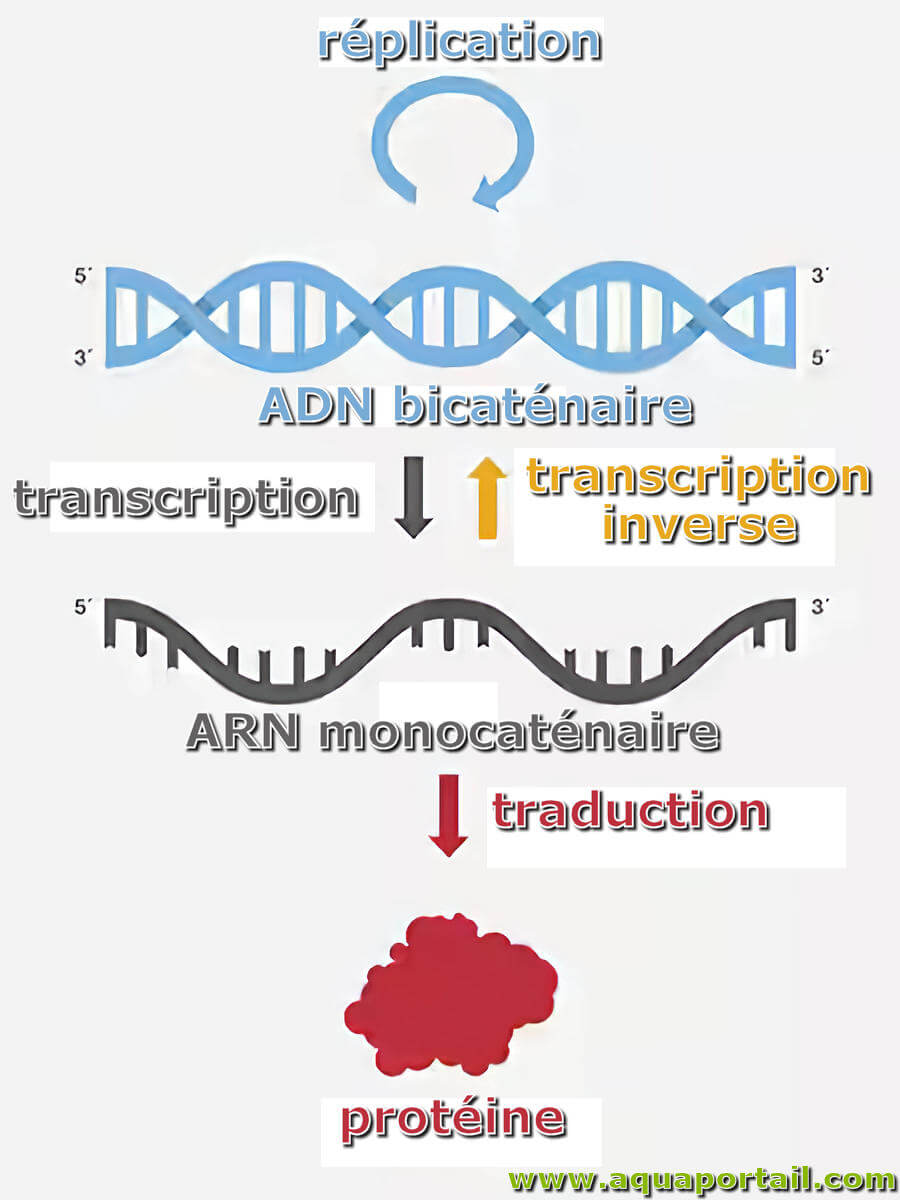
La transcriptase inverse, également appelée rétrotranscriptase et ADN polymérase ARN-dépendante, est une ADN polymérase qui transcrit l'ARN simple brin...
