Endonucléase
Définition
Une endonucléase est une nucléase qui se passe à l'intérieur, en dedans, par hydrolysation. Les endonucléases sont des enzymes qui catalysent la dégradation des liaisons phosphodiester dans différentes régions situées à l'intérieur d'une chaîne polynucléotidique. Cela les différencie des exonucléases, qui catalysent le clivage des liaisons phosphodiester aux extrémités des chaînes.
Les enzymes de restriction créent des endonucléases :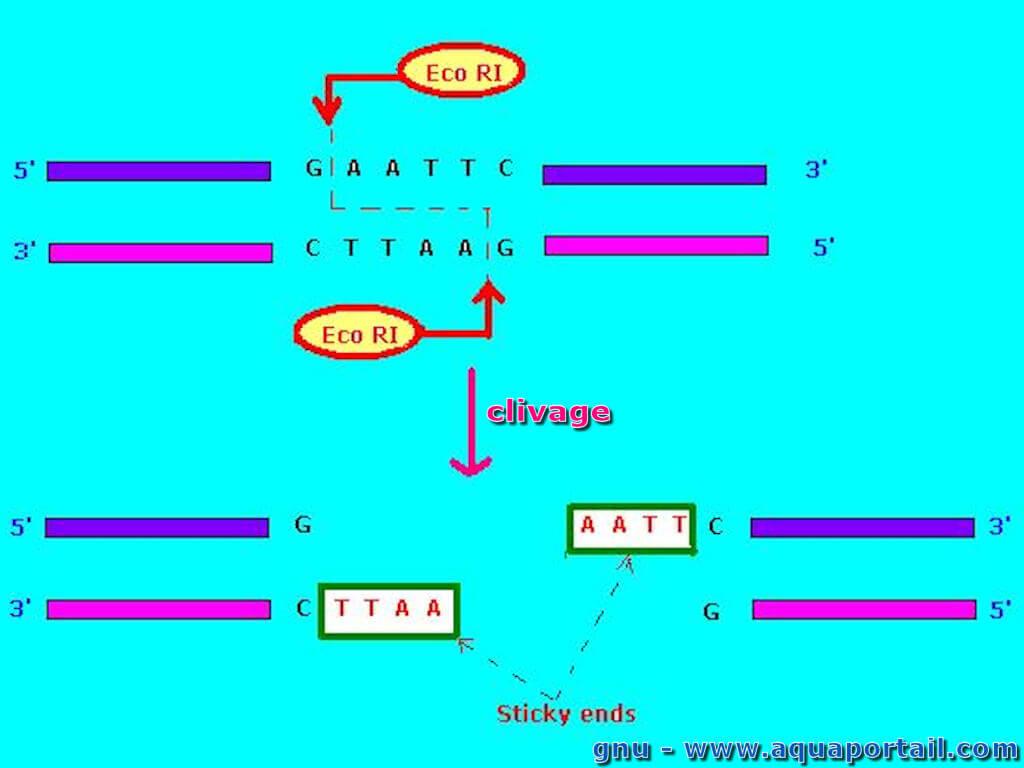
Les enzymes de restriction sont aussi appelées des endonucléases restrictives par leur rôle de bases.
Explications
Certaines endonucléases, telles que la désoxyribonucléase I, coupent l'ADN de manière relativement non spécifique (sans tenir compte de la séquence de base), alors que de nombreuses molécules, appelées enzymes de restriction ou endonucléases de restriction, ont des séquences nucléotidiques très spécifiques.
Enzymes de restriction
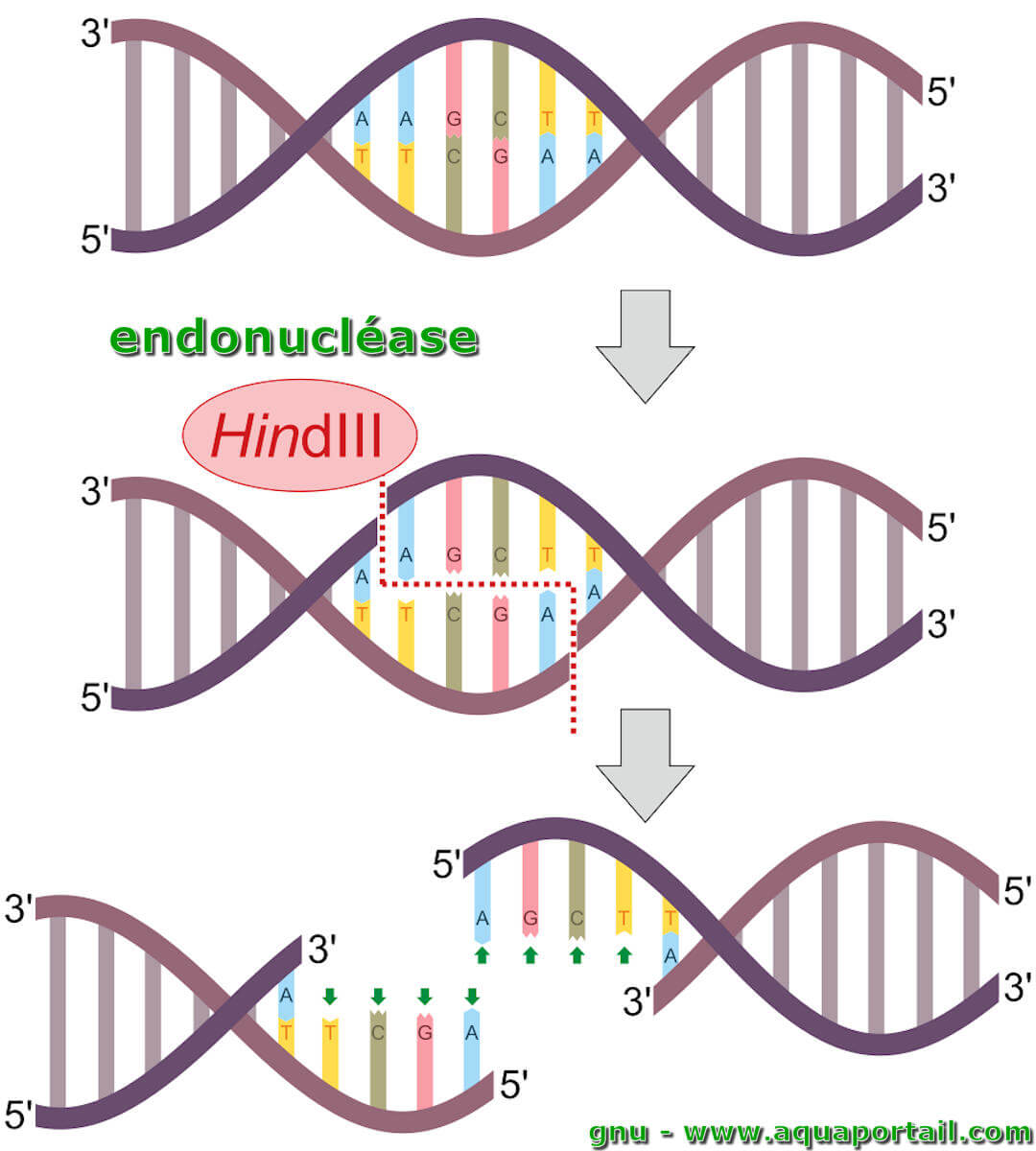
Les enzymes de restriction sont des endonucléases d'eubactéries et d'archées qui reconnaissent des séquences d'ADN très spécifiques. La séquence nucléotidique reconnue pour le clivage par une enzyme de restriction est appelée site de restriction. Typiquement, un site de restriction est une séquence palindromique d'une longueur de quatre à six nucléotides. La plupart des endonucléases de restriction clivent la molécule d'ADN double brin de manière inégale, laissant des extrémités monocaténaires complémentaires. Ces extrémités peuvent ensuite être reconnectées via un processus d'hybridation, elles sont donc appelées "extrémités adhésives". Une fois appariées, les liaisons phosphodiester des fragments peuvent être liées au moyen d'une ADN ligase.
Il existe des centaines d'endonucléases de restriction connues, chacune attaquant un site de restriction différent. Les extrémités de l'ADN clivées par la même endonucléase de restriction peuvent être appariées indépendamment de l'origine de l'ADN. Ces molécules d'ADN formées par des morceaux d'origines différentes sont appelées ADN recombinant; c'est-à-dire l'ADN formé par l'union de différents gènes pour créer de nouvelles combinaisons. Les endonucléases de restriction (ou enzymes de restriction) sont divisées en trois catégories, le type I, le type II et le type III, en fonction de leur mécanisme d'action.
Ces enzymes sont fréquemment utilisées en génie génétique pour produire de l'ADN recombinant pour une introduction ultérieure dans des cellules bactériennes, végétales ou animales; ainsi qu'en biologie synthétique.
Catégories
En fin de compte, il existe trois catégories d'endonucléases de restriction qui contribuent relativement à l'excision de séquences spécifiques. Les types I et III sont de grands complexes multi-sous-unités qui comprennent à la fois une activité d'endonucléase et une activité méthylase.
Les endonucléases de type I peuvent cliver l'ADN à des emplacements aléatoires, 1 000 paires de bases ou plus, loin de la séquence de reconnaissance, et nécessitent l'ATP comme source d'énergie.
Les endonucléases de type II se comportent légèrement différemment, ont été isolées par Hamilton Smith en 1970, sont les versions les plus simples des endonucléases et ne nécessitent pas d'ATP pour leur fonction. Certains exemples d'endonucléases de type II sont BamHI, EcoRI, EcoRV et HaeIII.
Les endonucléases de type III ne coupent pas l'ADN à plus de 25 paires de bases de la séquence de reconnaissance et nécessitent également de l'ATP dans le processus.
Notations
La notation des bases la plus utilisée pour les endonucléases de restriction est de la forme "VwxYZ", où "Vwx" correspond aux initiales de la nomenclature binomiale du mode de vie dont elle est issue (bactéries), "Y" est le nom du souche (et c'est facultatif); et "Z" (en chiffres romains) indique différentes enzymes de restriction appartenant au même mode de vie. Ainsi, par exemple, "EcoRI" signifie que l'endonucléase appartient à Escherichia coli ("Eco"); la souche est RY13 ("R"), et c'est la première découverte dans ce mode de vie : numéro "I".
Un autre exemple : "HaeII" et "HaeIII" se réfèrent à la bactérie Haemophilus aegyptius, tandis que les enzymes sont respectivement numéro II et numéro III. Les enzymes de restriction utilisées en biologie moléculaire reconnaissent généralement une courte séquence cible d'environ 4 à 8 paires de bases. Par exemple, EcoRI reconnaît et clive la séquence 5′-G.AATTC-3′.
Une endonucléase nécessite généralement un site de reconnaissance et une séquence de clivage (typiquement des bases nucléotidiques : A, C, G, T). Si le site de reconnaissance est en dehors de la région de clivage, alors on dit que l'endonucléase est de type I. Si la séquence de reconnaissance chevauche la séquence de clivage, alors l'endonucléase est considérée comme une enzyme de restriction et est type II.
Réparation de l'ADN
Les endonucléases peuvent cliver l'ADN double brin (ADNdb), ou l'ADN simple brin (ADNc), ou même l'ARN. Le présent article se concentre sur les endonucléases qui agissent sur ADNcd. De plus, les recherches actuelles visent le développement d'endonucléases de restriction synthétiques ou artificielles, ayant spécifiquement un site de reconnaissance unique dans un génome.
Les endonucléases de restriction ou les enzymes de restriction clivent typiquement les brins d'acide nucléique de deux manières différentes : avec des extrémités franches ou avec des extrémités adhésives.
Les endonucléases jouent un rôle important dans la réparation de l'ADN. Plus précisément, les endonucléases AP catalysent la dégradation de l'ADN exclusivement dans les sites AP (sites apuriniques-apyrimidines, c'est-à-dire les sites dans lesquels la base azotée a été perdue, mais la colonne phosphate-sucre est conservée) et préparent donc ADN pour l'excision ultérieure, synthèse réparatrice et ligaturée. Par exemple, lorsqu'une dépurinisation intervient, cette lésion laisse le sucre désoxyribose sans sa base azotée. L'endonucléase AP reconnaît ce sucre et coupe essentiellement l'ADN sur ce site, permettant ainsi à la réparation de l'ADN de se poursuivre plus tard. Les cellules d'E. coli contiennent deux endonucléases AP : l'endonucléase IV (endoIV) et l'exonucléase III (exoIII). Alors que chez les eucaryotes, il n'y a qu'un seul type d'endonucléase AP.
Mutations
Xeroderma pigmentosum est une maladie génétique rare, autosomique récessive, causée par une endonucléase UV spécifique. Les patients porteurs de cette mutation sont incapables de réparer les dommages à l'ADN causés par la lumière solaire.
L'anémie falciforme est une maladie provoquée par une mutation ponctuelle. La séquence modifiée par la mutation élimine le site de reconnaissance de l'endonucléase de restriction MstII.
Des mutations dans les endonucléases d'épissage d'ARNt provoquent une hypoplasie pontocérébelleuse. Les hypoplasies pontocérébelleuses (HPC) représentent un groupe de maladies génétiques autosomiques récessives causées par des mutations dans trois des quatre sous-unités de l'endonucléase ARNt qui constituent le complexe d'épissage.
Voir aussi la ribonucléase, ou encore les méganucléases.
Endonucléase apurinique/apyrimidinique humaine (APE1)
L'endonucléase apurinique/apyrimidinique (AP) humaine APE1 est l'une des enzymes clés participant à la réparation des dommages causés à l'ADN. Le rôle principal d'APE1 est l'initiation de la réparation des sites AP en catalysant l'incision hydrolytique de la liaison phosphodiester immédiatement en 5′ de la lésion.
En plus de l'activité AP-endonucléase, APE1 possède une activité exonucléase 3′-5′, qui est vraisemblablement responsable du nettoyage des extrémités 3′ non conventionnelles générées à la suite de dommages à l'ADN ou en tant qu'intermédiaires de transition dans les voies de réparation de l'ADN. Le mécanisme cinétique de l'élimination des nucléotides de l'extrémité 3′ dans le processus d'exonucléase 3′-5′ catalysé par APE1 a été étudié dans des conditions pré-stationnaires.
Les substrats d'ADN étaient des duplex de désoxyribonucléotides avec une extrémité 5′ pendante et il contenait un résidu 2-aminopurine fluorescent en 1ère, 2ème, 4ème ou 6ème position à partir de l'extrémité 3′ de l'oligonucléotide court. L'impact du nucléotide de l'extrémité 3′, qui contenait des bases non appariées, non endommagées ou des bases modifiées ainsi qu'un site abasique ou un groupe phosphate, sur l'efficacité de l'activité exonucléase 3′-5′ a été déterminé. Les données cinétiques ont révélé que l'étape limitante de l'élimination des nucléotides 3′ par APE1 dans le processus d'exonucléase 3′-5′ est la libération du nucléotide détaché du site actif de l'enzyme.
L'endonucléase apurinique/apyrimidinique humaine 1 (APE1), également connue sous le nom de facteur redox 1 (Ref-1) est une enzyme multifonctionnelle. La fonction physiologique principale d'APE1 est l'incision de la liaison phosphodiester immédiatement en 5′ vers un site apurinique/apyrimidinique (AP), qui génère une cassure simple brin avec des extrémités 5′-désoxyribose phosphate et 3′-hydroxyle. APE1 possède quelques activités mineures : 3′-5′ exonucléase, 3′-phosphodiestérase, 3′-phosphatase et RNase H. En plus de ces activités, APE1 peut reconnaître et convertir certains nucléotides endommagés ou modifiés tels que la 5,6-dihydrouridine, -anomères, dérivés éthéno, photo-adduits volumineux, dérivés benzéniques et 2′-désoxyuridine. Ce type d'activité APE1 a été nommé activité de "réparation d'incision nucléotidique" (NIR). L'hétérogénéité de la spécificité de substrat de l'enzyme a pu être élucidée par des données structurelles.
Synonymes, antonymes
Voir tous les synonymes pour "endonucléase".1 synonyme (sens proche) de "endonucléase" :
1 antonyme (sens contraire) :
Les mots ou les expressions apparentés à ENDONUCLÉASE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot ENDONUCLEASE est dans la page 3 des mots en E du lexique du dictionnaire.
Mots en E à proximité
endomixie endomorphine endomycorhize endomysium endonèvre endonucléaseendoparasite endopeptidase endopéridium endophage endophénotype
En rapport avec "endonucléase"

Une désoxyribonucléase est une enzyme qui catalyse l'hydrolyse des chaînes moléculaires d'acide désoxyribonucléique (ADN) en chaînes moléculaires plus courtes...
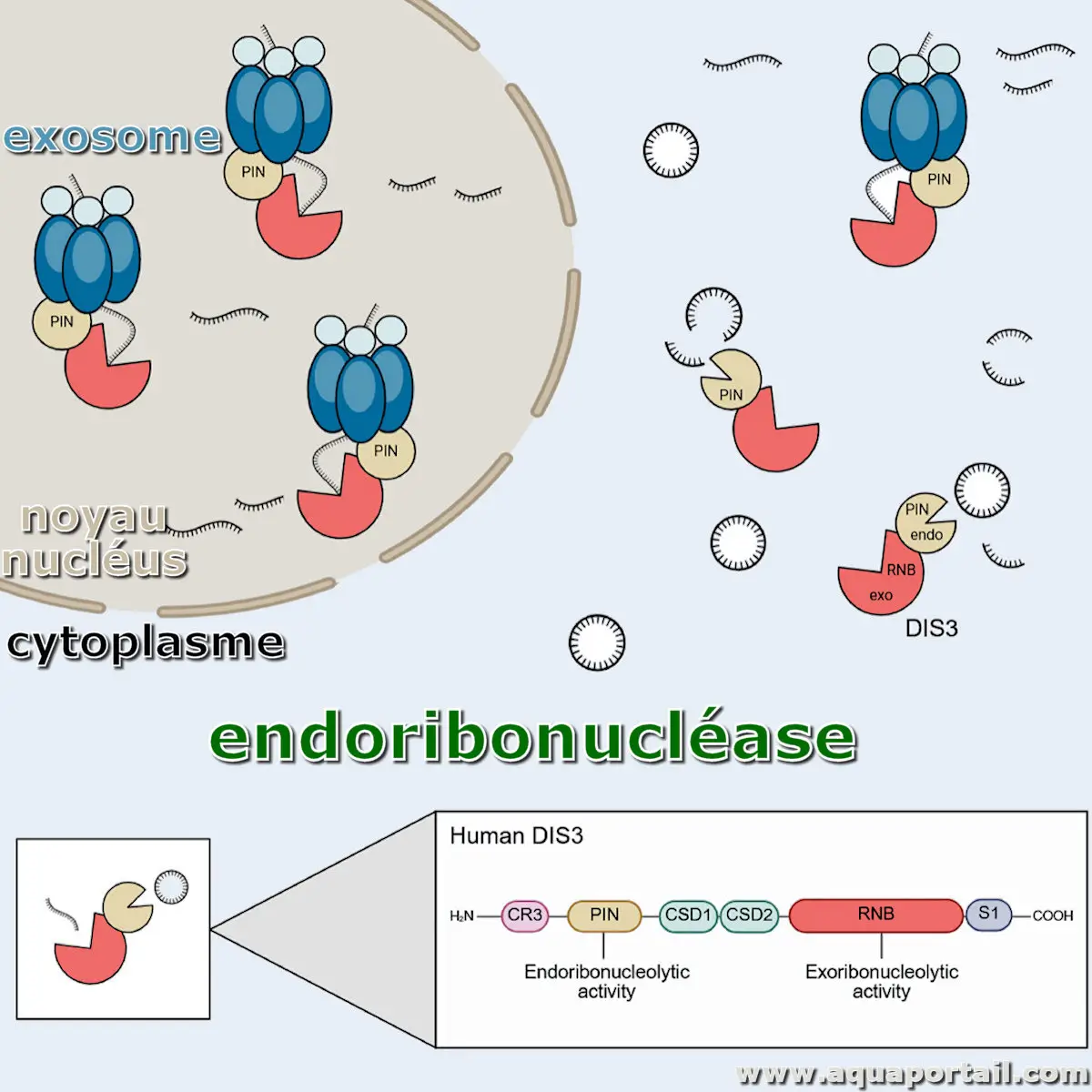
Une endoribonucléase est une enzyme appartenant au groupe plus large des ribonucléases, spécialisée dans le clivage de l'ARN.
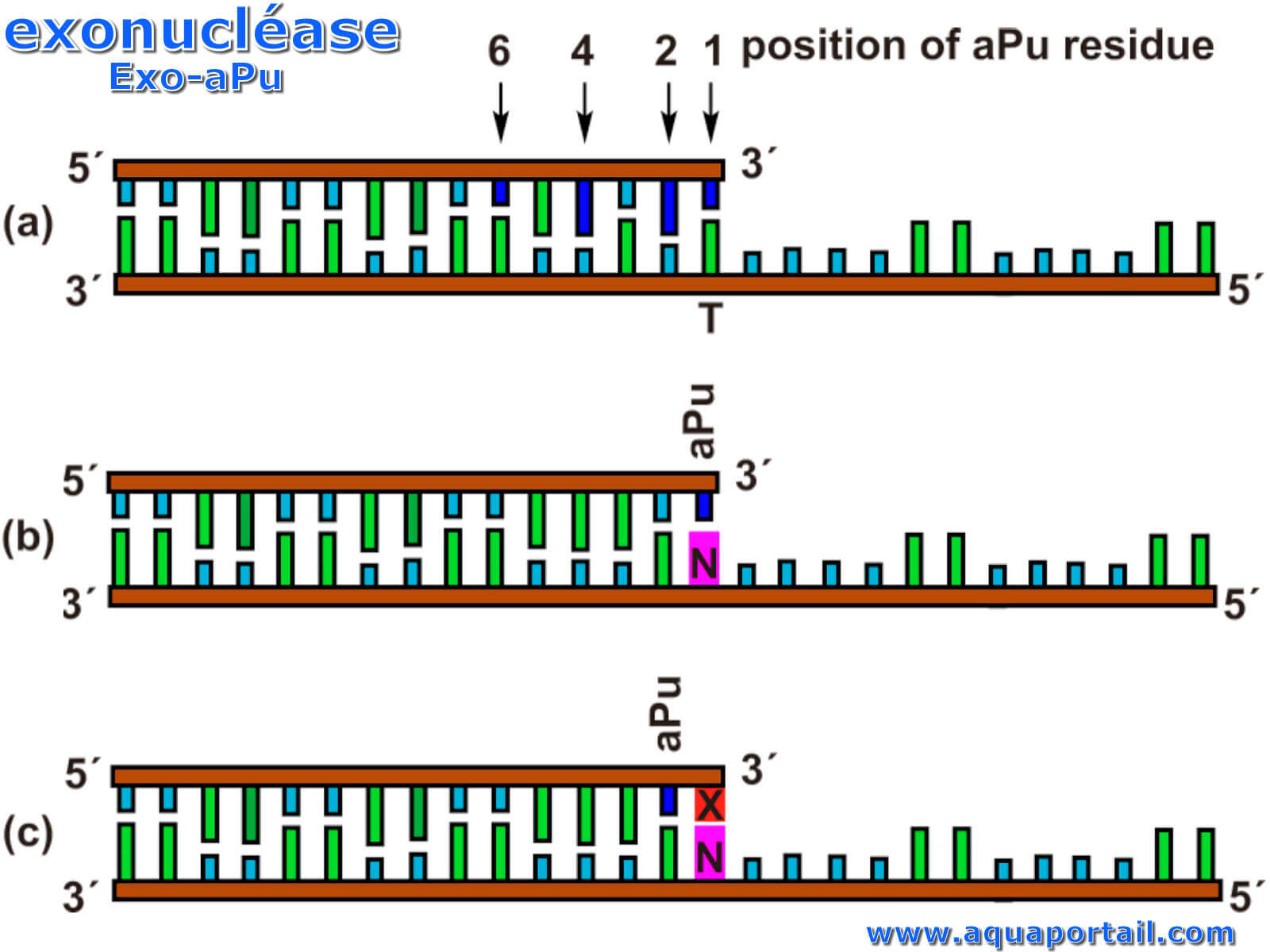
L'exonucléase distingue la partie qui est au-dehors de la nucléase. Les exonucléases sont des enzymes qui fonctionnent en coupant les nucléotides...
Les nucléases sont un groupe d'enzymes hydrolases, dont la fonction principale est la dégradation partielle ou totale des acides nucléiques.
