La dégradation de l'ARN
La dégradation de l'ARN joue un rôle fondamental dans le maintien de l'homéostasie cellulaire, pour intervenir en tant que mécanisme de surveillance éliminant les ARNm aberrants ou lors du traitement de l'ARN pour générer des transcrits matures.
La dégradation de l'ARNm et la maturation de l'ARNr :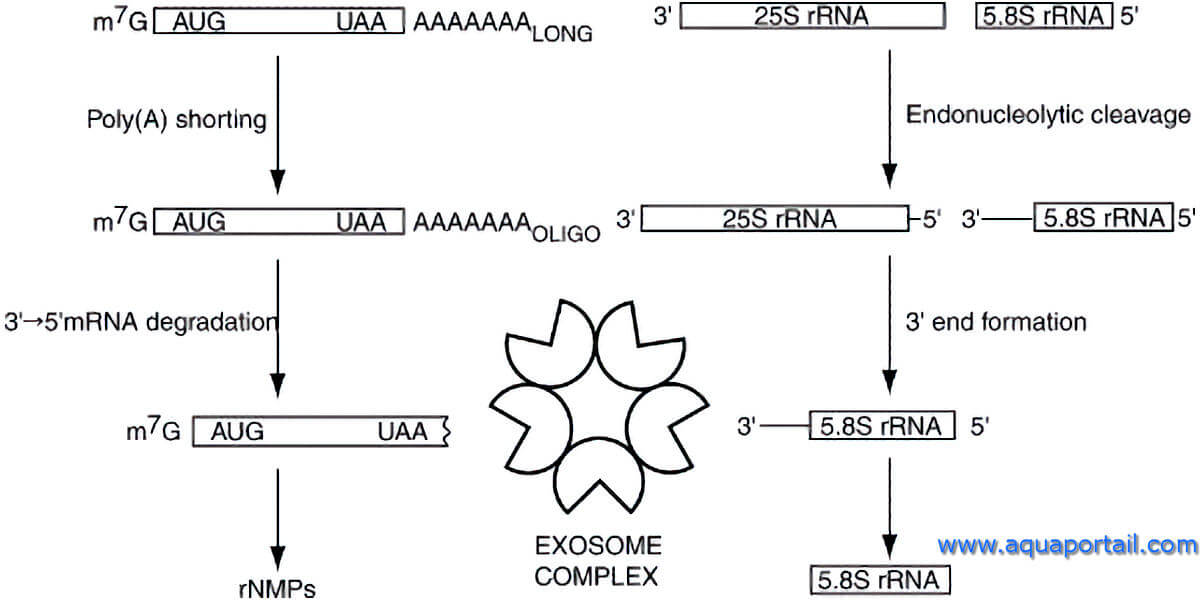
Rôles du complexe exosome dans la dégradation de l'ARNm et la maturation de l'ARNr 5.8S. Dans chaque processus, les exonucléases du complexe exosome hydrolysent l'ARN dans une direction 3′→5′.
Généralités
Les exoribonucléases 3′-5′ sont des médiateurs essentiels des voies de dégradation de l'ARN, et l'une de ces enzymes conservées au cours de l'évolution est la polynucléotide phosphorylase (PNPase). L'homologue humain de cette fascinante protéine enzymatique (phosphorylase polynucléotidique humaine, hPNPaseold-35) a été cloné il y a une dizaine d'années dans le contexte de la différenciation terminale et de la sénescence grâce à une nouvelle approche de "criblage de voies superposées".
Depuis lors, des informations importantes ont été recueillies sur cette exoribonucléase et son répertoire de fonctions en expansion. Des découvertes récentes concernent hPNPaseold-35 et elles continuent d'avoir un impact sur la compréhension de sa gamme croissante et diversifiée de fonctions.
Les exoribonucléases sont responsables du traitement et de la dégradation de l'ARN dans les cellules et affectent donc grandement le transcriptome des cellules. La méthode différentielle ARN-Seq permet une compréhension plus approfondie des rôles distincts des exoribonucléases. En utilisant cette méthode, il a été possible de déterminer que même si les trois exoribonucléases ont des modes d'action distincts, il existe un chevauchement fonctionnel bien que chacune affecte le métabolisme cellulaire de différentes manières. Dans ce chapitre, nous avons décrit une méthode d'analyse des données adaptée à l'étude des exoribonucléases. Cependant, il est possible d'adapter ce protocole à d'autres expériences différentielles d'ARN-Seq.
Épissage, transcription et dégradation de l'ARN
L'ARN peut subir un épissage, une transcription et une dégradation. La transcription produit une molécule d'ARN qui est une copie du gène entier, appelée transcrit primaire ou naissant. Ce transcrit naissant subit ensuite un épissage, dans lequel les régions non nécessaires à la fabrication de protéines (les régions introniques) sont supprimées tandis que les segments nécessaires à la production de protéines (les régions exoniques) sont conservés et rejoints.
L'épissage est un processus hautement régulé qui est effectué par un complexe protéique multimérique appelé splicéosome. Après l'épissage, la molécule d'ARNm est exportée du noyau et utilisée comme matrice pour la synthèse des protéines. De nombreux gènes produisent plus d'une forme d'ARNm (et donc de protéine) par un processus appelé épissage alternatif, dans lequel différentes combinaisons d'exons sont réunies.
Différentes protéines d'un même gène peuvent avoir des fonctions entièrement distinctes. Par exemple, dans les cellules C de la thyroïde, le gène de la calcitonine produit un ARNm codant pour la calcitonine, un inhibiteur des ostéoclastes, mais dans les neurones, le même gène produit un ARNm avec un complément d'exons différent via un épissage alternatif qui code pour un neurotransmetteur, le gène de la calcitonine. peptide apparenté.
Contrôle de la dégradation
Il semble y avoir un système actif de dégradation de l'ARN dans les cellules car elles transcrivent plus d'ARN qu'elles n'en accumulent. Les durées de vie utiles des espèces d'ARN varient : on s'attend à ce que l'ARN ribosomal ait une durée de vie plus longue que les introns excisés (dégradation nucléaire) ou les fragments d'espaceur, par exemple. La durée de vie des ARNm doit être étroitement régulée, bien que l'histoire de la manière dont cela intervient puisse ne pas être complète.
De toute évidence, les molécules d'ARN qui présentent des défauts dus à un traitement, un repliement ou un assemblage inapproprié avec des protéines sont rapidement identifiées par un système de surveillance et dégradées. La dégradation des ARNm est contrôlée, au moins en partie, par les microARN (miARN) et les petits ARN interférents (siARN). Ces petits ARN (21 à 26 nucléotides de longueur) peuvent déclencher le clivage de l'endonucléase ou éventuellement stimuler le taux de décapsulage de l'ARNm.
Chez l'homme, la seule endonucléase capable de clivage de l'ARNm est Ago2, une protéine argonaute. L'appariement de bases à l'extrémité 5′ du miARN (résidus 2 à 7) et l'ARNm attaqué établit l'interaction. L'appariement de bases entre les bases 10 et 11 du miARN et la cible d'ARNm établit le site de clivage. Les fragments d'ARNm générés par le clivage RISC sont dégradés par la machinerie générale de dégradation de l'ARNm. La majeure partie de la dégradation de l'ARNm existe à l'aide du complexe CCR4/Not, ciblé sur le 3′ UTR de l'ARNm et dirigé par des éléments et des protéines se liant à cette partie de l'ARNm. Un miARN peut fonctionner avec RISC et la protéine TTP (protéine de liaison à l'ARN spécifique à la séquence) pour cibler un ARNm contenant un ARE (élément riche en AU) pour la dégradation. Il s'agit de mécanismes complexes portant sur la dégradation des ARN et la répression de la traduction.
Dégradation par hydrolyse
La dégradation de l'ARN par hydrolyse du squelette phosphodiester est généralement considérée comme un obstacle à la cristallisation, en particulier dans l'étape de préparation de l'ARN où l'hydrolyse incontrôlée d'un ARN conduit à une hétérogénéité chimique. Cependant, l'hydrolyse contrôlée du squelette de l'ARN lors de la cristallisation, également connue sous le nom de "digestion en goutte", peut être avantageuse. Ceci est analogue à une dégradation protéolytique limitée dans plusieurs efforts de cristallisation qui conduisent à des cristaux d'un produit de dégradation.
L'hydrolyse de l'ARN est contrôlée en raison des contraintes stéréochimiques de la réaction d'hydrolyse : le groupe 2'-hydroxyle du sucre ribose attaque le groupe phosphate dans une configuration en ligne pour donner un clivage du squelette immédiatement en 3′ de l'hydroxyle attaquant. L'attaque n'intervient pas avec des bases dans une hélice de forme A ou une conformation restreinte (car l'hydroxyle doit être capable d'échantillonner la configuration en ligne) et intervient donc dans des régions qui sont en conformation flexibles. Les sites de clivage du squelette peuvent être facilement résolus par marquage P de l'ARN cristallisé et séparation des produits sur un gel de polyacrylamide dénaturant. L'isolement de chaque produit de clivage et le séquençage enzymatique révèlent l'emplacement de l'événement de clivage.
La gestion des sites de flexibilité conformationnelle telle que révélée par une hydrolyse limitée peut conduire à une meilleure diffraction des cristaux en raison d'un désordre réduit de la zone flexible ou de la création d'un site en conformation homogène d'un contact de réseau. Une approche populaire consiste à concevoir un système d'ARN en deux parties défini par la position de clivage. Les pièces séparées sont recuites après purification et soumises à des essais de cristallographie.
Des biologistes ont réalisé que le domaine P4–P6 contenait des clivages dus à la cristallisation et ont utilisé un système en deux parties comme moyen d'introduire des dérivés de nucléotides d'atomes lourds en synthétisant chimiquement un petit oligonucléotide avec de la 5-iodouridine. Le ribozyme glmS et le riboswitch FMN ont spécifiquement utilisé des systèmes en deux parties pour améliorer la cristallisabilité (cristallisation) et la résolution de diffraction. Dans le cas de FMN, la perte de la boucle coiffant l'hélice 4 a permis d'améliorer les contacts de réseau. Dans les deux cas, l'utilisation d'un système en deux pièces peut avoir soulagé la retenue du squelette et permis une conformation alternative qui était nécessaire pour établir un emballage cristallin plus serré.
Mesure des taux de dégradation de l'ARNm
Les méthodes pour mesurer les taux de dégradation de l'ARNm se divisent en deux catégories. La première approche implique des méthodes de chasse par impulsions dans lesquelles le pool cellulaire d'ARNm est marqué, par exemple, avec de l'uridine radioactive, puis le marqueur est chassé avec de l'uridine froide. Le renouvellement est mesuré comme le taux de diminution de la radioactivité dans l'ARNm du pool cellulaire. En principe, cette méthode présente l'avantage de ne pas perturber la croissance ou le métabolisme cellulaire. Néanmoins, il est généralement admis que les méthodes de chasse aux impulsions ne sont pas réalisables dans les cultures de bactéries puisque les durées de vie des ARNm sont comparables au temps nécessaire aux nucléosides externes pour s'équilibrer avec les pools cellulaires de nucléosides.
La deuxième approche, couramment utilisée, consiste à arrêter la transcription, puis à mesurer la dégradation de l'ARNm préexistant. Bien qu'il soit possible d'inactiver la transcription par la chaleur dans des souches mutantes d'E. coli exprimant une ARN polymérase thermolabile, la méthode la plus répandue utilise le médicament rifampicine pour arrêter la transcription. Les mesures classiques du renouvellement de l'ARNm avant l'ère de la génomique ont été effectuées à l'aide d'ARN en vrac radiomarqué ou de Northern Blot d'un transcrit spécifique. La dégradation a été traitée comme une réaction de premier ordre.
Les niveaux d'ARNm mesurés de 2 à 20 min après l'ajout de rifampicine ont été ajustés à une courbe de décroissance exponentielle. En général, des valeurs comprises entre 100 % et 20 % du niveau maximal ont été utilisées pour déterminer une demi-vie. L'avènement des études à l'échelle du génome a rendu nécessaire le développement de méthodes automatiques pour traiter de grands ensembles de données aboutissant à la détermination des taux de dégradation de milliers de messages.
En rapport avec "dégradation de l'ARN"

Une désoxyribonucléase est une enzyme qui catalyse l'hydrolyse des chaînes moléculaires d'acide désoxyribonucléique (ADN) en chaînes moléculaires plus courtes...
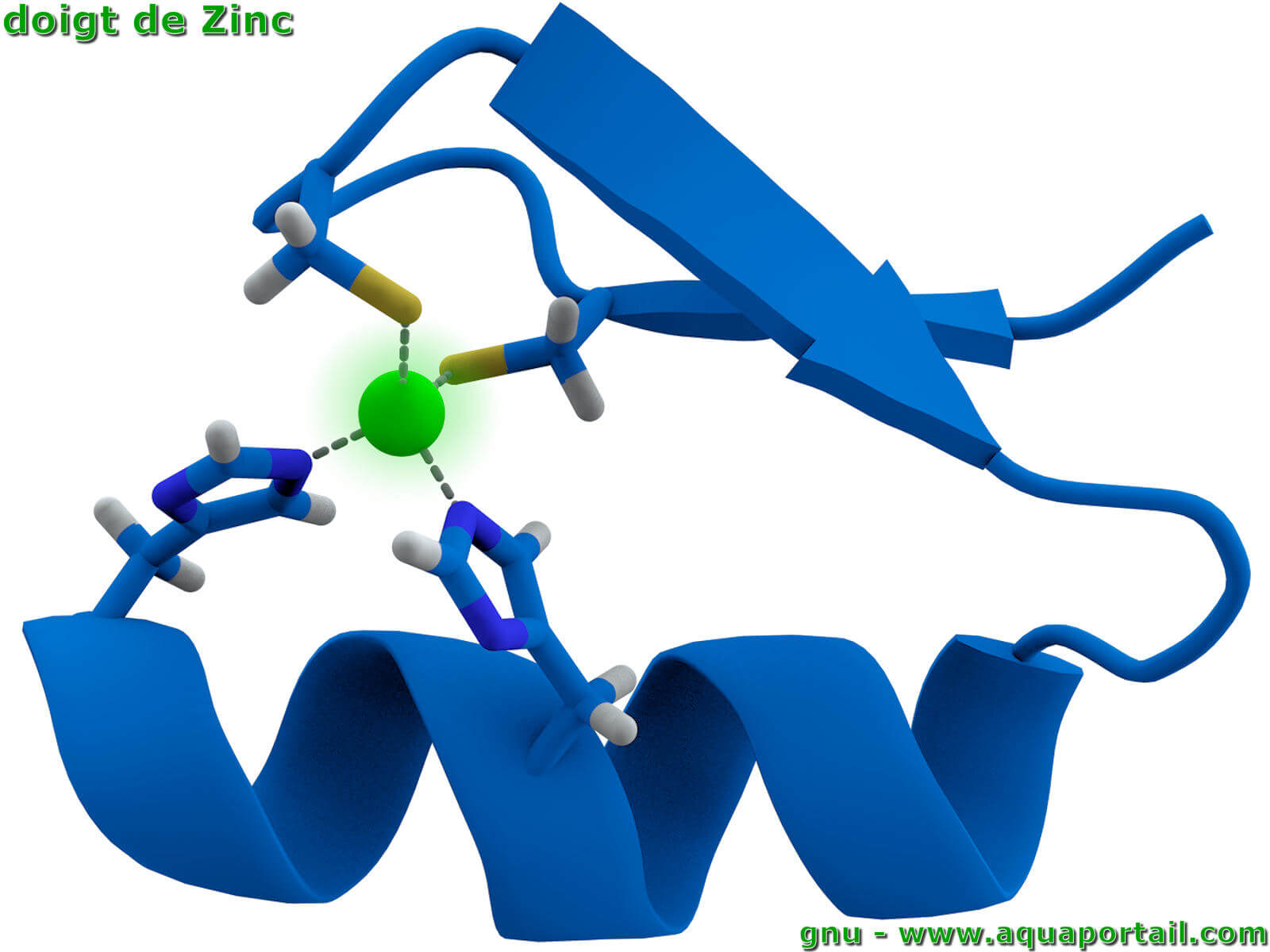
Un doigt de zinc, dans les protéines régulatrices de gènes, détermine le domaine protéique d'une trentaine d'acides aminés rassemblés autour d'un atome...
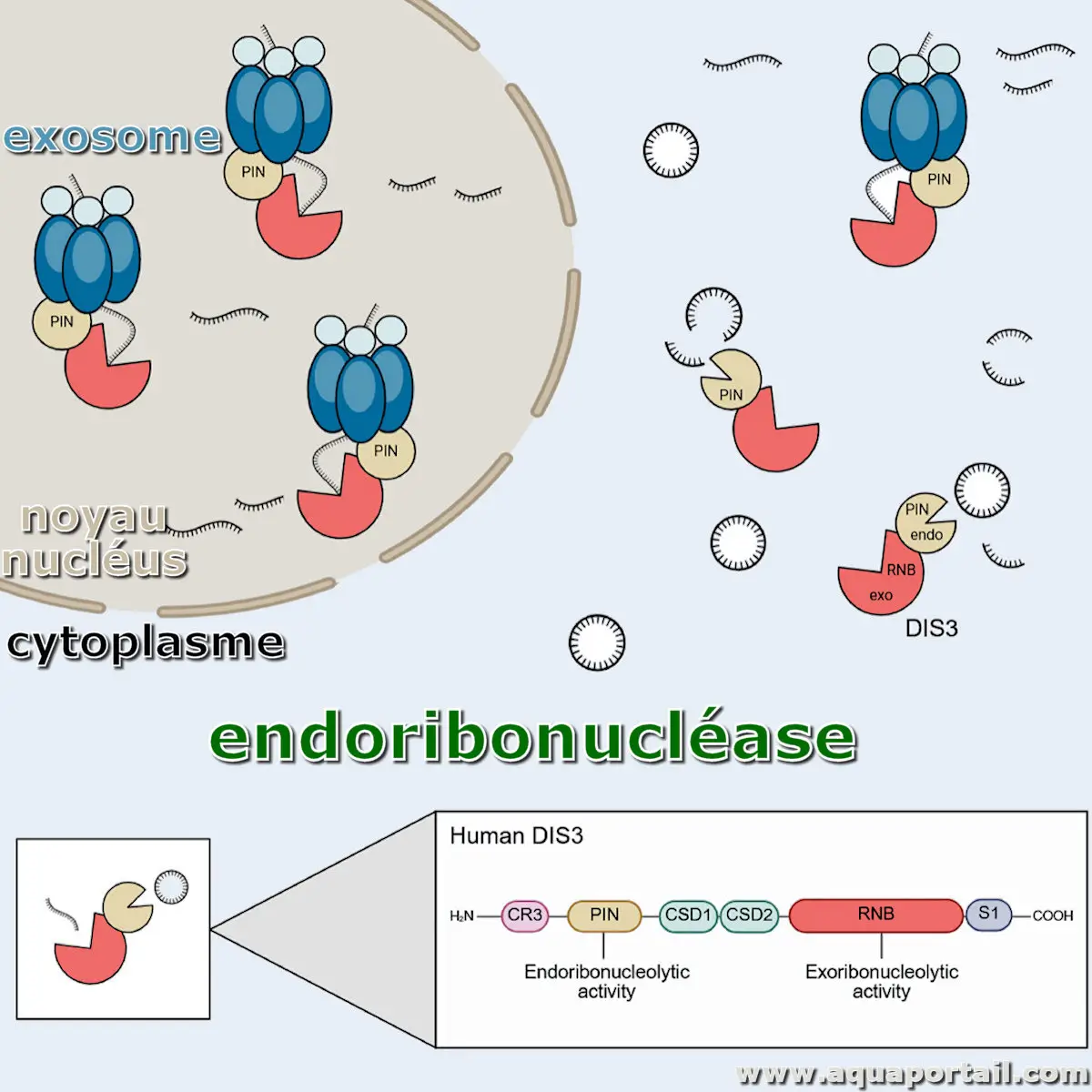
Une endoribonucléase est une enzyme appartenant au groupe plus large des ribonucléases, spécialisée dans le clivage de l'ARN.

Une ribonucléase, également appelée RNase, est une enzyme hydrolase qui catalyse la rupture des liaisons phosphodiester des ARN.
