Polymorphisme mononucléotidique
Définition
Le polymorphisme mononucléotidique est défini comme un locus avec des allèles qui diffèrent sur une seule base, l'allèle le plus rare ayant une fréquence d'au moins 1 % dans un ensemble aléatoire d'individus dans une population.
Le polymorphisme nucléotidique est une variation d'une seule paire de bases dans une séquence d'ADN. Il est une mutation d'une seule paire de bases à un locus spécifique dans un diploïde composé de deux allèles.
Des polymorphismes mononucléotidiques :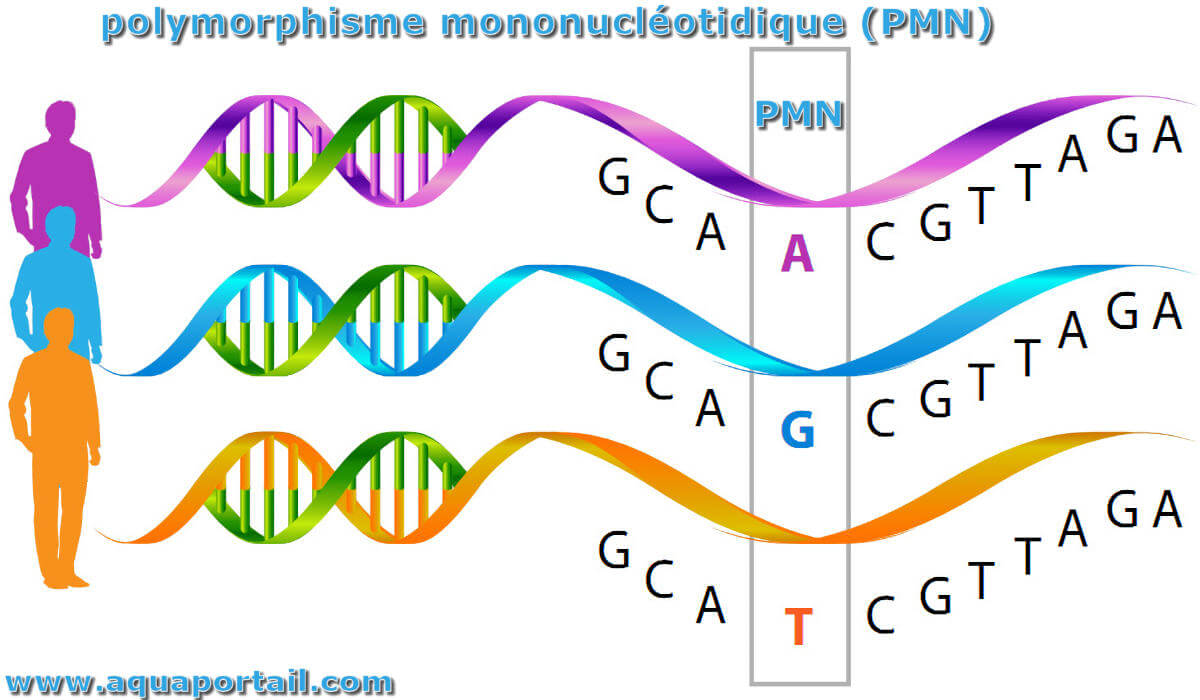
Les polymorphismes mononucléotidiques sont des mutations génétiques qui altèrent une seule base de l'ADN, provoquant une modification de la séquence des acides aminés et un dysfonctionnement d'une protéine correspondante.
Explications
Les polymorphismes mononucléotidiques sont des polymorphismes causés par des mutations ponctuelles qui donnent naissance à différents allèles contenant des bases alternatives à une position donnée du nucléotide dans un locus.
En raison de leur grande abondance dans le génome, les polymorphismes mononucléotidiques servent déjà de type de marqueur prédominant. De nombreuses techniques de séquençage et méthodes bioinformatiques sont appliquées pour le développement et l'identification de polymorphismes mononucléotidiques, telles que le séquençage aléatoire, le séquençage ciblé d'amplicons par PCR, le séquençage d'ARN et l'exploration bioinformatique des bases de données d'étiquettes de séquences exprimées (EST).
Applications du polymorphisme nucléotidique
Les technologies traditionnelles de génotypage des polymorphismes mononucléotidiques (SNP, de l'anglais single nucleotide polymorphism) conviennent à l'étude de quelques SNP dans le génome, ce qui aide à déterminer l'association entre les SNP et les traits économiques ou la maladie.
Les méthodes de génotypage de SNP à haut débit qui fournissent une haute densité de SNP sont utilisées dans la sélection génomique, la cartographie MAS et QTL. La sélection génomique permet de calculer une valeur d'élevage estimée (EBV) précise des animaux avant d'atteindre la maturité sexuelle, identifiant ainsi les animaux supérieurs à un âge plus précoce.
Les SNP sont également utilisés dans la cartographie des haplotypes, où des ensembles d'allèles ou de séquences d'ADN peuvent être regroupés afin qu'un seul SNP puisse identifier de nombreux SNP liés. Un tag SNP est un polymorphisme mononucléotidique représentatif dans une région du génome présentant un déséquilibre de liaison élevé.
Les SNP de balise sont utiles dans les études d'association de SNP sur le génome entier, dans lesquelles des centaines de milliers de SNP sur l'ensemble du génome sont génotypés. L'identification des SNP exploite de nouvelles méthodologies de sélection génomique chez les animaux de ferme.
La sélection génomique se traduit par un intervalle de génération réduit et une intensité de sélection accrue, ce qui permet de concevoir un programme de sélection rentable.
Rôle de biomarqueur
Les SNP sont des variants du génome présents naturellement dans la population humaine. Les SNP sont souvent prononcés comme des "snips". Chaque individu hérite d'une copie d'allèle de chaque parent, de sorte que le génotype individuel sur un site SNP est AA, BB ou AB. Le Human Genome Project, le SNP Consortium et d'autres groupes ont identifié environ 15 millions de variantes d'ADN communes, principalement des SNP.
Le polymorphisme mononucléotidique est défini comme un locus génomique où deux bases alternatives ou plus apparaissent avec une fréquence appréciable (> 1 %). Les SNP sont le type de variation le plus fréquent dans le génome humain, intervenant une fois toutes les plusieurs centaines de paires de bases dans tout le génome.
Pour tout SNP donné, le SNP peut intervenir dans une région codante mais ne pas entraîner de changement d'acide aminé, il peut intervenir dans une région codante avec un changement d'acide aminé, il peut intervenir dans une région régulatrice où le résultat est un changement de expression génique, ou il peut intervenir dans une région entre les gènes.
Marqueur de cancer
La possibilité de détecter rapidement des mutations génétiques dans des milieux physiologiques grâce à la biopsie liquide a attiré l'attention de la communauté des sciences des matériaux. Les propriétés physiques des nanoparticules combinées à des méthodes de transduction robustes garantissent une sensibilité et une spécificité améliorées d'un test donné et sa mise en oeuvre dans des dispositifs de point de service à usage courant.
L'art de la recherche sur l'utilisation des nanoparticules d'or dans le développement de biocapteurs colorimétriques permet la détection du polymorphisme mononucléotidique comme biomarqueur du cancer. Les principaux mécanismes demandent des tests assistés soit par des machines moléculaires à base d'ADN, soit par des réactions enzymatiques.
En biologie humaine
Les polymorphismes mononucléotidiques (SNP) sont des variations de paires de bases (pb) à des endroits spécifiques du génome. Par définition, une variation génétique à un seul locus pb n'est pas considérée comme un SNP à moins qu'au moins deux allèles aient des fréquences supérieures à 1 % dans une grande population d'individus non apparentés.
Le génome humain haploïde comprend environ 3 milliards de pb. Le nombre de SNP est estimé à 10–11 millions, ce qui donne une moyenne d'un SNP pour 275 pb. La grande majorité des SNP n'ont que deux allèles car le taux de mutation à une position pb particulière est extrêmement faible et il est très peu probable que deux mutations ponctuelles existent à la même position au fil du temps.
Pour cette raison, les SNP ont été largement utilisés pour cartographier l'histoire des populations en identifiant la distribution des allèles SNP parmi les populations existantes et passées. Les marqueurs SNP sont également le meilleur choix pour la construction d'un ensemble dense de marqueurs polymorphes qui peuvent être utilisés pour étudier l'association entre les marqueurs et un trait ou une maladie particulière.
Les SNP ont de nombreuses utilisations potentielles dans les enquêtes génétiques médico-légales, y compris l'estimation de l'ethnicité, des traits humains ou des maladies.
En génomique
La grande majorité des différences entre les individus sont des mutations ponctuelles dues aux SNP. Les SNP sont une classe très abondante de polymorphismes de séquences d'ADN que l'on trouve dans les génomes végétaux et animaux; ils sont plus répandus et ont une fréquence d'occurrence plus élevée que les microsatellites. De plus, les SNP sont génétiquement plus stables que les microsatellites.
Le principal avantage des SNP, cependant, est la possibilité de dépister la présence du polymorphisme sans électrophorèse sur gel en utilisant des méthodes à haut débit, telles que la technologie des microréseaux. Les marqueurs moléculaires d'ADN de première génération couramment utilisés (RFLP et RAPD) sont tous à base de gel.
Les SNP peuvent être utilisés aux mêmes fins que les marqueurs actuellement disponibles (RFLP, AFLP et RAPD), et comprennent : la construction de cartes de liaison, l'empreinte ADN, l'analyse de la diversité et la cartographie des traits.
Contrairement aux autres marqueurs à base d'ADN, les SNP sont directement basés sur des polymorphismes de séquence connus, et des quantités considérables de données de séquence sont nécessaires pour développer un marqueur SNP. C'est cette caractéristique des SNP qui les rend idéaux pour les marqueurs basés sur des gènes candidats.
Les SNP sont conservés au cours de l'évolution et sont donc utilisés dans l'analyse QTL et dans les études d'association à la place des microsatellites.
Les informations de séquence actuellement disponibles pour la vigne, les séquences BAC-end (BES), le séquençage shotgun, les EST (Expressed Sequence Tags, étiquette de séquence exprimée) et les deux séquences génomiques complétées, permettent d'identifier des millions de SNP électroniques (eSNP).
En 2007, des chercheurs ont identifié plus de deux millions de SNP dans la séquence hétérozygote du génome de la vigne ENTAV115 dont plus de 87,6 % (soit 1 751 176 SNP) pouvaient être cartographiés sur des chromosomes et au moins un SNP pouvait être détecté dans 86,7 % des gènes ancrés. En moyenne, la fréquence des SNP dans le génome était de 4 SNP/kb dans les 19 groupes de liaison. Ces eSNP sont une ressource précieuse pour le génotypage à haut débit.
Synonymes, antonymes
Voir tous les synonymes pour "polymorphisme mononucléotidique".3 synonymes (sens proche) de "polymorphisme mononucléotidique" :
- polymorphisme d'un seul nucléotide
- polymorphisme nucléotidique
- SNP
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à POLYMORPHISME MONONUCLÉOTIDIQUE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
L'expression POLYMORPHISME MONONUCLEOTIDIQUE est dans la page 7 des mots en P du lexique du dictionnaire.
Mots en P à proximité
polymorphie polymorphiquepolymorphisme polymorphisme de longueur des fragments polymorphisme génétique polymorphisme mononucléotidiquepolynucléaire polynucléé polynucléotide polyodonte polyol
En rapport avec "polymorphisme mononucléotidique"
Est nucléotidique tout composé se rapportant aux nucléotides, typiquement des molécules organiques de type acide nucléique comme l'ADN ou l'ARN.

En génétique, le polymorphisme est la présence simultanée de plusieurs morphes dans la population de génomes présentant des variations alléliques.
polymorphisme de longueur des fragments
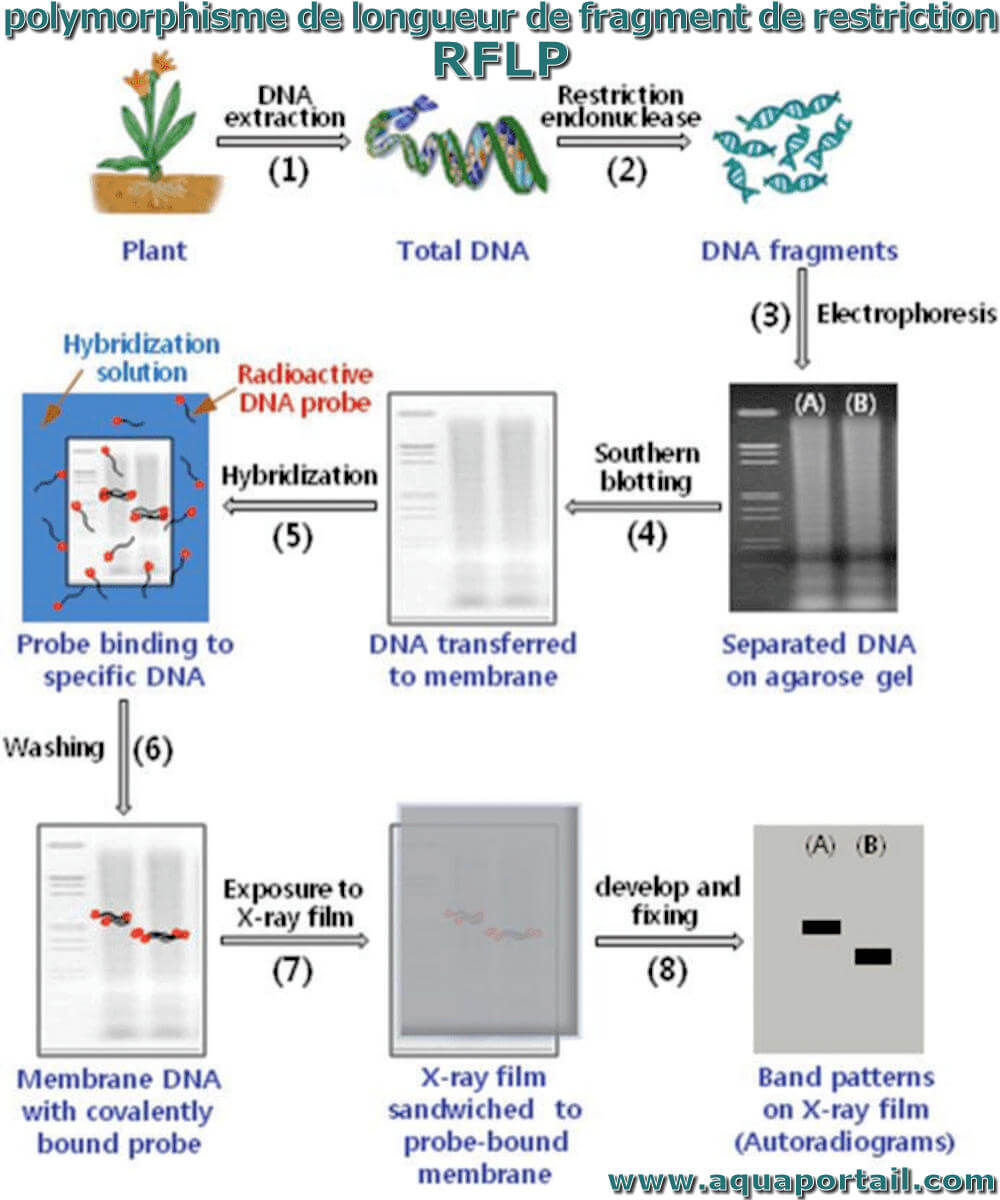
Le polymorphisme de longueur des fragments de restriction, ou RFLP, est une méthode pour déterminer l'empreinte génétique par marqueurs moléculaires.
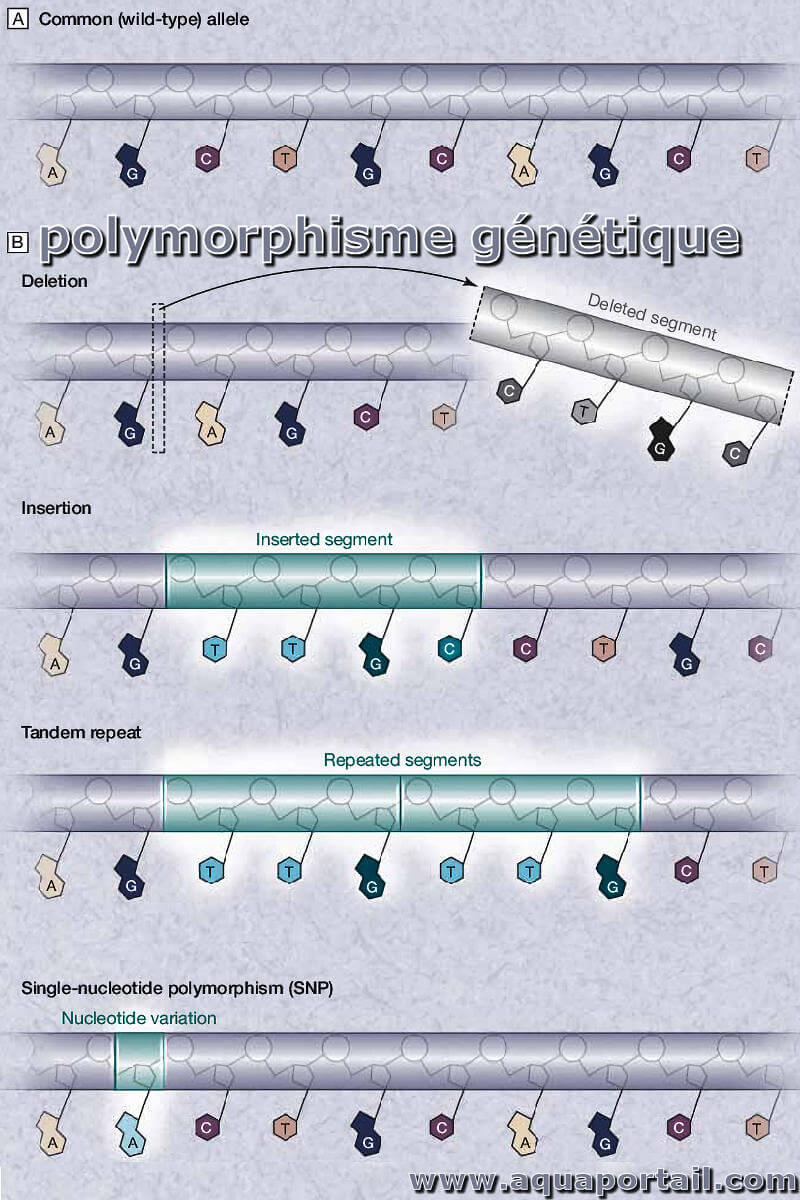
Le polymorphisme génétique, tel qu'il est lié à la génomique, fait référence à la présence de deux formes variantes ou plus d'une séquence génétique d'ADN...
