Mutation par décalage de cadre
Définition
Une mutation par décalage de cadre dans un gène fait référence à l'insertion ou à la suppression de bases nucléotidiques en nombres qui ne sont pas des multiples de trois. Dans une mutation par décalage du cadre de lecture (décalage ribosomique), un changement a lieu dans le positionnement relatif du ribosome.
Une mutation par décalage du cadre de lecture :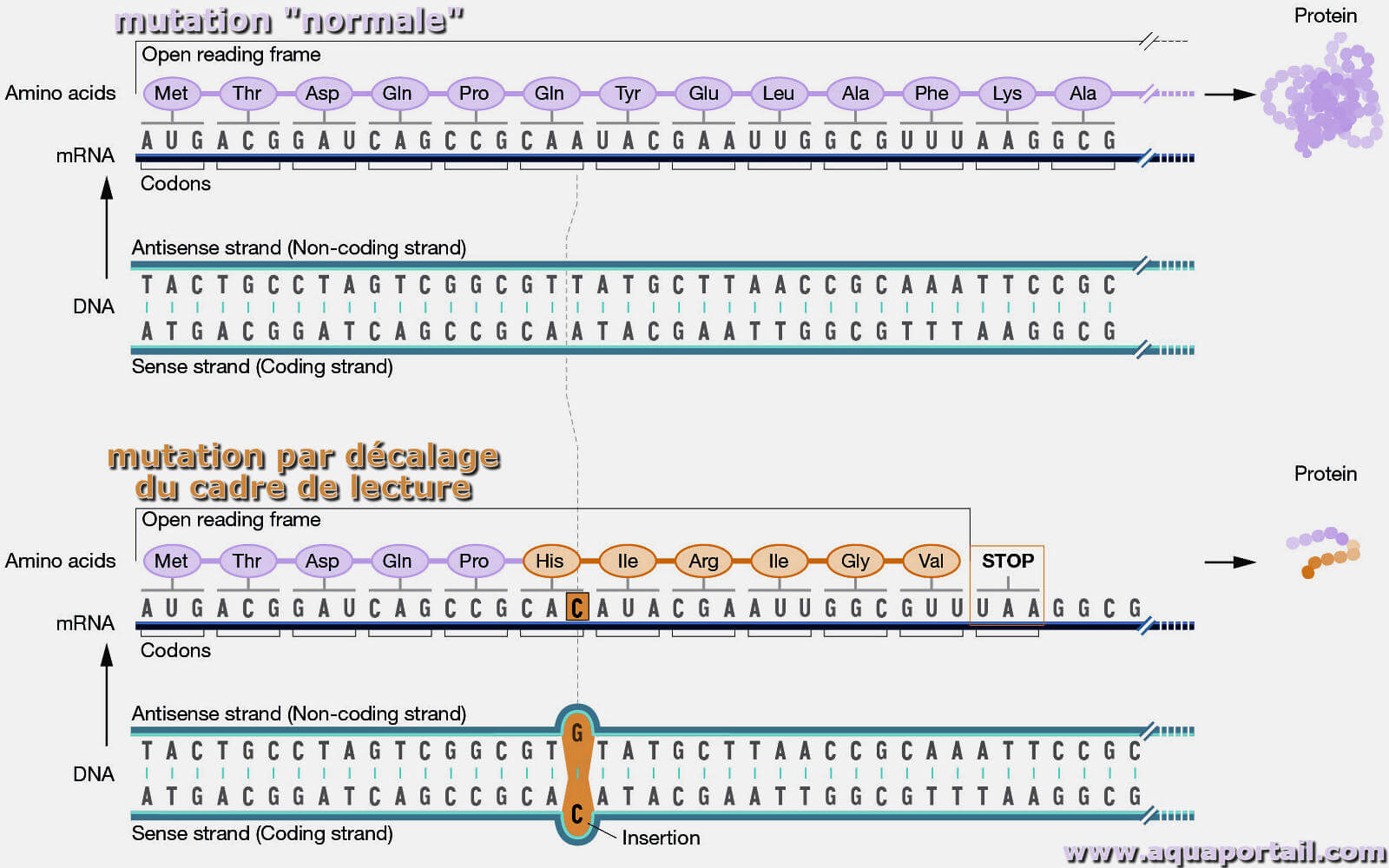
Dans une mutation de décalage de cadre pour l'ADN, un segment manquant peut amener la cellule à arrêter efficacement la lecture de ce gène. Les conséquences dépendent des informations stockées dans la partie du génome où le problème est intervenu. Si le gène remplit une fonction critique, un problème médical important peut en résulter.
Explications
Une mutation par décalage du cadre (provenant de la suppression ou de l'insertion) entraîne souvent une protéine d'une longueur différente de la protéine d'origine, avec une nouvelle section d'acides aminés apparemment aléatoires attachés à l'extrémité de la protéine qui n'ont rien à voir avec la séquence. d'acides aminés qui était là avant.
Les insertions, les suppressions et les mutations ponctuelles peuvent toutes générer une mutation de codon non-sens, arrêtant directement la traduction. Les mutations de terminaison de chaîne se traduisent dans la majorité des cas par un β-ARNm raccourci, souvent instable et rapidement dégradé.
La majorité de ces mutations qui interviennent dans les exons 1 et 2 entraînent le phénotype typique de βo-thalassémie hérité de façon récessive. En revanche, les mutations de décalage de cadre et mutations non-sens qui surviennent plus tard dans la séquence de la β-globine dans l'exon 3 produisent souvent un phénotype clinique plus sévère que le trait typique de la β-thalassémie et sont dites héréditaires de manière dominante.
Nombre de décalage
Les mutations de décalage de cadre sont des délétions ou des ajouts de 1, 2 ou 4 nucléotides qui modifient le cadre de lecture du ribosome et provoquent l'arrêt prématuré de la traduction au niveau d'un nouveau codon non-sens ou de terminaison de chaîne (TAA, TAG et TGA).
Une mutation par décalage de cadre dans un gène fait référence à l'insertion ou à la suppression de bases nucléotidiques en nombres qui ne sont pas des multiples de 3. Ceci est important car une cellule lit le code d'un gène par groupes de trois bases lors de la fabrication d'une protéine. Chacun de ces "codons triplets" correspond à l'un des 20 acides aminés différents utilisés pour construire une protéine.
Si une mutation perturbe ce cadre de lecture normal, la séquence entière du gène suivant la mutation sera lue de manière incorrecte. Cela peut entraîner l'ajout des mauvais acides aminés à la protéine et/ou la création d'un codon qui empêche la protéine de croître plus longtemps.
Production
Les mutations de décalage de cadre sont produites par des molécules qui peuvent s'insérer (s'intercaler) entre les bases normales pour créer des erreurs lors de la synthèse de l'ADN. Ce sont généralement des molécules plates, telles que les colorants d'acridine, qui ont une nature hydrophobe (rappelez-vous que l'empilement de bases hydrophobes est une force contributive dans la structure de l'hélice double-brin -bicaténaire-).
Une mutation de décalage de cadre est produite soit par insertion soit par suppression d'une ou plusieurs nouvelles bases. Étant donné que le cadre de lecture commence au site de départ, tout ARNm produit à partir d'une séquence d'ADN mutée sera lu hors cadre après le point d'insertion ou de suppression, produisant une protéine non-sens.
Comme pour une mutation ponctuelle, une mutation par décalage de cadre peut produire un codon de terminaison. De plus, les mutations de décalage de cadre, comme les mutations ponctuelles, sont moins délétères si elles sont proches du carboxyle terminal.
Évolution compensatoire intramoléculaire
Les mutations de décalage de cadre sont l'un des exemples de compensation intramoléculaire. Cependant, il existe de nombreux autres mécanismes qui favorisent l'évolution compensatoire intramoléculaire. L'une des preuves les plus claires de l'évolution compensatoire intramoléculaire a été décrite dans les structures tige-boucle des molécules d'ARN.
L'évolution compensatoire dans la structure intramoléculaire :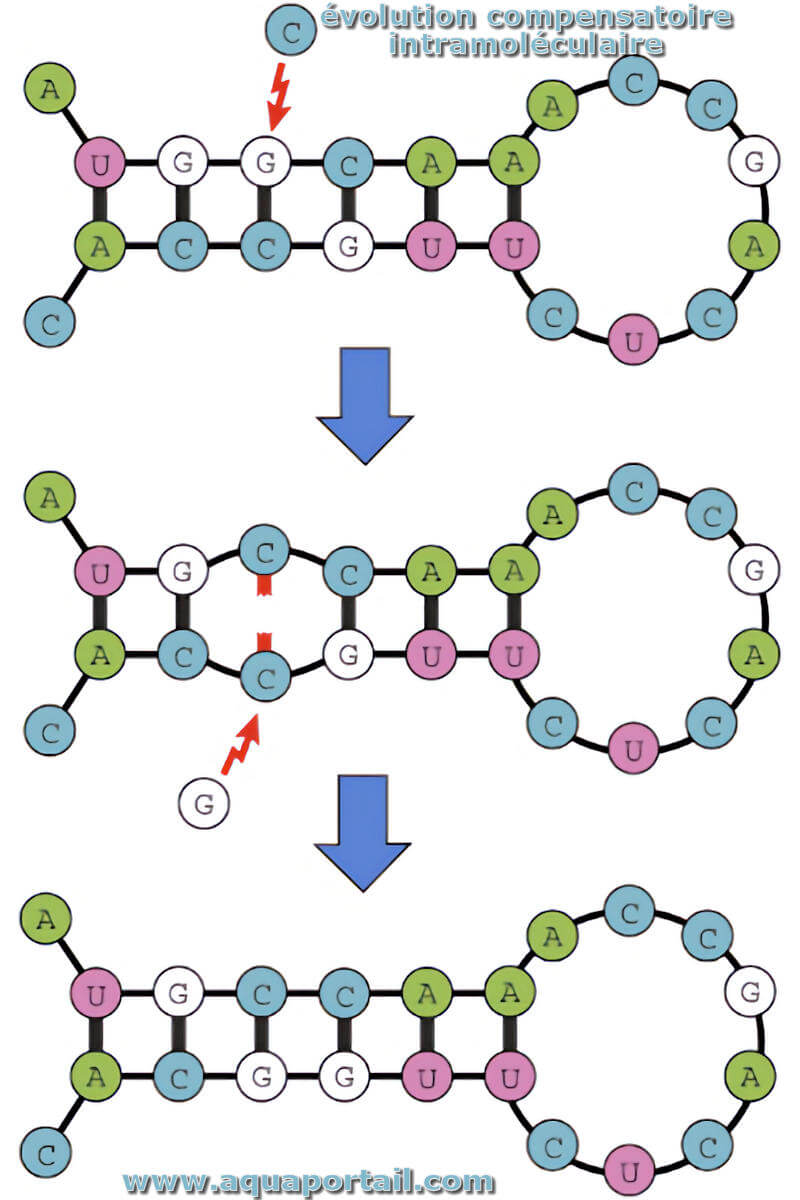
Exemple d'évolution compensatoire dans la structure tige-boucle de l'ARN. Les paires Watson et Crick sont liées entre elles par des liaisons hydrogène. Le changement de séquence d'ARN de G à C dans la région souche (qui est une mutation C à G dans le brin codant du génome) romprait le couplage, mais la liaison pourrait être restaurée par une mutation supplémentaire dans le brin opposé.
Comme ci-dessus, les molécules d'ARN transcrites forment souvent des paires Watson-Crick entre les bases A et U, et entre les bases C et G, dans une structure tige-boucle. Des mutations dans un brin écarteraient l'appariement et la structure secondaire des molécules d'ARN pourraient devenir instables ou passer à un état différent.
Cependant, si la base codant pour le brin opposé a une autre mutation qui pourrait former un appariement correct, les deux ribonucléotides pourraient former à nouveau un appariement correct. De nombreuses structures d'ARN telles que l'ARNt et la structure de l'ARN ribosomique ont le potentiel de favoriser l'évolution compensatoire, et des études ont montré que l'évolution compensatoire est un mode répandu d'évolution de la séquence d'ARN.
Les protéines sont repliées dans des structures tridimensionnelles complexes et de nombreux résidus d'acides aminés interagissent les uns avec les autres dans le processus de repliement, ce qui offre une énorme opportunité d'évolution compensatoire entre différents sites d'acides aminés.
Altération génétique et phénotype clinique
Des mutations homozygotes non-sens ou de décalage de cadre dans le gène NKX2.2 codant pour le facteur de transcription NKX2.2 provoquent un diabète sucré néonatal permanent.
À ce jour, trois patients de deux familles consanguines indépendantes ont été rapportés. Les trois patients présentaient des anomalies très importantes de la sécrétion d'insuline mais aucune altération de la fonction exocrine du pancréas, et deux d'entre eux présentaient également plusieurs manifestations extrapancréatiques dont un retard de développement modéré à sévère affectant les fonctions motrices et intellectuelles, une hypotonie, une déficience auditive bilatérale, une cécité corticale et une petite taille. entre autres
Instabilité des microsatellites
Les mutations de décalage de cadre dans les microsatellites peuvent être identifiées par extraction d'ADN à partir de tissus normaux et tumoraux (généralement des tissus inclus en paraffine), amplification de microsatellites sélectionnés par PCR et analyse de la taille des fragments par électrophorèse sur gel ou un séquenceur automatisé.
L'inclusion de paraffine est une technique standard utilisée dans les laboratoires cliniques et de recherche pour créer un bloc de tissu fixé au formol et inclus en paraffine (FFPE). Le tissu fixé au formol subit un traitement tissulaire puis est noyé dans de la paraffine (cire) pour créer un bloc FFPE ou un bloc de paraffine.
Des critères ont été élaborés pour normaliser la classification moléculaire de l'instabilité des microsatellites en utilisant soit un panel de microsatellites défini par consensus Bethesda, soit un panel révisé qui utilise davantage de marqueurs mononucléotidiques. La sensibilité du panel révisé des tests d'instabilité des microsatellites est d'au moins 90 %.
Seules les tumeurs occasionnelles de patients présentant des mutations pathogènes connues du gène de réparation des mésappariements sont des MSS. La spécificité du panel révisé est également très élevée. On pense que l'absence de mutations dans jusqu'à 20 % des familles HNPCC atteintes de tumeurs MSI-H est due à une incapacité à détecter des variants germinaux inhabituels.
La sensibilité et la spécificité du panel Bethesda original sont plus faibles en raison de l'échec des marqueurs dinucléotidiques, trinucléotidiques et tétranucléotidiques pour détecter les tumeurs déficientes en réparation des mésappariements, et de la mauvaise classification des tumeurs MSI-L en raison de résultats faussement positifs utilisant les mêmes marqueurs moléculaires.
Terminaison et surveillance de l'ARN
Des mutations ponctuelles ou des mutations de décalage de cadre peuvent introduire des codons d'arrêt prématurés dans une séquence d'ARNm, créant un ARNm non-sens. On s'attendrait à ce que des ARNm non-sens produisent des protéines tronquées, mais, en fait, de telles protéines tronquées sont rarement détectées dans la cellule. On pense que la cellule est protégée par un mécanisme de surveillance de l'ARNm connu sous le nom de désintégration médiée par l'ARNm non-sens (NMD), qui médie la dégradation rapide des ARNm non-sens.
Le mécanisme de ce processus est encore en cours d'élaboration. Cependant, on pense qu'il implique la liaison de la terminaison de la traduction à la dégradation de l'ARNm. La plupart des régions codantes d'ARNm contiennent plusieurs versions d'une courte séquence dégénérée connue sous le nom d'élément de séquence en aval (DSE). Ainsi, un codon d'arrêt non-sens, mais pas un codon d'arrêt légitime, devrait apparaître en conjonction avec une DSE. Lorsqu'un codon d'arrêt apparaît dans le contexte d'une DSE, des facteurs supplémentaires s'assemblent avec le complexe de terminaison.
Synonymes, antonymes
Voir tous les synonymes pour "mutation par décalage de cadre".0 synonyme (sens proche) pour "mutation par décalage de cadre".
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à MUTATION PAR DÉCALAGE DE CADRE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
L'expression MUTATION PAR DECALAGE DE CADRE est dans la page 5 des mots en M du lexique du dictionnaire.
Mots en M à proximité
mutarotation mutase mutation mutation faux-sens mutation non-sens mutation par décalage de cadremutation ponctuelle mutation spontanée mutationnisme mutique muton
En rapport avec "mutation par décalage de cadre"

En génétique, une mutation est une modification spontanée et aléatoire de la structure génétique, du gène ou du chromosome, qui aboutit à un effet détectable...

La mutation faux-sens est une altération génétique dans laquelle une substitution d'une seule paire de bases modifie le code génétique d'une manière...
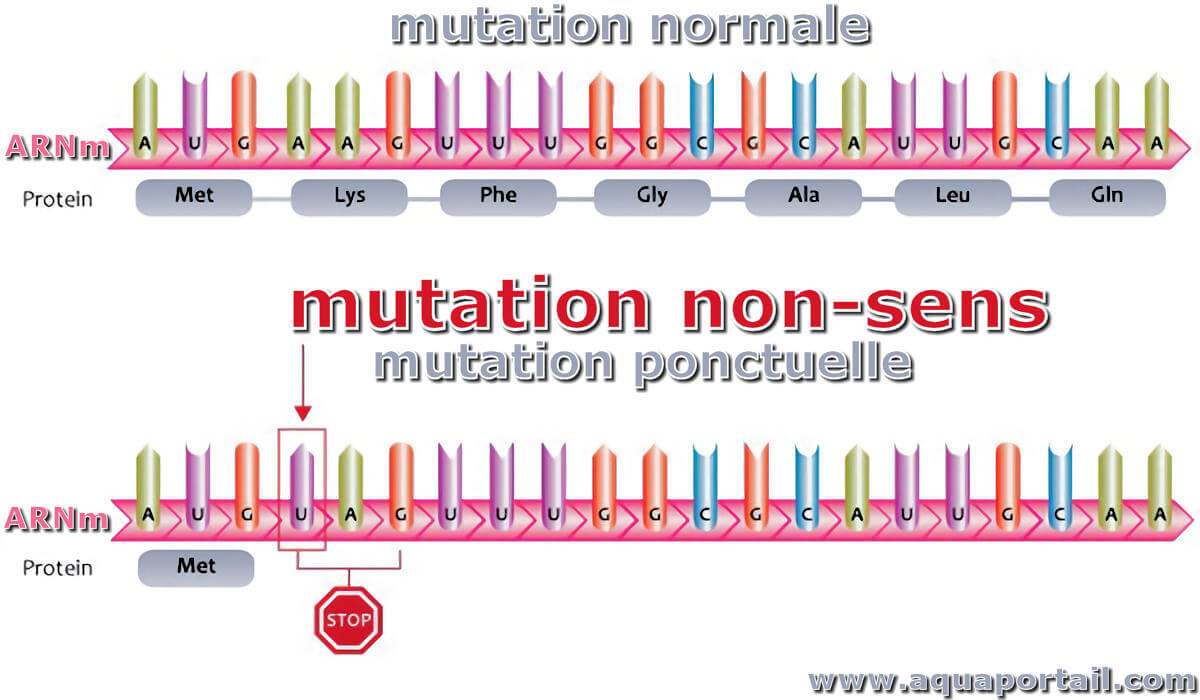
Une mutation non-sens est une mutation par substitution qui modifie le codon afin qu'il signale un arrêt précoce de la traduction.
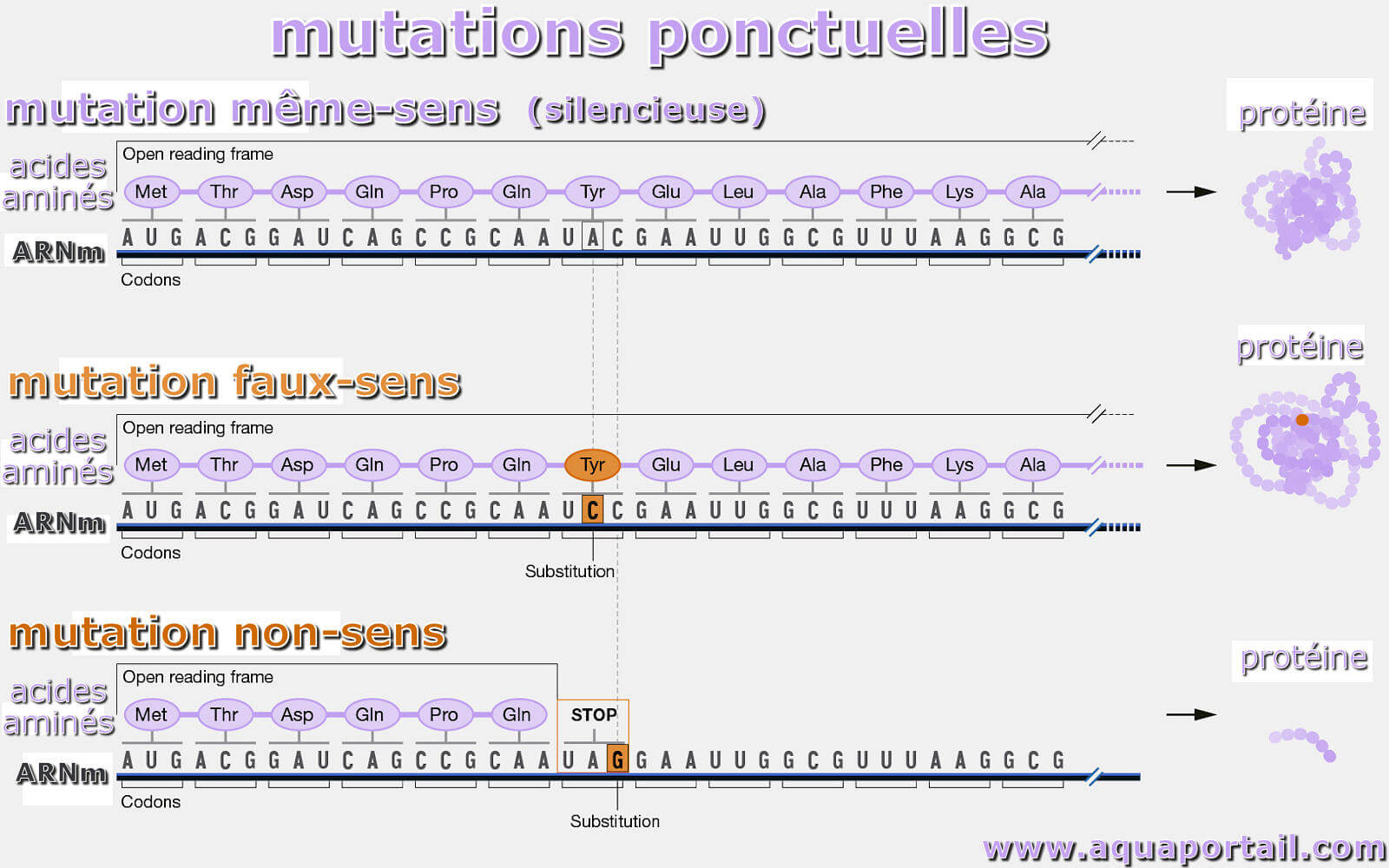
Une mutation ponctuelle est une mutation affectant seulement un ou très peu de nucléotides dans une séquence de gène.
