Ubiquitination
Définition
L'ubiquitination fait référence au processus dans lequel les molécules d'ubiquitine (une sorte de protéine de faible poids moléculaire) classent les protéines intracellulaires sous l'action d'une série d'enzymes spéciales, sélectionnent les molécules de protéines cibles et modifient spécifiquement la protéine cible. Ces enzymes spéciales comprennent les enzymes d'activation de l'ubiquitine, les enzymes de conjugaison de l'ubiquitine et les ubiquitine ligases, appelées E1, E2 et E3.
Processus d'ubiquitination des protéines :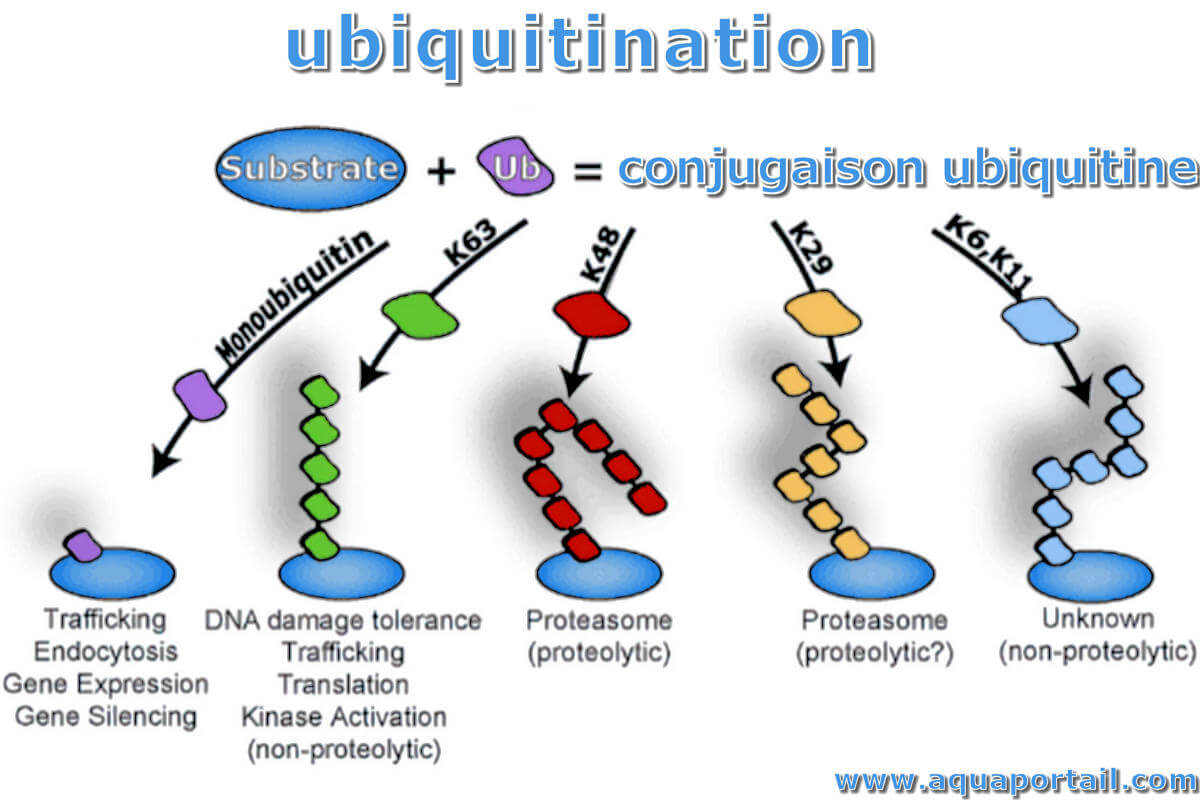
Au cours du processus d'ubiquitination, le devenir des protéines ubiquitinées dépend fortement de la longueur et du type de chaîne d'ubiquitine.
Explications
L'ubiquitination est une forme de modification post-traductionnelle dans laquelle l'ubiquitine-protéine est attachée à une protéine substrat. Il s'agit d'un processus en trois étapes impliquant trois enzymes : l'enzyme d'activation de l'ubiquitine (E1), l'enzyme de conjugaison de l'ubiquitine (E2) et l'ubiquitine-protéine ligase (E3).
Rôle
L'ubiquitination joue un rôle important dans la localisation, le métabolisme, la fonction, la régulation et la dégradation des protéines. Dans le même temps, il participe également au cycle cellulaire, à la prolifération, à l'apoptose, à la différenciation, aux métastases, à l'expression génique, à la régulation transcriptionnelle, à la transduction du signal, à la réparation des blessures, à l'inflammation et à la régulation immunitaire de presque toutes les activités de la vie.
Étapes
L'ubiquitination est étroitement liée aux tumeurs et aux maladies cardiovasculaires.Le processus comprend trois étapes principales :
- Activation : Au début de la cascade d'ubiquitination, les enzymes E1 se lient aux protéines ATP-Mg2+ et ubiquitine et catalysent l'adénylation C-terminale de l'ubiquitine. Dans l'étape suivante, la cystéine catalytique sur l'enzyme E1 attaque le complexe ubiquitine-AMP par substitution acyle, produisant des liaisons thioester et des groupes partants AMP. Enfin, l'ubiquitine E1 est transférée à l'enzyme E2 par transestérification du complexe protéique, où E2 catalyse la cystéine pour attaquer l'ubiquitine E1 à l'arrière du complexe protéique. Cependant, le processus de sulfatation de transport est complexe car les enzymes E1 et E2 forment des complexes intermédiaires, qui subissent tous deux une série de changements de conformation pour se lier.
- Conjugaison : L'enzyme activant l'ubiquitine active l'ubiquitine en attachant de manière covalente des molécules aux résidus de cystéine sur leurs sites actifs. La protéine activée a ensuite été transférée sur de la cystéine E2. Une fois liées à l'ubiquitine, les molécules E2 se lient à l'une des nombreuses ubiquitine ligases ou protéines E3 via une région de liaison structurellement conservée. La molécule E3 se lie au substrat de la protéine cible et transfère les résidus de lysine qui imprègnent la protéine de la cystéine E2 à la protéine cible. Les enzymes E2 sont caractérisées par leur structure hautement conservée, connue sous le nom de pli catalytique de conjugaison de l'ubiquitine (UBC).
- Ligature : l'ubiquitine ligase E3 catalyse l'étape finale de la cascade d'ubiquitination. Le plus souvent, ils produisent des liaisons isopeptidiques entre la lysine de la protéine cible et la glycine à l'extrémité C-terminale de la protéine. L'enzyme E3 agit comme un module de reconnaissance de substrat et interagit avec E2 et le substrat. Certaines enzymes E3 activent également les enzymes E2. Deux types principaux d'E3 sont définis par deux motifs de séquence distincts, le domaine RING et le domaine HECT (homologue à l'extrémité du groupe carboxyle E6-AP). Ces deux ligases médient le transfert d'Ub de différentes manières, car Ub sur E2 est transféré à Cys active dans le domaine HECT pour former des intermédiaires avant de se fixer au substrat, tandis que RING E3, qui manque de résidus Cys actifs, est utilisé comme pont pour faciliter le transfert direct de Ub de E2 au substrat.
Des cellules spécifiques ne contiennent généralement que quelques types de molécules E1, une plus grande diversité d'E2 et de très grands types d'E3. Par conséquent, la molécule E3 responsable de l'identification et de la liaison du substrat est le mécanisme de spécificité du substrat de la dégradation du protéasome. Chaque type d'E2 peut être associé à plusieurs E3.
Désubiquitination (déubiquitination)
La voie ubiquitine-protéasome est un important système de régulation de la dégradation des protéines dans les cellules. Grâce à l'ubiquitination des protéines substrats et à la dégradation du protéasome, diverses activités cellulaires peuvent être affectées ou régulées, notamment : la transcription des gènes, la régulation du cycle cellulaire, la réponse immunitaire, la fonction des récepteurs cellulaires, la croissance tumorale et les processus inflammatoires.
En raison de la fonction étendue et réversible de cette voie, elle est strictement régulée dans le corps, et la régulation de l'enzyme de désubiquitination est un lien important. À l'heure actuelle, il est confirmé qu'il existe de nombreuses enzymes de désubiquitination dans les cellules, qui sont principalement divisées en cinq types, qui sont principalement la famille des hydrolases carboxy-terminales de l'ubiquitine et la famille des enzymes de traitement spécifiques de l'ubiquitine.Ces différents types d'enzymes de désubiquitination sont capables d'hydrolyser les liaisons entre les chaînes d'ubiquitine sur les protéines substrats, de désubiquitiner et de réguler inversement la dégradation des protéines, affectant ainsi la fonction des protéines.
Enzymes de déubiquitination
Les enzymes de déubiquitination sont une grande famille de protéases. Il hydrolyse principalement les molécules d'ubiquitine à partir de protéines liées à l'ubiquitine ou de protéines précurseurs en hydrolysant des liaisons ester, des liaisons peptidiques ou des liaisons isopeptidiques à l'extrémité carboxy de l'ubiquitine. Diverses enzymes déubiquitinantes ont été isolées et identifiées à partir de levures et d'humains.
Sur la base de la similitude structurelle (c'est-à-dire, l'homologie de séquence d'acides aminés) de ces enzymes de désubiquitination et des mécanismes d'action possibles, les enzymes de désubiquitination actuelles peuvent principalement se diviser en cinq types, qui sont principalement la famille des hydrolases carboxy-terminales de l'ubiquitine et la famille des enzymes de traitement spécifiques à l'ubiquitine.
Synonymes, antonymes
Voir tous les synonymes pour "ubiquitination".2 synonymes (sens proche) de "ubiquitination" :
- ubiquitinylation
- ubiquitylation
1 antonyme (sens contraire) :
Les mots ou les expressions apparentés à UBIQUITINATION sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot UBIQUITINATION est dans la page 1 des mots en U du lexique du dictionnaire.
Mots en U à proximité
ubac ubidécarénone ubiquinone ubiquiste ubiquitaireubiquitinationubiquitine UFP UICN uliginaire uligineux
En rapport avec "ubiquitination"
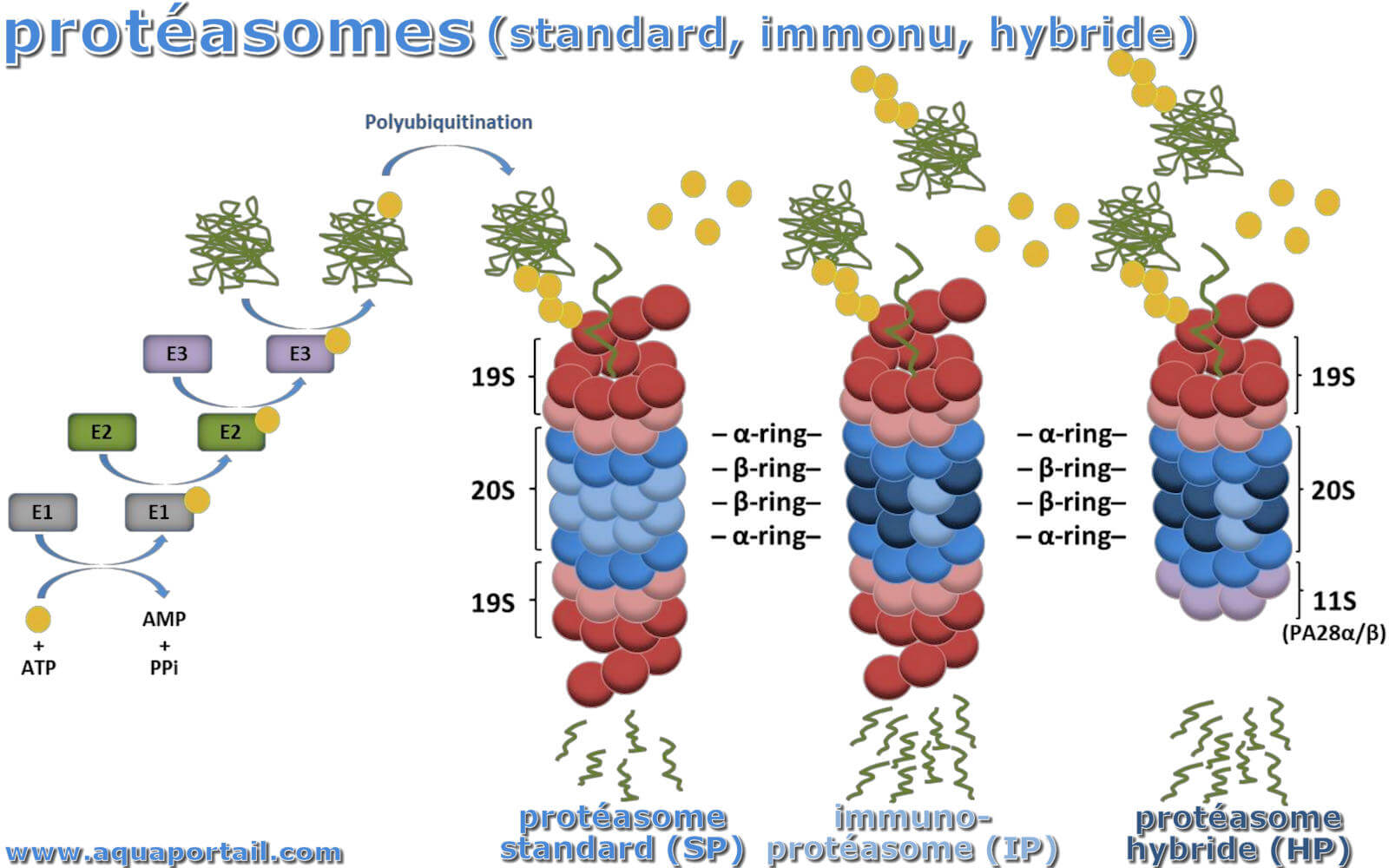
Le protéasome est un complexe de protéase hautement organisé situé dans le cytosol ainsi que dans le noyau des cellules eucaryotes.
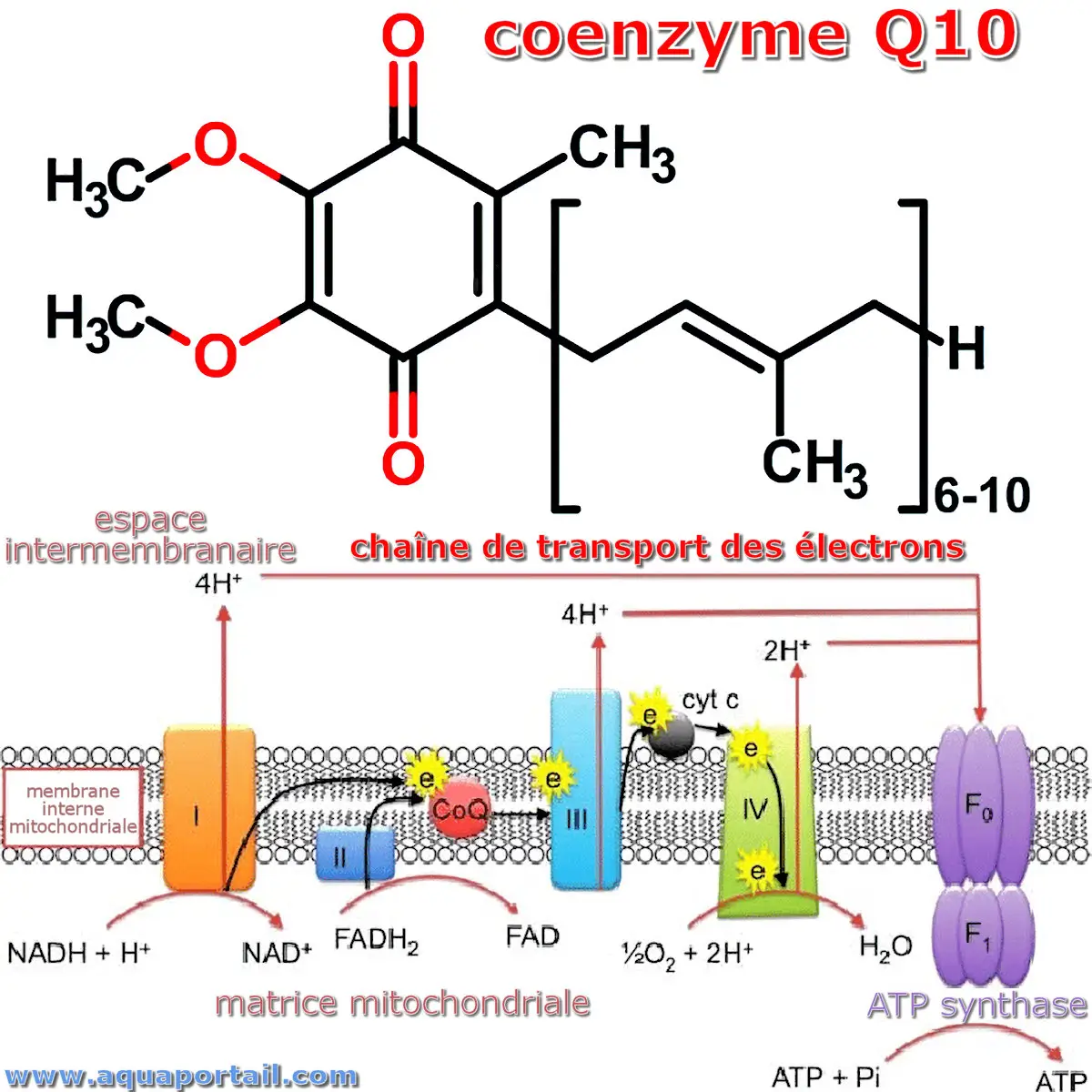
Une ubiquinone, ou coenzyme Q10, désigne une classe de transporteurs non protéiques d'une chaîne de transport des électrons.

Une personne ubiquitaire ou un être ubiquiste paraît exister dans plusieurs lieux à la fois, ou qui se trouve bien partout, qui est partout à la fois.
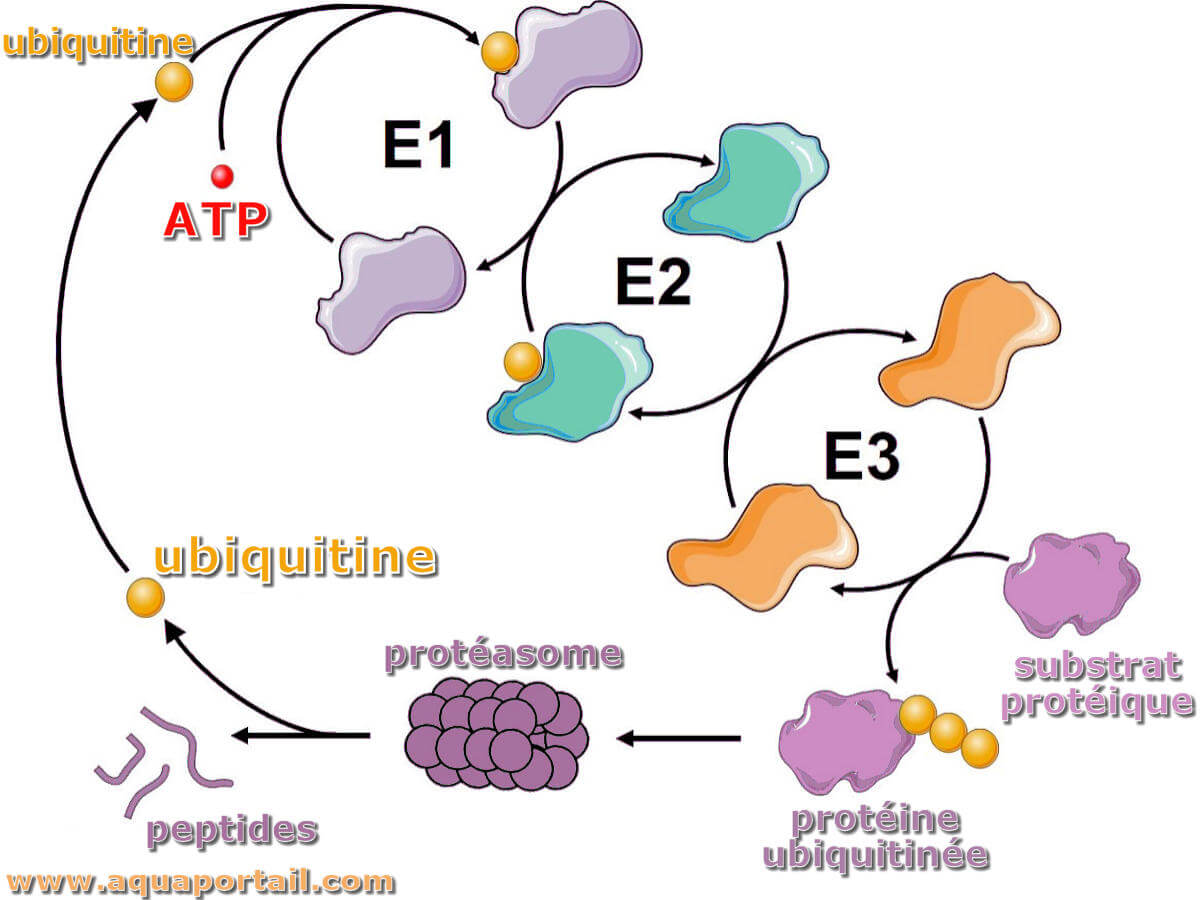
L'ubiquitine est une protéine de 76 acides aminés, thermostable, bien conservée au cours de l'évolution et jouant un rôle dans la dégradation des protéines.
