Protéasome
Définition
Le protéasome est un complexe de protéase hautement organisé situé dans le cytosol ainsi que dans le noyau des cellules eucaryotes. Il comprend une particule centrale catalytique 20S et deux particules régulatrices 19S, qui forment ensemble la structure 26S. Le protéasome 26S est responsable de la dégradation de la plupart des protéines ubiquitynées par un processus en plusieurs étapes impliquant la reconnaissance de la chaîne de polyubiquitine, le dépliement du substrat et la translocation du substrat dans le site actif de la cavité de la particule centrale.
Les protéasomes :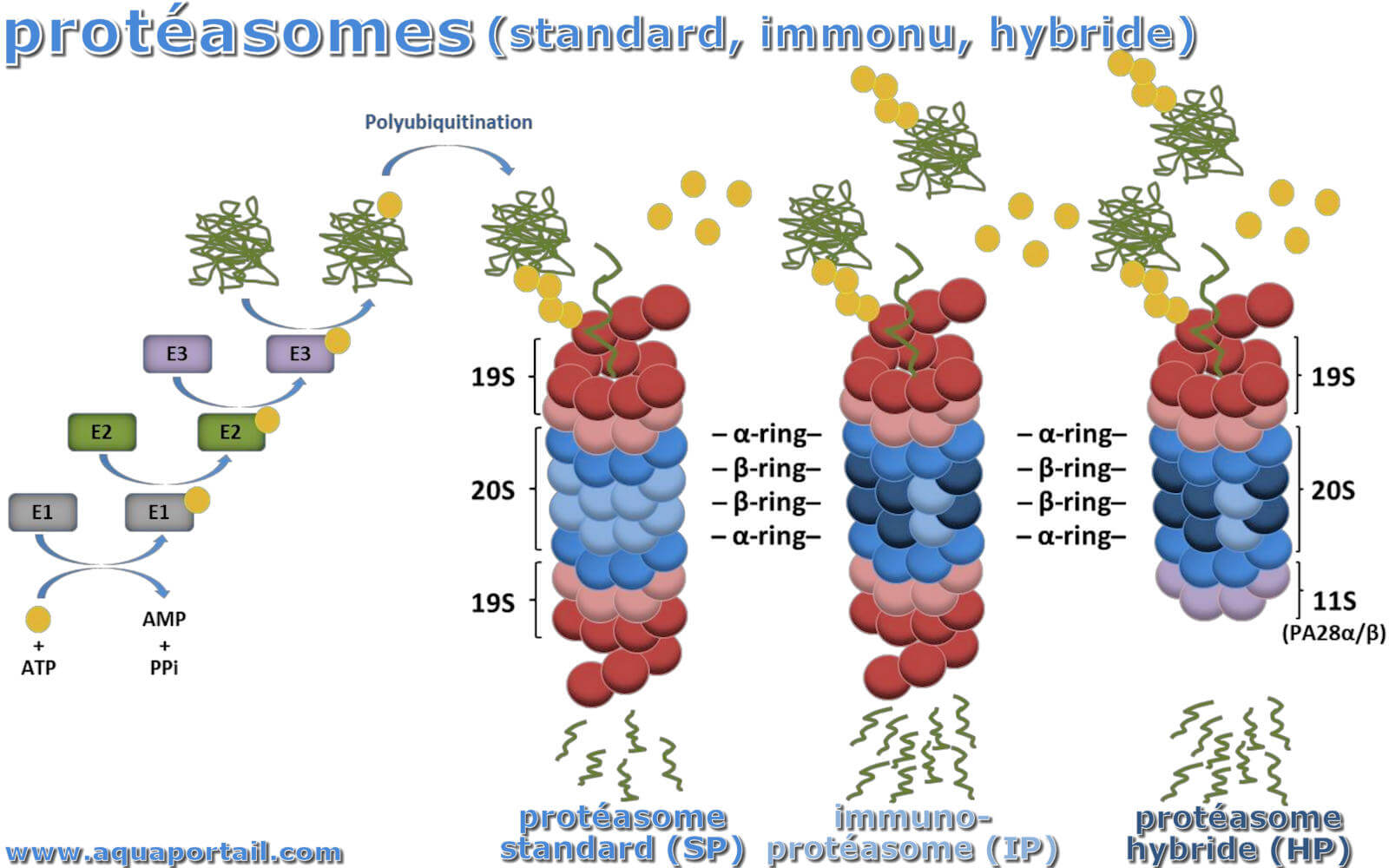
Voie de conjugaison de l'ubiquitine des substrats protéiques et leur dégradation ultérieure par des complexes de protéasome.
Explications
Dans la voie de conjugaison de l'ubiquitine des substrats protéiques et leur dégradation ultérieure par des complexes de protéasome, l'activation de l'ubiquitine nécessite son transfert vers une enzyme d'activation de l'ubiquitine E1 sous hydrolyse de l'ATP. Dans une deuxième étape, l'ubiquitine activée est transférée à une enzyme de conjugaison de l'ubiquitine E2 et utilisée pour la modification covalente des protéines intracellulaires avec le support des ubiquitine ligases E3.
Les substrats protéiques peuvent être marqués avec une seule molécule d'ubiquitine sur un site accepteur, ce qui entraîne une mono-ubiquitination. La molécule d'ubiquitine peut également être soumise à une ubiquitination, conduisant ainsi à la formation de diverses chaînes de polyubiquitine, parmi lesquelles les liaisons K48 représentent les signaux de ciblage de protéasome les plus répandus.
Le protéasome est récemment apparu comme une cible thérapeutique prometteuse pour le traitement du cancer. Par exemple, Velcade (bortézomibe), un inhibiteur du protéasome, a d'abord été approuvé pour le myélome multiple réfractaire et a été étendu au lymphome à cellules du manteau. De plus, les inhibiteurs du protéasome de nouvelle génération, tels que le carfizomibe et le salinosporamide A, ont déjà été développés et sont utilisés dans des essais cliniques. Ainsi, de nouvelles recherches approfondies se sont concentrées sur le protéasome dans les domaines fondamentaux et cliniques.
Complexes de protéasomes
Les principaux complexes de protéasomes comprennent les protéasomes standard (sp), les immunoprotéasomes (IP) et les protéasomes hybrides (HP) qui se distinguent par la nature de leurs sous-unités catalytiques et/ou régulateurs (19S, 11S). Il est entendu qu'à la fois IP et HP présentent une plus grande capacité de dégradation des protéines modifiées par l'ubiquitine que SP.
Protéasome 26S
Le protéasome 26S est composé d'au moins 33 sous-unités différentes et disposées en deux sous-complexes : une particule centrale protéolytique (CP; également connue sous le nom de protéasome 20S) et une ou deux particule(s) régulatrice(s) terminale(s) 19S (RP; également connue sous le nom de PA700; ne pas confondre avec le P680-P700 !).
Structure du protéasome 26S :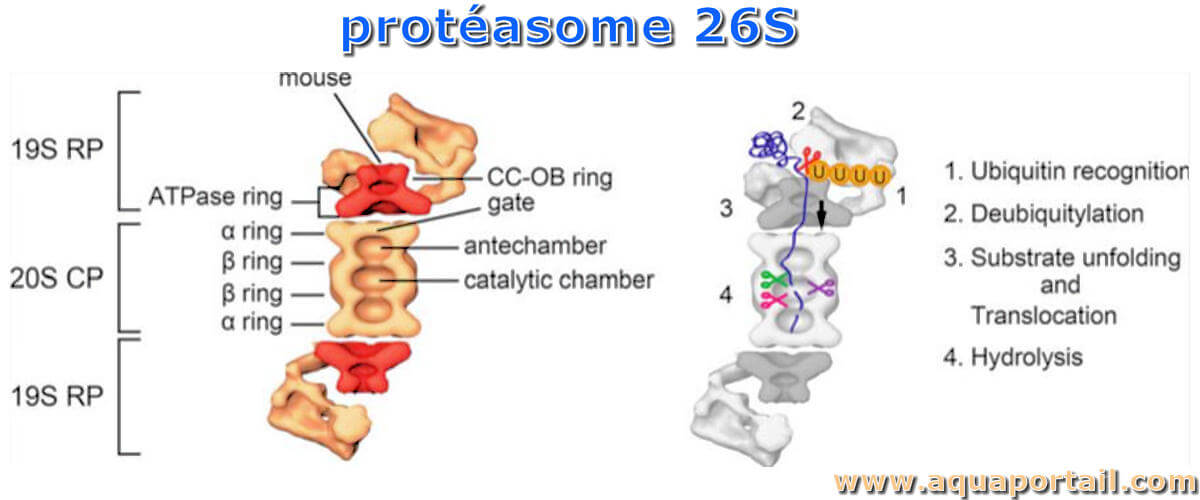
Le protéasome 26S se compose de la particule centrale catalytique 20S (CP) et de la particule régulatrice 19S (RP).
Le CP est formé de quatre anneaux empilés : deux anneaux α extérieurs et deux anneaux β intérieurs. Les sous-unités 19S ATPase forment également une structure à double anneau, appelée anneau CC-OB et anneau ATPase. Les chaînes de polyubiquitine avec quatre protéines d'ubiquitine ou plus servent de signal de ciblage pour le protéasome 26S. La protéine substrat est dépliée, transloquée dans la CP et dégradée en oligopeptides. Parallèlement, les chaînes de polyubiquitine sont désassemblées par des sous-unités enzymatiques désubiquitynantes.
Le CP est une structure en forme de tonneau d'environ 730 kDa constituée de quatre anneaux heptamériques, tandis que le 19S RP est un complexe d'environ 930 kDa constitué de 19 sous-unités différentes. La RP 19S se lie à une ou aux deux extrémités de la CP latente pour former un protéasome enzymatiquement actif.
Le CP 20S (alias protéasome 20S) est un complexe protéique bien organisé avec un coefficient de sédimentation de 20S et une masse moléculaire d'environ 730 kDa. Le CP dégrade progressivement les protéines substrats, générant des oligopeptides dont la longueur varie de 3 à 15 résidus d'acides aminés. Les produits peptidiques résultants sont ensuite hydrolyses en acides aminés par des oligopeptidases et/ou des amino-carboxyle peptidases. Chez les eucaryotes supérieurs, les oligopeptides générés par le protéasome peuvent être utilisés par les molécules du complexe majeur d'histocompatibilité (CMH) de classe I pour la présentation d'antigènes intracellulaires/endogènes au système immunitaire.
Le RP régule la dégradation du substrat en se liant aux substrats polyubiquitinés, en éliminant les chaînes de polyubiquitine, en dépliant les protéines du substrat, en ouvrant la porte du CP et en transférant les substrats dépliés dans le CP où se trouvent les sites catalytiques. Le RP 19S comprend au moins 19 sous-unités intégrales différentes avec des masses moléculaires allant de 10 à 110 kDa qui peuvent être sous-classées en deux groupes, particule régulatrice des sous-unités triple-ATPase (Rpt1–6) et particule régulatrice des sous-unités non-ATPase (Rpn1–15). Bien que deux sous-unités Rpn, Rpn4 et Rpn14, aient été considérées à tort comme des sous-unités intégrales, les deux protéines se sont avérées être respectivement des facteurs de transcription et des facteurs d'assemblage du protéasome. Le RP peut être divisé en deux sous-complexes, le couvercle et la base; la base comprend six sous-unités AAA+ (ATPases associées à diverses activités cellulaires) différentes d'ATPase (Rpt1-Rpt6) et trois sous-unités non-ATPase (Rpn1, Rpn2 et Rpn13), tandis que le couvercle comprend neuf sous-unités non-ATPase (Rpn3, 5–9, 11, 12 et 15). La connexion entre le couvercle et la base est stabilisée par le sous-ensemble Rpn10.
Assemblage du protéasome
Ces dernières années, plusieurs groupes se sont intéressés aux mécanismes impliqués dans l'organisation des structures complexes des protéasomes 26S. Pour assurer une dégradation rapide et complète du substrat, le protéasome 26S lui-même doit être formé correctement et rapidement dans la structure sophistiquée à partir de plus de 66 sous-unités. À cet égard, le protéasome 26S a trois anneaux, l'anneau CP, l'anneau CP β et l'anneau 19S ATPase, chacun étant formé correctement à partir de six ou sept sous-unités structurellement apparentées mais distinctes.
On sait maintenant qu'une série de chaperons dédiés au protéasome sont impliqués dans l'assemblage efficace et correct de la base CP et 19S, respectivement. Fait intéressant, les deux assemblages sont des processus à plusieurs étapes initiés par la formation de sous-assemblages spécifiques comme décrit.
Bien que les mécanismes sous-jacents à l'assemblage CP soient bien établis, la façon dont l'assemblage RP procède est quelque peu controversée. Néanmoins, l'étude de l'assemblage du protéasome est fondamentalement importante et pourrait servir de base à la conception et au développement de nouveaux médicaments anticancéreux qui ciblent la biogenèse du protéasome.
Système ubiquitine-protéasome (UPS)
Le système ubiquitine-protéasome (UPS) est le principal système de dégradation des protéines intracellulaires et non lysosomales. Grâce à sa capacité unique à éliminer les protéines anciennes, endommagées, mal repliées et/ou régulatrices de manière très spécifique, l'UPS est virtuellement impliquée dans presque tous les aspects de la vie eucaryote. L'importance critique de l'UPS est particulièrement visible dans les cellules immunitaires qui subissent un remodelage fonctionnel rapide et profond lors de la reconnaissance d'un agent pathogène.
L'activation immunitaire innée et/ou adaptative est en effet caractérisée par un certain nombre de changements substantiels ayant un impact sur divers processus cellulaires, notamment l'homéostasie des protéines, la transduction du signal, la prolifération cellulaire et le traitement des antigènes, qui sont tous étroitement régulés par l'UPS. Dans cette revue, nous résumons et discutons des progrès récents dans notre compréhension des mécanismes moléculaires par lesquels l'UPS contribue à la génération d'une réponse immunitaire adéquate. À cet égard, nous discutons également des conséquences du dysfonctionnement de l'UPS et de son rôle dans la pathogenèse des troubles immunitaires récemment décrits, notamment le cancer et les maladies auto-inflammatoires.
Dans les cellules eucaryotes, le système ubiquitine-protéasome (UPS) contrôle presque tous les processus cellulaires de base - tels que la progression dans le cycle cellulaire, la transduction du signal, la mort cellulaire, les réponses immunitaires, le métabolisme, le contrôle de la qualité des protéines et le développement - en dégradant les cellules à courte durée de vie. protéines régulatrices ou structurellement aberrantes. Les protéines destinées à la dégradation sont modifiées par un petit marqueur de dégradation appelé ubiquitine (Ub) modifié par une série de synergies d'ubiquitination. Des cycles répétés de conjugaison d'ubiquitine conduisent à la formation d'une chaîne de polyubiquitine sur la protéine cible. Les chaînes de polyubiquitine avec quatre ubiquitine ou plus sont ciblées par le protéasome 26S et la protéine cible est dégradée en un oligopeptide. Concomitamment, les chaînes de polyubiquitine sont désassemblées par des enzymes de déubiquitination,qui clivent les molécules d'ubiquitine du brin pour permettre la recirculation de l'ubiquitine.
Synonymes, antonymes
Voir tous les synonymes pour "protéasome".0 synonyme (sens proche) pour "protéasome".
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à PROTÉASOME sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot PROTEASOME est dans la page 9 des mots en P du lexique du dictionnaire.
Mots en P à proximité
protactile protamine protandreprotandrie protéase protéasomeprotection de l'environnement protection des berges protée protéiforme protéinase
En rapport avec "protéasome"

La protéolyse est la dégradation des protéines soit par des enzymes spécifiques, appelées peptidases, soit par dégradation intracellulaire.
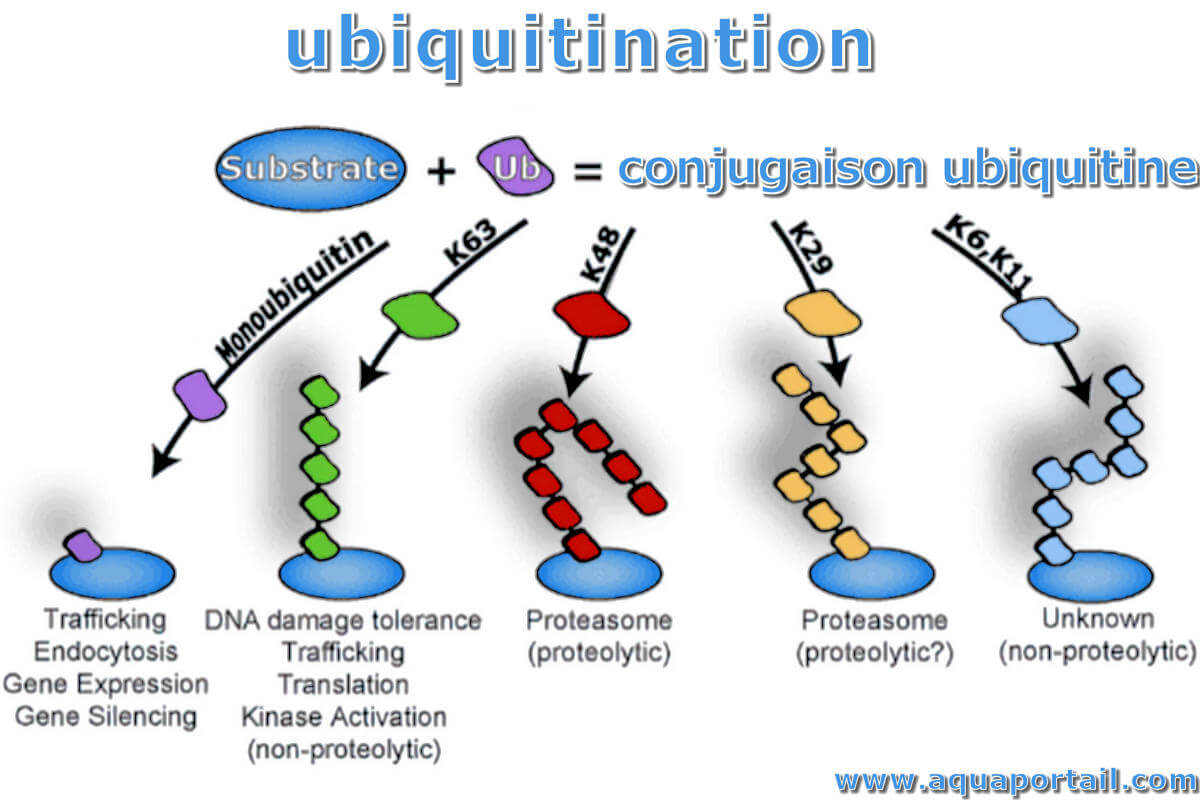
L'ubiquitination fait référence au processus dans lequel les molécules d'ubiquitine (une sorte de protéine de faible poids moléculaire) classent les protéines...
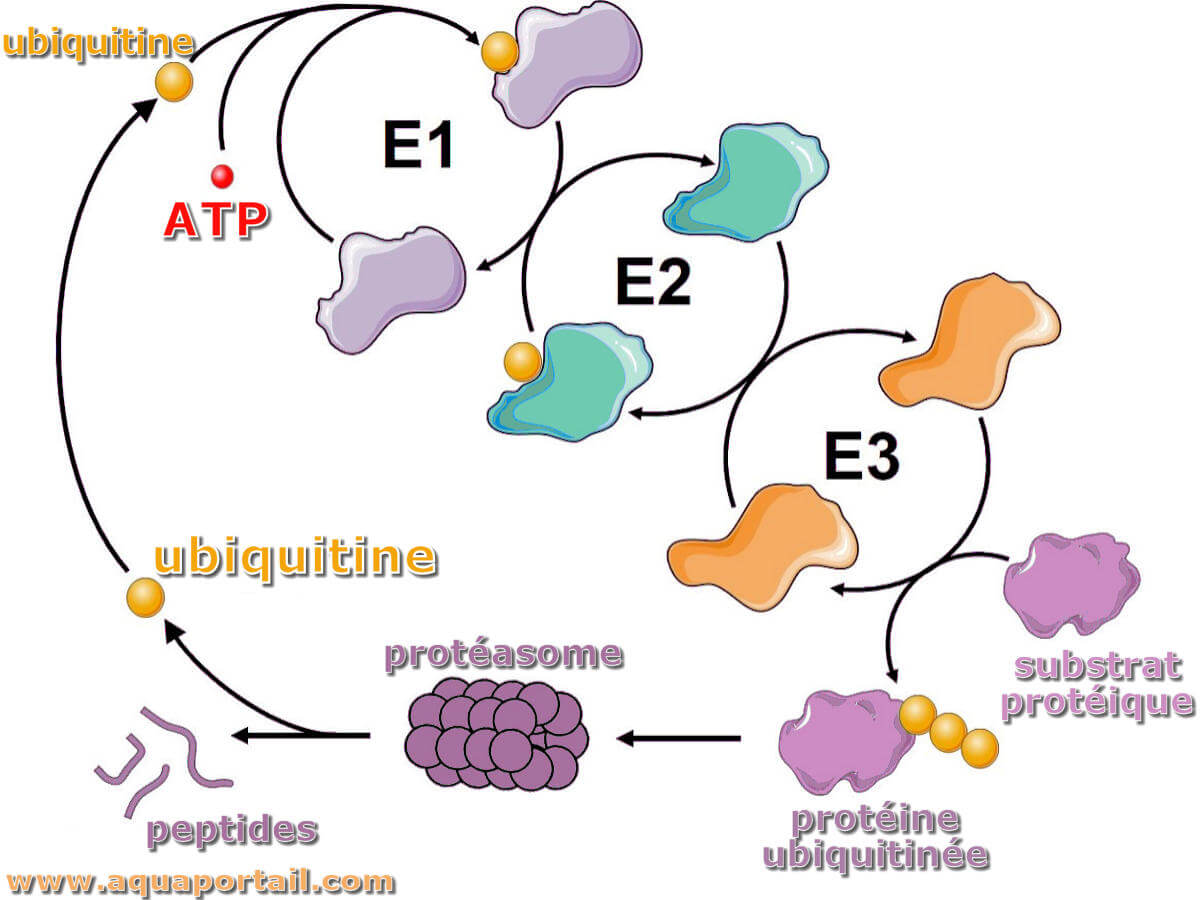
L'ubiquitine est une protéine de 76 acides aminés, thermostable, bien conservée au cours de l'évolution et jouant un rôle dans la dégradation des protéines.
Une protéase est une peptidase, une enzyme protéolytique qui coupe une liaison peptidique au sein d'une séquence protéique, mais pas aux extrémités.
