Aptamère
Définition
Un aptamère est un acide nucléique simple brin (ADN et ARN) isolé à partir de banques d'oligonucléotides par sélection in vitro, par enrichissement exponentiel en présence du ligand (méthode SELEX), appelés anticorps chimiques.
Les aptamères sont des acides nucléiques ayant la propriété de se lier à une molécule ou à une protéine. Les premiers aptamères ont été isolés au début des années 90 dans les laboratoires de Jack Szostak et Larry Gold.
L'aptamère d'ARN de la biotine :
La structure d'un aptamère d'ARN spécifique de la biotine montre la surface et le squelette de l'aptamère en jaune. La biotine (sphères) s'insère parfaitement dans une cavité de la surface de l'ARN.
Les aptamères qui sont des oligonucléotides (puisque d'autres aptamères peuvent être des molécules peptidiques) sont créés généralement à partir d'un grand pool de séquences aléatoires et utilisés principalement pour la recherche et les applications cliniques (par exemple en tant que médicaments macromoléculaires). Les aptamères sont capables de se lier à des sites cibles spécifiques par le biais d'interactions non covalentes. Ils sont utilisés pour leurs capacités de reconnaissance moléculaire; ils sont parfois préférés aux anticorps car les aptamères peuvent être conçus et produits synthétiquement complètement in vitro et avec peu ou pas d'immunogénicité.
Explications et mode de fonctionnement
Les aptamères sont des oligonucléotides simple brin de taille comprise entre 70 et 100 nucléotides capables de reconnaître, de manière spécifique et avec une haute affinité, plusieurs types de molécules cibles au moyen d'un repliement tridimensionnel de leur chaîne. Ils sont généralement obtenus en les sélectionnant dans des banques d'oligonucléotides combinatoires, en utilisant la méthode SELEX (Evolution systématique des ligands par enrichissement exponentiel), ce qui permettra de sélectionner cet oligonucléotide dans la bibliothèque utilisée qui se lie avec plus d'affinité à la molécule.
Ces oligonucléotides ont une région centrale de taille variable et avec une séquence aléatoire, et deux régions flanquantes de séquence connue. La longueur totale de l'aptamère varie généralement entre 70 et 100 nucléotides, y compris une séquence fixe ou une première séquence à chaque extrémité de l'aptamère, pour l'amplification PCR (c'est l'une des phases de la méthode de sélection). La longueur de la région centrale avec séquence aléatoire est généralement comprise entre 30 et 60 nucléotides.
Ces molécules sont capables d'adopter des structures globulaires leur permettant de présenter des propriétés de reconnaissance moléculaire complexes et sophistiquées, capables de se lier de manière stable et très spécifique à leurs cibles. Ces cibles moléculaires peuvent être de petites molécules telles que l'ATP, des protéines, des acides nucléiques et des structures multimères complexes.
Il existe également un type d'aptamère basé sur les chaînes peptidiques, conçu pour interagir avec d'autres protéines dans les cellules. Ils consistent en une boucle peptidique variable attachée aux extrémités d'une protéine de support. Cette structure augmente l'affinité de l'aptamère peptidique à des niveaux comparables à ceux des anticorps (gamme nanomolaire).
Découverte et histoire
Les premiers aptamères ont été isolés en 1990 par Larry Gold et Craig Tuerk. Ceux-ci consistaient en des ligands d'ARN qui reconnaissaient l'ADN polymérase du bactériophage T4. Parallèlement et indépendamment à Gold et Tuerk, Andy Ellington et Jack Szostak, à travers ce qu'ils ont appelé la sélection in vitro, ont sélectionné des ligands à base d'ARN contre plusieurs colorants organiques. Le laboratoire de Szostak a inventé le terme aptamère aux oligonucléotides qui interagissaient sélectivement avec les colorants. Deux ans plus tard, la société pharmaceutique Gilead Sciences a breveté la sélection in vitro d'oligonucléotides d'ADN spécifiques de la thrombine, dans le but clair d'utiliser les aptamères obtenus comme produits de traitement et de diagnostic des pathologies humaines.
Depuis la découverte des aptamères, des groupes de recherche du monde entier se sont penchés sur les aptamères. En 2001, le processus SELEX a été automatisé dans le laboratoire dirigé par Andy Ellington de l'Université du Texas à Austin et de SomaLogic, Inc. (Bouder, CO), réduisant ainsi la durée des cycles de sélection de six semaines à seulement trois jours.
Ces dernières années, le concept d '"aptamères intelligents" ou de "ligands intelligents" a été introduit. Ceci définit les aptamères sélectionnés avec des paramètres de réaction de molécule cible prédéfinis, tels que :
- Constante d'équilibre (Kd).
- Constantes de vitesse (koff, kon).
- Paramètres thermodynamiques (ΔH, ΔS).
En 2002, des aptamères naturels ont été découverts dans des organismes vivants. Deux groupes de recherche dirigés par Ronald Breaker et Evgeny Nudler ont découvert un régulateur génétique basé sur les acides nucléiques. Ces structures ont été appelées riboswitches et sont formées par une plate-forme d'expression et un aptamère par lequel le riboswitch interagit avec une molécule cible affectant l'activité du gène.
Enrichissement exponentiel en présence du ligand
La méthode SELEX de son acronyme en anglais "évolution systématique des ligands par enrichissement exponentiel", également appelée sélection in vitro ou évolution in vitro, est la technique utilisée pour la sélection et l'isolement des aptamères. Il s'agit d'une technique de chimie combinatoire appliquée à la biologie moléculaire.
Le processus commence par la synthèse d'une grande bibliothèque d'oligonucléotides, constitués de séquences d'ADN ou d'ARN générées de manière aléatoire et avec un nombre de nucléotides constant, comme mentionné ci-dessus, dans lesquelles toutes les séquences ont des extrémités constantes. Pour une région randomisée de n nucléotides, le nombre de séquences possibles est de 4n.
Les séquences de la banque sont placées en présence du ligand cible. Ensuite, au moyen d'un repliement tridimensionnel, les différents oligonucléotides peuvent se lier ou non à la cible. Les séquences non liées sont éliminées.
Les séquences liées sont élues et amplifiées par PCR (via les amorces d'extrémité) pour la préparation de cycles de sélection ultérieurs dans lesquels la rigueur des conditions sera augmentée pour identifier les séquences possédant la meilleure propriété chimique recherchée (affinité, inactivation). de la protéine, modifications de la conformation pour la détermination, etc.).
Applications
Ces molécules sont présentées comme une réelle alternative aux anticorps monoclonaux dans la recherche biomédicale, tant dans le domaine du diagnostic, du fait de leur application en tant que capteurs moléculaires, que dans celui de la thérapeutique, puisqu'elles sont capables d'interférer dans les fonctions biologiques des molécules. Diana En ce qui concerne les propriétés chimiques et biologiques, leur utilisation contre les anticorps monoclonaux présente plusieurs avantages, tels que leur potentiel à obtenir contre les protéines non immunogènes, leur capacité de régénération, leur stabilité à la température ambiante, la possibilité de modifier leur structure. (pégylation, etc.), la réduction de leurs coûts de production, car ils peuvent être obtenus par synthèse chimique, en évitant l'utilisation d'animaux ou de cellules, leur grande reproductibilité et leur capacité de marquage. Des applications ont également été détectées en tant qu'outils de séparation analytique.
Les pionniers espagnols dans la sélection des aptamères par enrichissement exponentiel en présence du ligand sont le groupe de recherche dirigé par le Dr Víctor M. González de l'Irycis (Institut de recherche en santé Ramón y Cajal), qui ont mis au point des aptamères contre des protéines telles que KMP-11 et les histones H2A et H3 de leishmania infantum. Son savoir-faire a conduit à un accord avec la société APTUS Biotech, pour le développement d'un service à la demande pour la sélection et la production d'aptamères.
Les recherches menées depuis la découverte d'aptamères ont conduit à l'approbation par la FDA du premier médicament à base d'aptamère destiné au traitement de la dégénérescence maculaire néovasculaire liée à l'âge. Ceci est connu sous le nom de Macugen (Pegaptanib), commercialisé en Espagne par Pfizer. La molécule est un oligonucléotide pégylé qui se lie avec une spécificité et une affinité élevées au facteur de croissance de l'endothélium vasculaire extracellulaire (VEGF165), en inhibant son activité. Par ailleurs, la société américaine basée à Cambridge, dans le Massachusetts, Archemix a fondé toute sa recherche et son développement sur le développement d'aptamères en tant que nouveau type de traitement direct pour la prévention et le traitement des maladies chroniques et aiguës. ARC1779, sa molécule vedette, est un antagoniste du facteur de von Willebrand et fait actuellement l'objet d'une étude de phase 2 chez des patients chez qui un syndrome coronarien aigu a été diagnostiqué et qui ont subi une angioplastie coronaire.
En raison des attentes créées par la recherche sur ces molécules, le laboratoire Ellington a développé une base de données pour la sélection in vitro et les tests SELEX, appelée The Aptamer Database, qui répertorie toutes les expériences publiées.
Les aptamères ont une série de caractéristiques particulières, telles que la demi-vie dans le sang de ces molécules non modifiées est très courte, en raison de leur dégradation rapide par les nucléases très abondantes dans ce milieu. De plus, en raison de leur faible poids moléculaire, ils sont facilement éliminés du corps par les reins, ce qui diminue leur toxicité. Bien que cela soit également dû à cela, les applications actuelles de ces aptamères non modifiés se concentrent sur le traitement d'états transitoires, tels que la coagulation sanguine, ou le traitement localisé d'organes, tels que les yeux, lorsque l'accouchement est possible. Un exemple de ceci est un aptamère dont la cible est la ténascine, développé par Schering AG et NeXstar Pharmaceuticals en tant que marqueur d'imagerie tumorale du cancer.
Au cours des dernières années, il a considérablement amélioré son application intracellulaire, ainsi que sa résistance aux nucléases, par des modifications telles que la position 2'du sucre.
D'autres exemples d'aptamères sélectionnés contre les protéines cibles identifiées dans la littérature sont l'aptamère contre la transcriptase inverse du VIH-1, l'aptamère contre l'ARN polymérase dépendante de l'ARN du virus de l'hépatite C, contre la protéine gp 120 du VIH-1, aptamère contre la protéine NS3 du virus de l'hépatite.
Synonymes, antonymes
Voir tous les synonymes pour "aptamère".2 synonymes (sens proche) de "aptamère" :
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à APTAMÈRE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot APTAMERE est dans la page 7 des mots en A du lexique du dictionnaire.
Mots en A à proximité
appétitifappétition appressorium apprimé approche écosystémique aptamèreaptère aptérie aptérygote apyrène apyrexie
En rapport avec "aptamère"
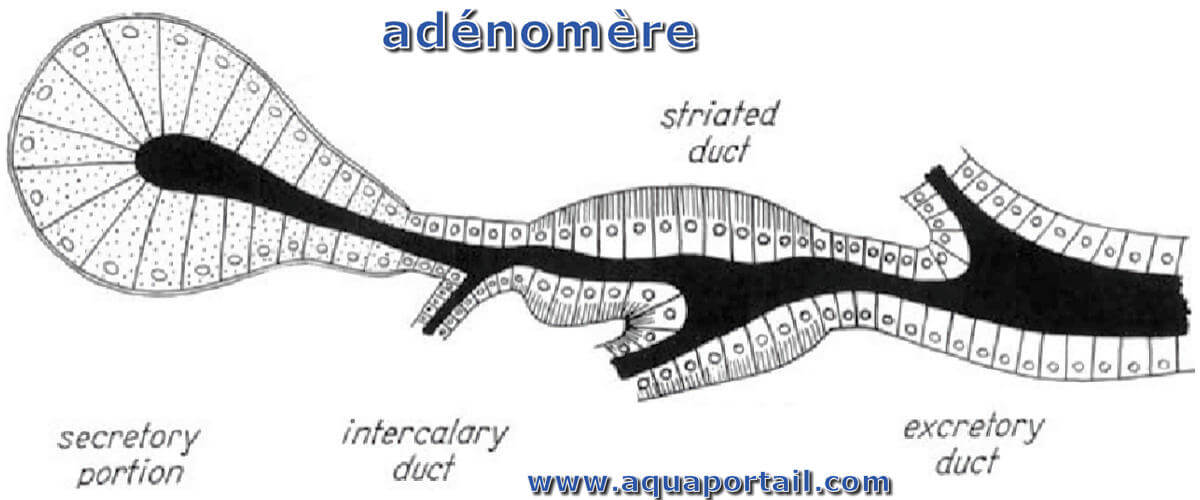
Un adénomère est une structure formée de cellules épithéliales qui sont disposées de manière caractéristique dans chaque glande.
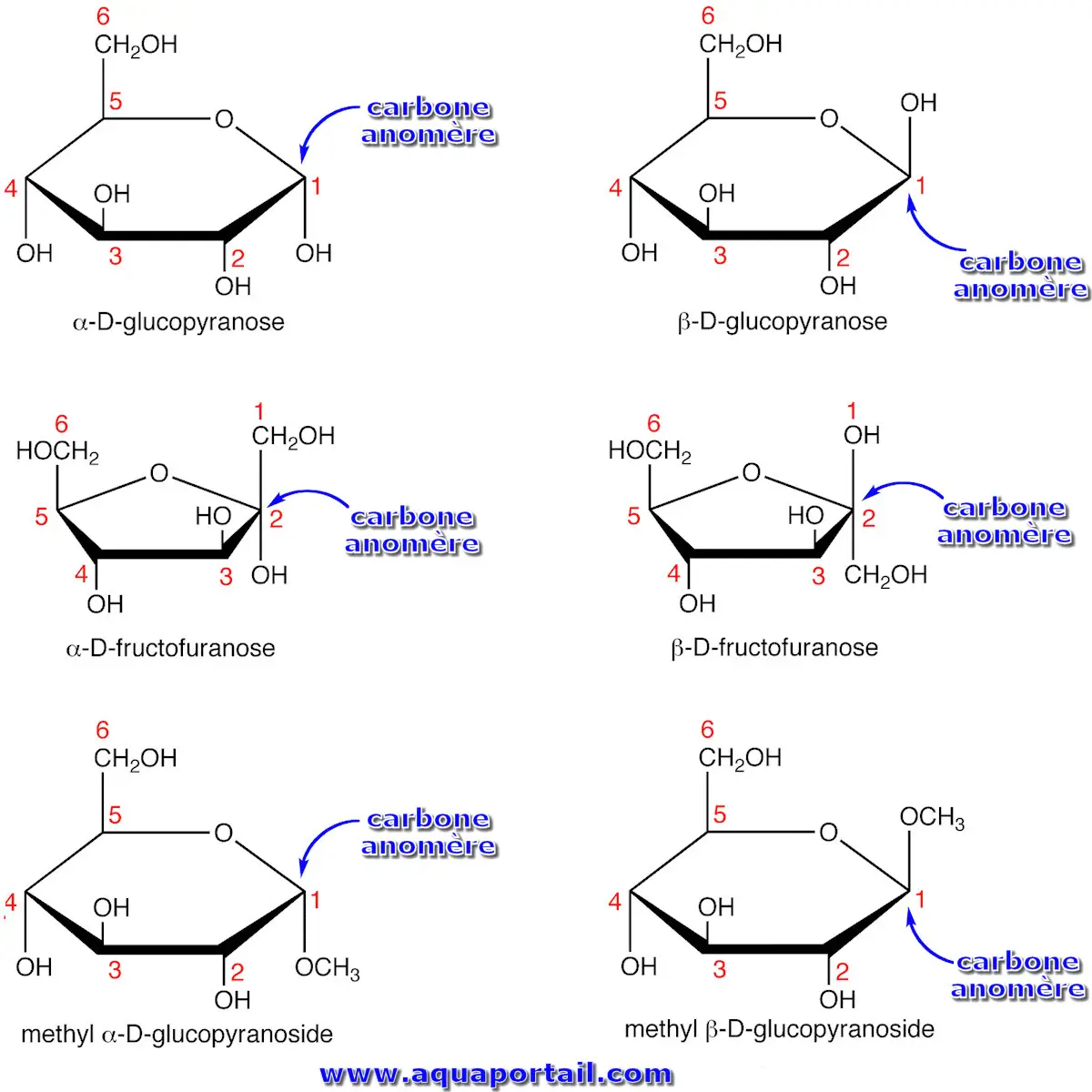
Un anomère est un type de stéréoisomère qui diffère par sa configuration au niveau du carbone hémiacétal ou acétal.
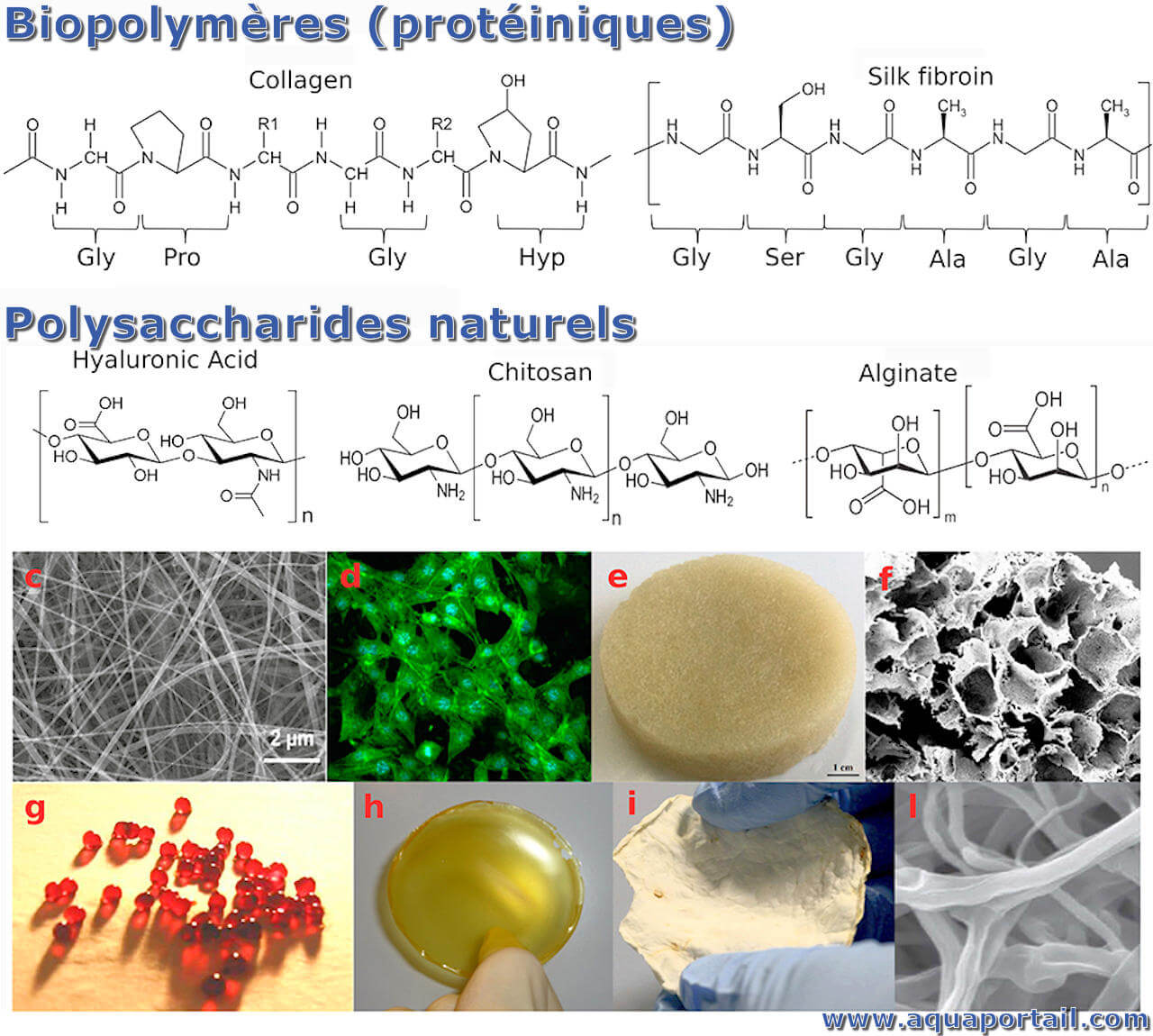
Un biopolymère est une macromolécule biologique qui résulte de la polymérisation de molécules plus simples ou monomères.
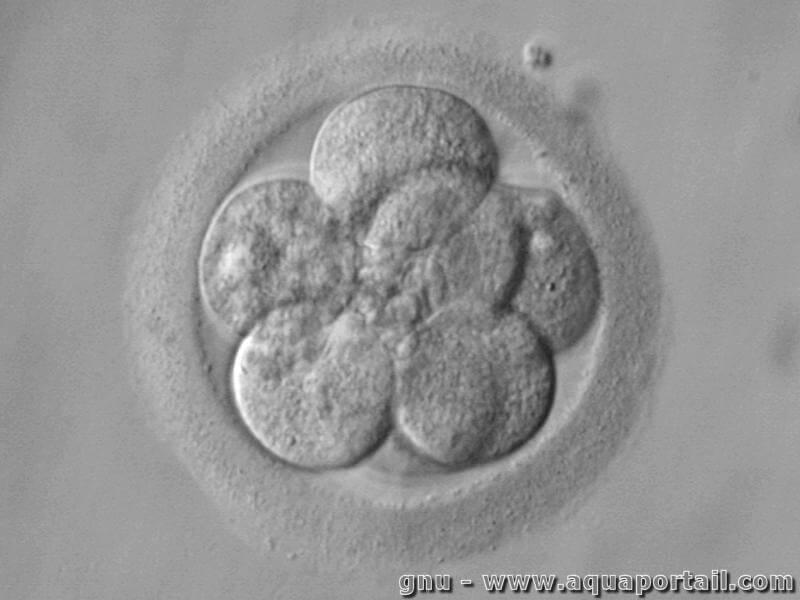
Un blastomère est chacune des cellules issues de la segmentation d'un zygote, provenant des premières divisions cellulaires (clivages) de l'oeuf fécondé.
