Recombinaison homologue
Définition
La recombinaison homologue est une voie de réparation de la rupture double brin de l'ADN qui protège le génome de l'instabilité chromosomique. Ce processus évolutif fondamental est essentiel pour la plasticité du génome. La recombinaison homologue participe au maintien de la stabilité du génome tout en facilitant la diversité génétique.
La recombinaison homologue :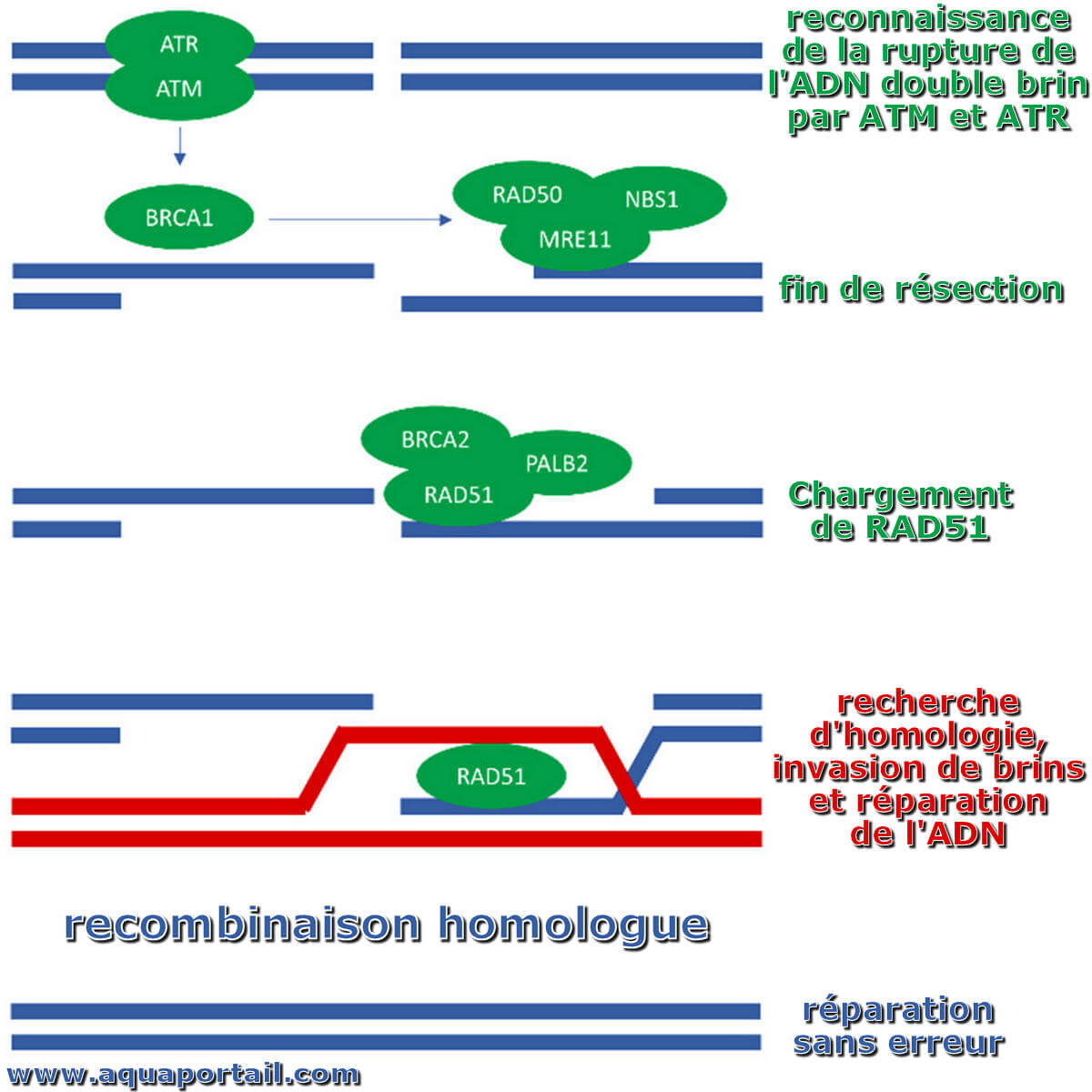
Réparation par recombinaison homologue (RH). La recombinaison homologue commence par la reconnaissance des cassures d'ADN double brin par ATM et ATR. ATM et ATR activent BRCA1, qui joue un rôle clé dans le recrutement des protéines de réparation nécessaires à la résection terminale de l'ADN. La résection de l'extrémité de l'ADN génère une longue queue d'ADN simple brin 3 'qui peut envahir le brin d'ADN homologue (chromatide soeur). Après résection finale, RAD51 est chargé sur la queue d'ADN simple brin à l'aide de BRCA2 et PALB2. Le brin envahit alors le brin d'ADN homologue où la réparation réelle de l'ADN est effectuée. Puisqu'une chromatide soeur est utilisée comme matrice pour la réparation de l'ADN, la RH est considérée comme un processus relativement sans erreur. Il convient de noter qu'en plus des protéines décrites ici, de nombreuses autres protéines sont impliquées dans les RH.
Explications
La recombinaison homologue est un type de recombinaison génétique dans lequel des séquences de nucléotides sont échangées entre deux molécules d'ADN similaires ou identiques. Elle est une forme de réparation de l'ADN.
La recombinaison homologue permet à la cellule d'accéder et de copier des informations de séquence d'ADN intactes en trans, en particulier pour réparer les dommages à l'ADN affectant les deux brins de la double hélice. Ici, nous discutons des transactions d'ADN et des activités enzymatiques nécessaires à ce processus élégamment orchestré dans le contexte de la réparation des cassures double brin de l'ADN dans les cellules somatiques. Cela comprend la recherche d'homologie, l'invasion de brins d'ADN, la synthèse d'ADN de réparation et la restauration de chromosomes intacts. Les aspects de la topologie de l'ADN affectant les étapes individuelles sont mis en évidence. Dans l'ensemble, la recombinaison est une voie dynamique avec de multiples intermédiaires métastables et réversibles conçus pour obtenir une réparation de l'ADN avec une grande fidélité.
La recombinaison homologue (RH) est essentielle pour accéder à la redondance de l'information génétique qui existe sous la forme de chromatides soeurs ou de chromosomes homologues lorsque les deux brins de la double hélice d'ADN sont compromis. Les contextes biologiques importants de la RH sont le support de la réplication de l'ADN et la réparation des cassures double brin de l'ADN dans les cellules somatiques et pendant la méiose. La réparation des doubles brins par recombinaison homologue dans les cellules somatiques illustre la biochimie des enzymes impliquées dans les étapes individuelles.
Rôles
Les rôles de la recombinaison homologue sont dans la réparation des cassures double brin (DSB) de l'ADN, la réactivation des fourches de réplication arrêtées et leurs conséquences (sensibilité aux radiations, méiose et application à la manipulation du génome) sont discutés. La recombinaison homologue est en concurrence avec d'autres voies pour la réparation double brin. Ainsi, le choix de la voie de réparation double brin se déroule en deux étapes :
- résection d'ADN simple brin versus jonction canonique d'extrémités non homologues;
- jonction alternative d'extrémités versus la recombinaison homologue sur ADN réséqué.
La recombinaison homologue (RH) est une épée à double tranchant qui favorise le maintien de la stabilité du génome mais peut générer une instabilité du génome. Les situations d'instabilité génétique induite par les recombinaisons homologues et les stratégies développées pour se protéger contre l'excès de RH sont : la régulation au cours du cycle cellulaire (la RH est active en phase S/G2 lorsque les chromatides soeurs sont présentes), la détoxification des intermédiaires RH abortifs, et la répression de l'initiation des RH. Ensuite, la dérégulation des RH dans les tumeurs et les stratégies anticancéreuses ciblant les RH sont discutées.
Le rôle des recombinaisons homologues dans l'évolution moléculaire des familles multigéniques est capital dans le processus d'évolution concertée.
Mécanismes de réparation
La recombinaison homologue (RH) est une voie mécaniquement conservée qui assure le maintien de l'intégrité génomique. Au cours de la méiose, la RH entraîne des événements de croisement d'ADN entre chromosomes homologues qui produisent la diversité génétique inhérente aux cellules germinales. La connexion physique établie entre les homologues lors de l'événement de croisement est essentielle pour faciliter une ségrégation correcte des chromosomes. La RH est également impliquée dans le maintien de la stabilité génomique des cellules somatiques en restaurant la réplication après qu'une fourche de réplication bloquée a rencontré une lésion de l'ADN ou une rupture de brin, ainsi qu'en suivant des stress exogènes tels que les rayonnements ionisants qui induisent des ruptures double brin de l'ADN. L'importance de la RH peut être mesurée par la conservation des gènes et des fonctions RH de la bactérie à l'homme (eucaryote).
Transformation médiée par les organites
La transformation d'organite médiée par la recombinaison homologue offre la possibilité de transférer le transgène dans une position présélectionnée dans le génome de l'hôte. Les vecteurs de transformation des chloroplastes exploitent deux séquences de plastes, qui flanquent le transgène; la fonction de l'ADN flanquant est de favoriser la recombinaison homologue à un locus prédéterminé dans le génome du chloroplaste, une fois que le vecteur a été délivré dans le protoplaste hôte soit par bombardement de particules, soit après traitement au PEG. Il a été démontré que les plantes transformées par des plastes expriment des transgènes à un niveau élevé grâce au nombre élevé de copies de plastes présents dans les cellules végétales.
Recombinaison non-homologue
La recombinaison non-homologue, ou la jonction d'extrémités non homologues (en anglais, non-homologous end joining, NHEJ), est l'une des façons de réparer les cassures double brin de l'ADN. Ce processus est dit non homologue car les extrémités endommagées de la chaîne sont reliées directement par une ligase, sans nécessiter de matrice homologue, contrairement au processus de recombinaison homologue. La recombinaison non-homologue est nettement moins précise que la recombinaison homologue et entraîne souvent une perte de nucléotides, des translocations ou fusions des télomères, tandis que les deux derniers peuvent être des signes d'une cellule tumorale. La recombinaison non-homologue existe chez des représentants de tous les règnes de la nature. De plus, dans les cellules de mammifères, il sert de principal moyen de réparation des cassures double brin.
Recombinaison homologue vs. non-homologue :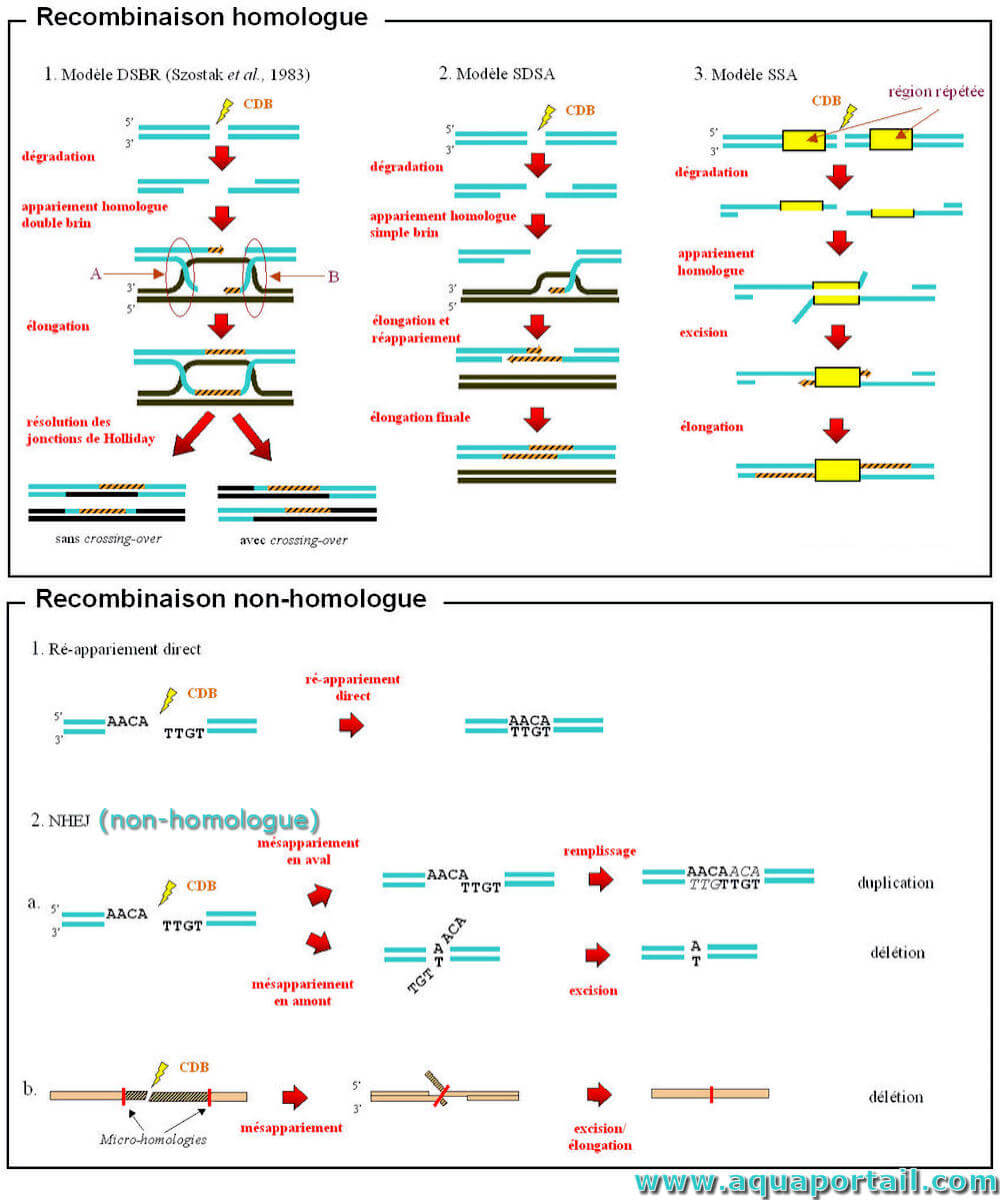
Comparaison entre les mécanismes de recombinaison homologue et ceux de recombinaison non-homologue.
Le choix entre la réparation homologue et non homologue des dommages à l'ADN est régulé au stade initial de la recombinaison, le découpage des régions saillantes simple brin. Les extrémités non coupées peuvent être jointes par la recombinaison non-homologue, et l'élimination même de quelques nucléotides pendant l'étape de coupe supprime la recombinaison non-homologue, et la réparation est effectuée via une recombinaison homologue. La recombinaison non-homologue peut survenir tout au long du cycle cellulaire, mais est le plus actif dans la phase G1 lorsqu'il n'y a pas de matrices disponibles pour la recombinaison homologue. Un rôle important dans la régulation de la recombinaison non-homologue est joué par la kinase cycline-dépendante (cdk1; cdc28 chez la levure), qui est désactivée en phase G1 et exprimée en phases S et G2. Cdk1 phosphoryle la nucléase Sae2, ce qui déclenche la coupe des extrémités.
Le déclenchement de la voie de recombinaison non-homologue commence par le recrutement de la protéine 53BP1 [en] dans la zone endommagée, ce qui favorise une réparation supplémentaire de la cassure double brin le long de la voie de recombinaison non-homologue. Jusqu'au moment de couper les extrémités, le passage à la recombinaison homologue est possible, ce qui est obtenu en attirant la protéine antagoniste 53BP1 - BRCA1 vers la zone endommagée. Si BRCA1 déplace 53BP1, alors la cassure double brin sera réparée par recombinaison homologue. En plus de 53BP1 et BRCA1, RIF1 et la nucléase CtIP impliqué dans la coupe terminale aux premières étapes de la recombinaison homologue. Ainsi, 53BP1 et RIF1 dirigent la réduction le long de la voie de jonction des extrémités non homologues, tandis que BRCA1 et CtIP dirigent la réduction le long de la voie de recombinaison homologue.
La recombinaison homologue pour réparer les cassures double brin ne peut être utilisée que dans les phases S et G2, lorsqu'une matrice de réparation apparaît à la suite de la duplication de l'ADN (par conséquent, la recombinaison non-homologue, qui est active pendant tout le cycle cellulaire, est le principal mécanisme de réparation des cassures double brin dans les cellules de mammifères). Les exceptions sont les régions du génome qui contiennent des répétitions, telles que les répétitions de gènes codant pour l'ARNr (ADNr). Dans l'ADNr, la matrice de réparation d'une cassure double-brin dans la répétition est disponible tout au long du cycle cellulaire; il peut s'agir de n'importe quelle autre répétition. Dans le cas de l'ADNr, les dommages mineurs sont rapidement réparés par recombinaison non-homologue à l'intérieur du nucléole(le temps d'écoulement d'une recombinaison non-homologue est d'environ 30 minutes et la recombinaison homologue est d'environ 7 heures), et des lésions volumineuses et complexes se déplacent avec les protéines des centres fibrillaires et le composant fibrillaire dense vers la périphérie, formant ce que l'on appelle la coiffe nucléolaire. Dans la calotte nucléolaire, toutes les étapes sauf les toutes premières de recombinaison homologue interviennent, avec l'approche des répétitions d'ADNr, ce qui favorise la recombinaison.
La recombinaison non-homologue n'intervient pas dans les calottes nucléolaires. Le choix d'une voie de réparation de rupture double brin est également influencé par la complexité de l'endommagement. La recombinaison non-homologue est généralement utilisée pour réparer des lésions mineures.
Synonymes, antonymes
Voir tous les synonymes pour "recombinaison homologue".0 synonyme (sens proche) pour "recombinaison homologue".
1 antonyme (sens contraire) :
- recombinaison non-homologue
Les mots ou les expressions apparentés à RECOMBINAISON HOMOLOGUE sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
L'expression RECOMBINAISON HOMOLOGUE est dans la page 1 des mots en R du lexique du dictionnaire.
Mots en R à proximité
récifalisterecirculation de l'eau récolte recombinaison recombinaison génétique recombinaison homologuerecombinant reconditionnement récréatif récrétion recristallisation
En rapport avec "recombinaison homologue"

Le brassage génétique correspond aux recombinaisons génétiques au sein d'une population entière et sur plusieurs générations.
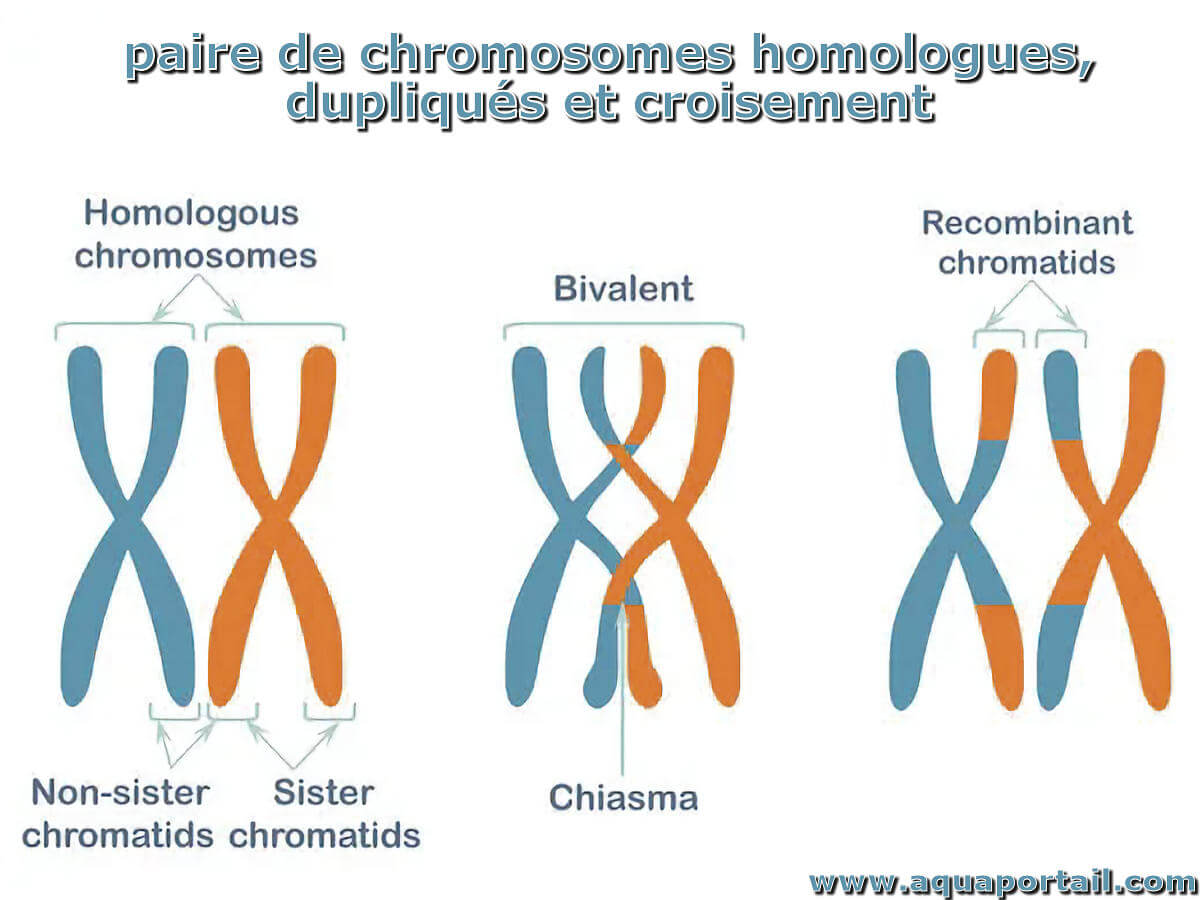
Un homologue, en biologie, indique ce qui a été hérité d'un ancêtre commun (issu de l'homologie).

Deux molécules sont homologues lorsque leurs séquences protéiques ou nucléotidiques présentent des similitudes : même élément pour une position donnée dans...
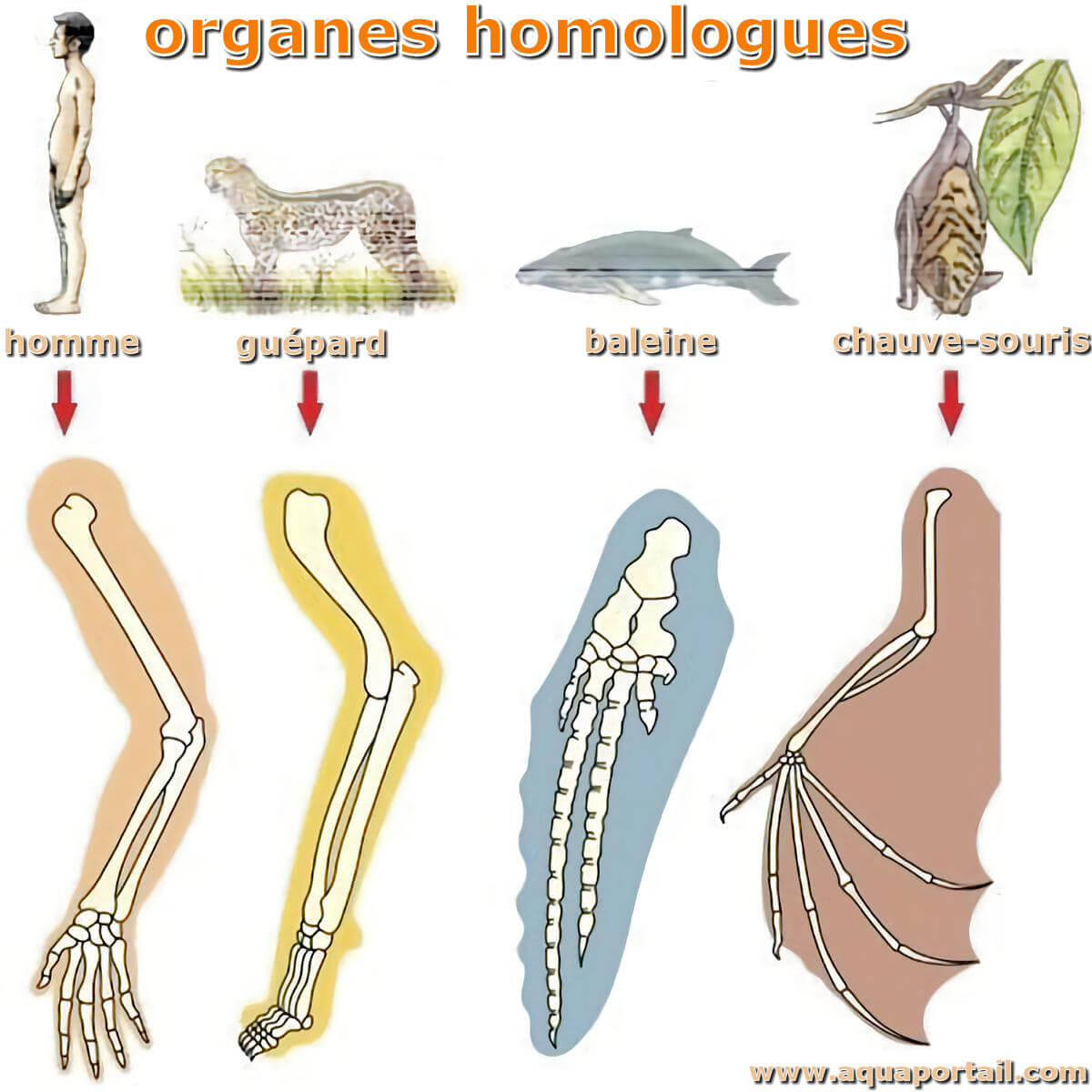
Des organes homologues se présentent par paire, et présentent le même plan d'organisation et les mêmes relations avec les organes voisins.
