Rétrotransposon
Définition
Un rétrotransposon est un transposon dont la séquence présente une homologie avec celle d'un rétrovirus. Il se réplique par transcription inverse d'une copie d'ARN et intègre l'ADN produit dans de nouveaux sites du génome hôte.
Organisation d'un rétrovirus et de rétrotransposons :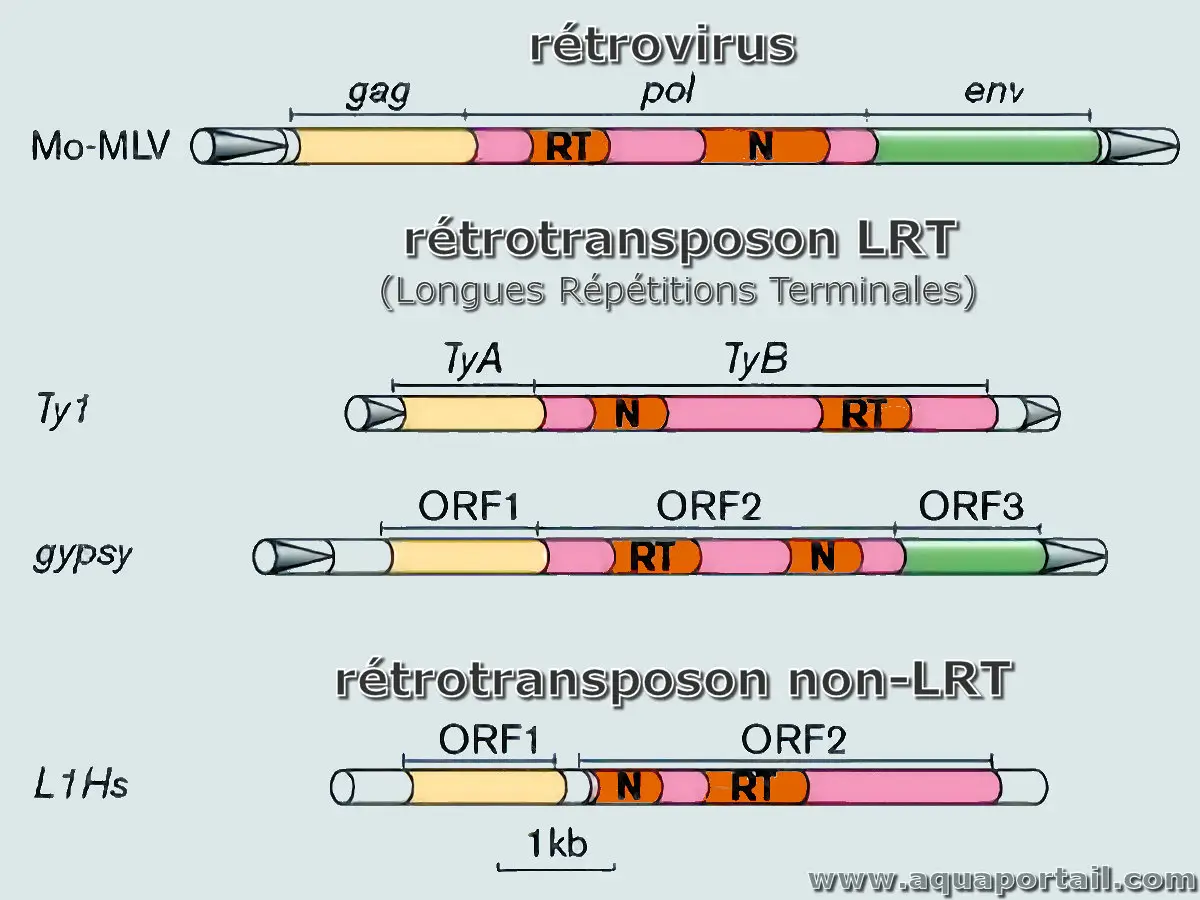
Schéma de l'organisation des génomes du rétrovirus "Moloney murine leucemia virus" (Mo-MLV), des rétrotransposons Ty1 contenant des longues répétitions terminales (LRT) de S. cerevisiae et de D. melanogaster, et des rétrotransposons non-LRT L1H de l'homme. Les rétrovirus et les rétrotransposons à longue répétition terminale (LRT) partagent une organisation structurelle commune. La principale différence entre ces rétroéléments est la présence d'un gène d'enveloppe fonctionnelle (env) dans les rétrovirus, absent ou non fonctionnel dans les rétrotransposons LRT.
Explications
Le rétrotransposon est un élément transposable qui subit une transposition d'un endroit à un autre dans le génome d'une cellule en formant un transcrit d'ARN intermédiaire à partir duquel une copie de l'ADN de l'élément transposable est réalisée à l'aide d'une transcriptase inverse et insérée dans le génome à un nouvel emplacement.
Un rétrotransposon est un élément génétique répandu au cours de l'évolution. Les rétrotransposons comprennent des fractions importantes des génomes des métazoaires.
Les rétrotransposons, souvent simplement appelés rétroposons ou rétroléléments, sont une autre classe d'éléments génétiques mobiles résidant dans le génome de toutes les classes d'organismes. Effectivement, le rétrotransposon est un type d'élément génétique mobile qui utilise un ARN intermédiaire et une transcriptase inverse pour la transposition.
Les rétrotransposons LRT (à longue répétition terminale, LTR en anglais) sont des éléments génétiques mobiles, c'est-à-dire des séquences d'acides nucléiques qui peuvent changer de position au sein d'un génome, voire passer d'une espèce à une autre.
La source de l'activité enzymatique responsable de la transposition des rétrotransposons du type dépourvus de répétitions terminales (LRT) a été identifiée : dans les éléments L1Hs, elle est codée par le deuxième cadre ouvert de lecture et est une nucléase apparentée aux endonucléases apuriniques de réparation.
Rôle dans l'évolution
Les rétrotransposons sont des composants omniprésents de l'ADN de nombreux organismes eucaryotes, notamment celui des plantes. Ils se trouvent chez tous les eucaryotes mais pas chez les procaryotes.
Il existe une corrélation directe entre la taille d'un génome eucaryote et l'abondance mais pas nécessairement le type de rétrotransposons. Par exemple, 3 % du petit génome de levure est composé de rétrotransposons, qui appartiennent tous à la classe LRT. Le génome humain, beaucoup plus vaste,contient plus de 30 % de rétrotransposons, principalement de la classe non-LRT. Enfin, 75 % du génome du maïs, encore plus grand, sont des rétrotransposons, principalement de la classe LRT.
Les rétrotransposons établissent généralement des associations à long terme avec le génome de l'hôte. Cela diffère des transposons dont on pense qu'ils ne sont actifs que pendant une courte période dans n'importe quel génome et qui dépendent de transferts horizontaux entre espèces pour leur survie à long terme. La transmission verticale prédominante (à travers la lignée germinale) des rétrotransposons est la plus prononcée dans les éléments non LRT. Les éléments L1 se sont lentement accumulés tout au long des 100 millions d'années d'histoire du génome des mammifères. Les éléments R2 sont des composants stables du génome des arthropodes depuis plus de 500 millions d'années.
On pense que ces relations stables des rétrotransposons avec le génome hôte ont donné naissance à des stratégies d'insertion spécialisées. Tous les rétrotransposons de la levure s'insèrent soit dans l'hétérochromatine, soit immédiatement en amont des gènes d'ARNt, où ils n'interfèrent pas avec l'expression des gènes de l'hôte. De même, divers rétrotransposons chez les arthropodes s'insèrent à des endroits spécifiques des gènes d'ARNr ou des séquences des télomères de leur hôte.
Classification
Les premiers rétrotransposons découverts présentaient une structure et une capacité de codage protéique similaires à celles des rétrovirus vertébrés, qui contiennent de longues répétitions terminales (LRT). Bien que ces éléments ne contiennent pas d'étape extracellulaire dans leur cycle de vie, tous les aspects de leur rétrotransposition intracellulaire sont similaires à celle des rétrovirus.
Le deuxième type de rétrotransposon découvert avait une structure différente des rétrovirus et codait pour un ensemble différent de protéines. Ce deuxième type ne contenait pas les LRT associés aux rétrovirus et le premier type de rétrotransposon, se terminant plutôt par une simple queue poly(A). Ce deuxième type a été appelé rétrotransposons non viraux, rétrotransposons poly(A), rétroposons et éléments nucléaires intercalés longs (LINE). Compte tenu de l'importance des LRT dans tous les aspects des rétrotranspositions rétrovirales et de la facilité d'identification des LRT, la plupart des auteurs ont appelé ces deux groupes de rétrotransposons les sous-classes LRT et non-LRT. Récemment, cette "sous-classe" a été remplacée par un nouveau terme taxonomique "ordre" dans un système de classification unifié, tel que décrit par Thomas Wicker et ses collègues, pour marquer les différences majeures dans le mécanisme d'insertion, l'organisation globale et l'enzymologie.
Sur la base de ce système, tous les rétrotransposons identifiés jusqu'à présent peuvent être classés en cinq ordres : les rétrotransposons LRT, les éléments de type Dictyostelium à séquence répétée intermédiaire (DIRS), les éléments de type Penelope (PLE), les LINE et les éléments nucléaires intercalés courts (SINE).
Un certain nombre de rétrotransposons LRT ont été récemment découverts et ne peuvent être classés dans aucun des genres ci-dessus en raison de leur manque de séquences codantes. Les TRIM (rétrotransposons à répétition terminale en miniature) sont constitués de petits LRT encadrant un court domaine central de séquences non codantes, tandis que les LARD (dérivés de grands rétrotransposons) sont des dérivés défectueux plus grands qui contiennent un grand domaine interne sans similitudes significatives avec les domaines codants des rétrotransposons.
Rétroéléments des mammifères
La tendance inhérente des rétrotransposons à amplifier leur nombre de copies crée des insertions mutagènes potentiellement nocives pour l'hôte, conduisant à considérer les rétrotransposons comme des parasites génomiques.
Une réponse adaptative de l'hôte encouragerait sélectivement l'évolution de mécanismes répressifs dirigés contre les rétroéléments, ce qui exercerait à son tour une pression sélective sur les rétroéléments pour qu'ils résistent à la répression. Il a été suggéré que l'évolution de la famille APOBEC3 des cytidine désaminases, inhibiteurs cellulaires de la rétrotransposition des rétrotransposons de type LINE, SINE et LRT, pourrait avoir été motivée en partie par une réponse du génome à l'invasion des génomes de mammifères par les rétrotransposons.
En plus de la mutation aléatoire d'éléments actifs dans le génome, d'autres stratégies visant à inhiber la prolifération des rétrotransposons comprennent l'inactivation transcriptionnelle via des modifications épigénétiques de la chromatine et de l'ADN, et l'inactivation post-transcriptionnelle par interférence d'ARN.
De temps en temps, des rétroéléments échappent à la répression et subissent une expansion dans le génome affecté. Ainsi, l'activité continue d'un rétroélément dans le génome, comme celui des rétrovirus endogènes chez la souris, peut indiquer qu'un hôte est en retard dans la course évolutive visant à contrôler l'envahisseur du génome.
Synonymes, antonymes
Voir tous les synonymes pour "rétrotransposon".2 synonymes (sens proche) de "rétrotransposon" :
0 antonyme (sens contraire).
Les mots ou les expressions apparentés à RÉTROTRANSPOSON sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot RETROTRANSPOSON est dans la page 2 des mots en R du lexique du dictionnaire.
Mots en R à proximité
rétrométamorphisme rétromorphose rétroposon rétrotranscriptase rétrotransposition rétrotransposonrétrovirus rétuse réverbération réversion réversion sexuelle
En rapport avec "rétrotransposon"
Un élément génétique mobile, également appelé élément transposable, est un type de matériel génétique en mouvement qui peut traverser à l'intérieur...
La réplication en cercle roulant consiste en la coupure par une protéine circulaire initiatrice de réplication sur un brin parental pour générer l'amorce...

La transcription est un processus de synthèse de l'ARN complémentaire à partir d'une matrice d'ADN.

En génétique, un transposon est un petit segment d'ADN capable de se déplacer d'une région de la molécule d'ADN à une autre région.
