Exon
Définition
L'exon est une région d'un gène eucaryote (constitué de plusieurs parties) qui code pour une protéine. Chaque zone codante est séparée par une portion non codante (intron).
L'exon est la séquence de l'ARN prémessager qui n'est pas excisée lors de la maturation (maturation des ARN). L'ARNm (ARN messager) est donc formé par l'assemblage de tous les exons.
Des exons et introns en transcription :
Les séquences d'ADN qui codent pour les protéines sont appelées exons. Ils sont appelés exons car ils sont exprimés lors de la synthèse des protéines. Lors de l'épissage, les introns sont éliminés tandis que les exons sont réunis, formant un brin d'ARNm. L'enzyme qui effectue l'épissage s'appelle un spliceosome.
Explications
Avec les introns, les exons constituent la partie d'un gène (eucaryote ou archéobactérie) qui est transcrite par l'ARN polymérase au cours du processus de transcription. Les exons, après le processus d'épissage du transcrit primaire, appelé ARNh, se trouvent dans les ARNm matures; parfois, le transcrit primaire peut subir un processus d'épissage alternatif à la suite duquel, dans les ARNm matures, les introns ou des parties de ceux-ci peuvent également être trouvés.
On dit souvent à tort que les exons constituent la partie codante du génome. Ce n'est que partiellement vrai : s'il est vrai que toute la séquence qui code pour une protéine doit résider sur un ou plusieurs exons, il n'est pas toujours vrai qu'un exon code : des exons non codants ont été identifiés dans de nombreux gènes humains. Dans l'ADN des eucaryotes, un gène est composé d'un certain nombre d'exons et d'introns; la taille de ces derniers peut être très différente, même de l'ordre de centaines de milliers de bases.
De nombreuses phases des mécanismes d'élimination des exons lors de la maturation de l'ARNm sont encore inconnues alors que certains passages commencent à être connus : par exemple, les exons sont généralement délimités par deux paires de nucléotides, une GT et une AG, mais cela ne suffit pas. assez pour définir l'exon, il s'agit toujours d'un processus très précis qui doit identifier un petit exon de quelques dizaines ou centaines de bases au sein d'une région chromosomique pouvant représenter des centaines de milliers de bases et aussi si ces mécanismes sont moins, comme cela existe par exemple lorsque les séquences GT et AG subissent une mutation, les exons ne sont pas reconnus de manière appropriée et aboutissent à la production d'un ARN anormal, incapable de produire la protéine normale : c'est ce qui se passe dans nombreux patients atteints de maladies génétiques. L'ARNm mature, chez les eucaryotes, est généralement constitué non seulement par la partie codante (séquence d'ADN codante ou CDS), également par deux régions non traduites (UTR) placées l'une en 5′ et l'autre en 3′ du transcrit. Cela implique donc l'existence d'exons totalement ou partiellement non codants, c'est-à-dire ceux qui formeront les UTR de la transcription mature.
Un ARN nucléaire hétérogène (hnRNA) qui est un transcrit d'ARNm n'est pas encore soumis à une édition d'ARN ou à un pré-ARNm. Les exons peuvent inclure à la fois des séquences codant pour des acides aminés (en rouge) et des séquences non traduites. Les exons et dans certains cas également les introns ou parties d'entre eux sont réunis pour former un ARNm final fonctionnel; en fait, il faut préciser que l'ARNm mature n'est pas toujours composé exclusivement de caractères exoniques, mais peut parfois présenter des portions introniques. L'annotation 5′ et 3′ se réfère à la direction de l'épissage de l'ADN dans le chromosome et est utilisée pour distinguer les deux régions non traduites (UTR). Certains des exons formeront une partie ou la totalité de la région non traduite en 5′ ou la région non traduite en 3′ de chaque transcription. Les régions UTR sont importantes pour l'efficacité de la traduction de la transcription et pour contrôler le taux de traduction et la demi-vie de la transcription.
Les transcrits du même gène peuvent ne pas avoir la même structure exonique, car des parties de l'ARNm peuvent être éliminées par le processus d'épissage alternatif. Certains transcrits d'ARNm ont des exons sans ORF, de sorte qu'ils sont parfois considérés comme des ARN non codants.
Exonisation
L'exonisation est la création d'un nouvel exon, à la suite de mutations dans les séquences introniques. Les messagers polycistroniques ont de multiples cadres de lecture ouverts (ORF) dans le même transcrit et de petites régions de séquences non traduites entre chaque ORF.
En plus de la division déjà mentionnée en exons codants et non codants, les exons peuvent donc être divisés en constituants et en alternatives. Les exons constitutifs d'un gène sont présents dans tous les ARNm produits par ce gène, mais les exons alternatifs se trouvent uniquement dans un sous-ensemble des transcrits issus du gène. Les protéines résultant de la traduction de transcrits alternatifs du même gène sont appelées isoformes.
Remixage
Les exons sont impliqués dans un processus de remixage, un mécanisme qui permet la création de nouveaux gènes. Les preuves qui soutiennent l'existence de ce phénomène sont les suivantes :
- au sein des protéines codées par les gènes, il semble que chaque exon code une unité indépendante de la protéine (domaine);
- de nombreux gènes et les protéines respectives composées d'unités répétées (telles que les immunoglobulines) ont partiellement évolué grâce à la duplication et à la divergence des exons : la présence d'introns entre chaque exon rendrait la duplication plus probable;
- des exons similaires se retrouvent parfois dans des gènes non apparentés, ce qui pourrait indiquer que certains exons ont probablement été réutilisés dans des gènes codant pour différentes protéines (par exemple, le gène du récepteur LDL contient des exons corrélés de façon évolutive avec les exons présents dans les gènes). gène codant pour le précurseur du facteur de croissance épidermique et ceux du gène du complément C9).
La plus grande longueur des introns, comparée à celle des exons, assure la présence plus facile des événements de recombinaison dans les introns, plutôt que le niveau des exons. Par conséquent, les exons sont plus susceptibles d'être redistribués plutôt qu'interrompus.
Nombre d'exons
Le nombre d'exons dans les gènes humains est en moyenne de 9 et est généralement lié à la longueur du gène. Cependant, il existe parfois des variations, comme dans le cas du gène de Titina, caractérisé par un nombre très élevé d'exons (363). Les dimensions des exons internes sont peu variables normalement et en moyenne égales à 122 paires de bases. En revanche, les exons 3′ des gènes peuvent être considérablement plus longs. De plus, tout en considérant les dimensions des exons, nous pouvons affirmer cette variabilité réduite des dimensions, nous ne pouvons en dire autant des dimensions des introns, ce qui peut au contraire être très variable.
Synonymes, antonymes
Voir tous les synonymes pour "exon".0 synonyme (sens proche) pour "exon".
1 antonyme (sens contraire) :
Les mots ou les expressions apparentés à EXON sont des termes qui sont directement liés les uns aux autres par leur signification, générale ou spécifique.
Le mot EXON est dans la page 5 des mots en E du lexique du dictionnaire.
Mots en E à proximité
exogamie exogène exogénote exombrelle exomolécule exonexondation exondé exonique exonisation exonucléase
En rapport avec "exon"
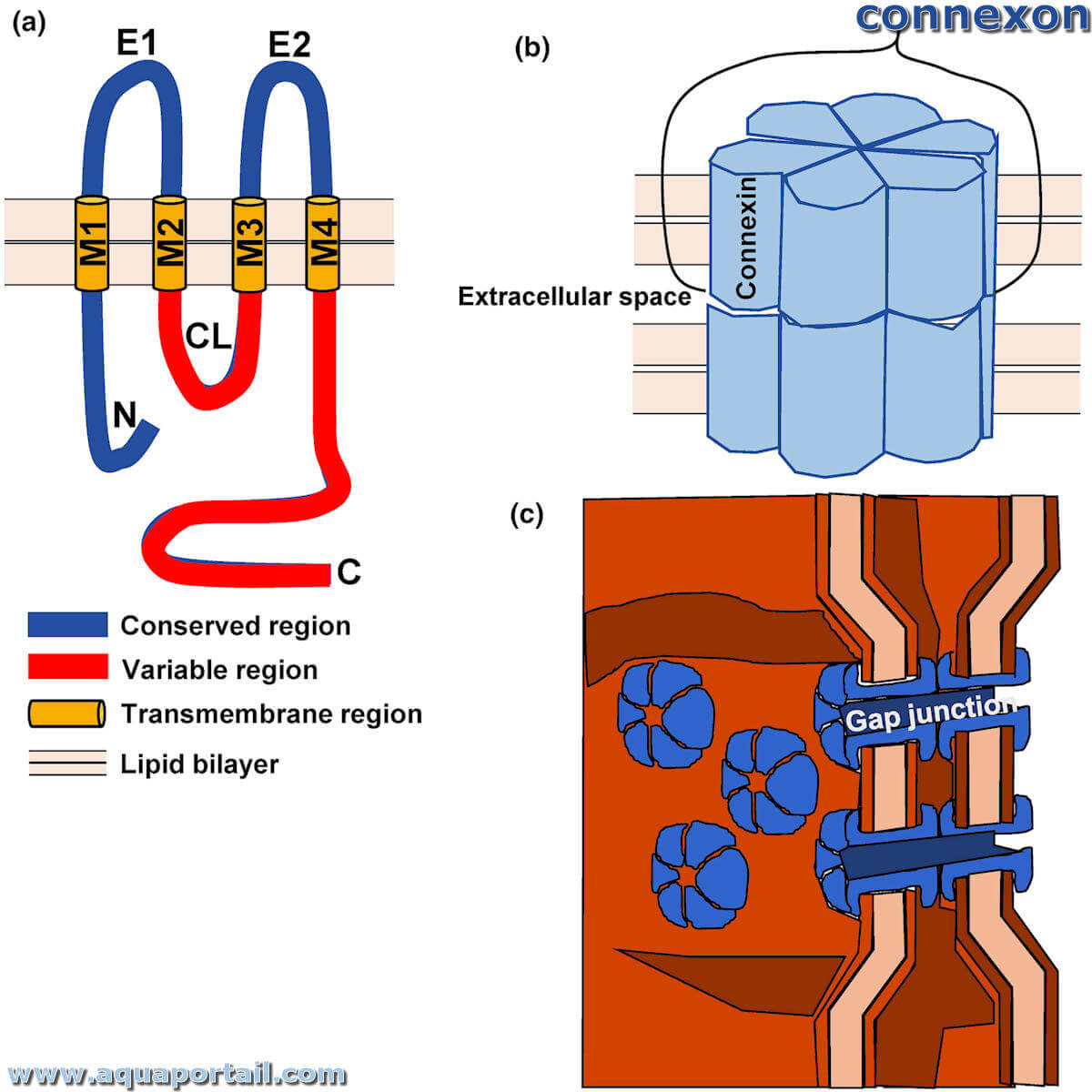
Un connexon est une structure transmembranaire formée de 6 sous-unités protéiques identiques ou connexines chacune formée de 4 hélices α-transmembranaires.
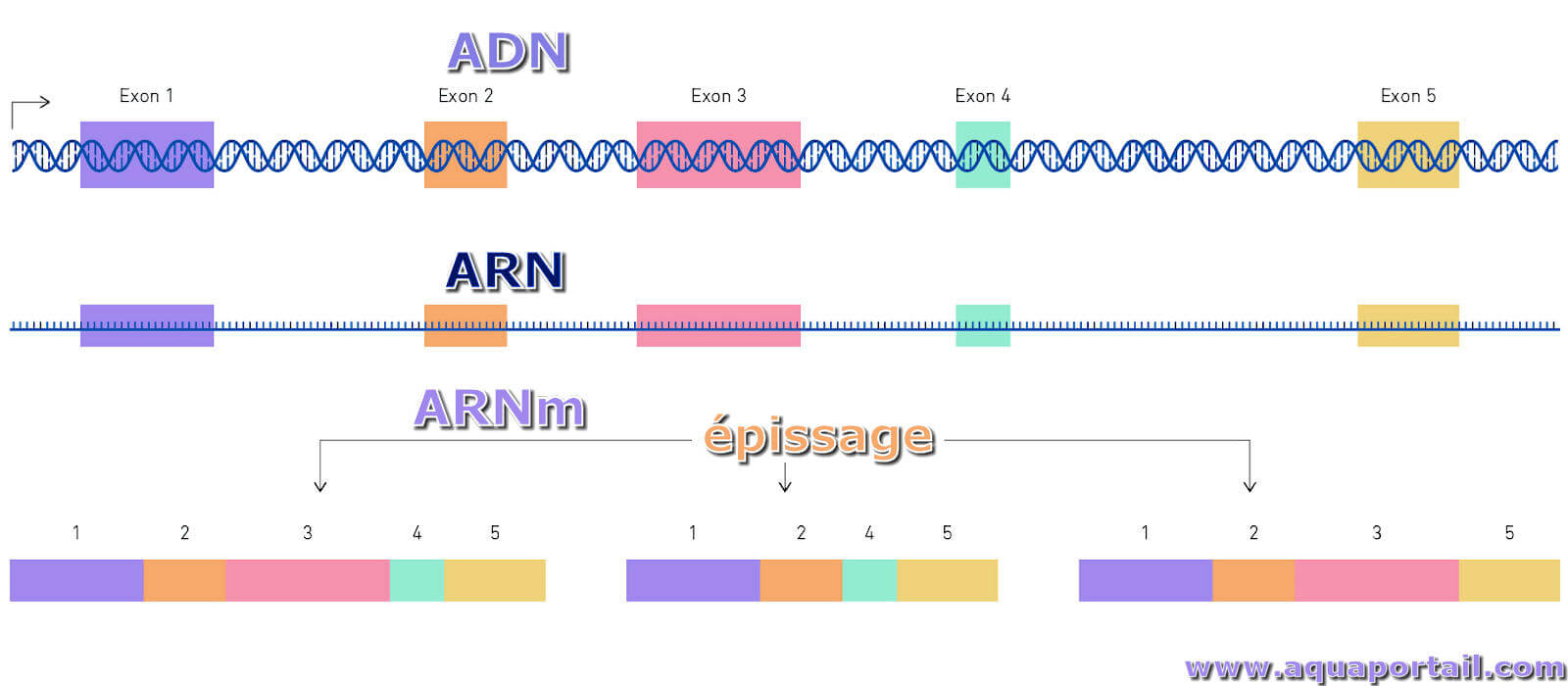
L'épissage est le traitement du transcrit primaire (ARN) d'un gène, aboutissant à la production d'un ARN messager dirigeant la synthèse d'une protéine.

Ce qui est exondé est hors de l'eau, en surface ou au-dessus, tandis qu'une autre partie est immergée.
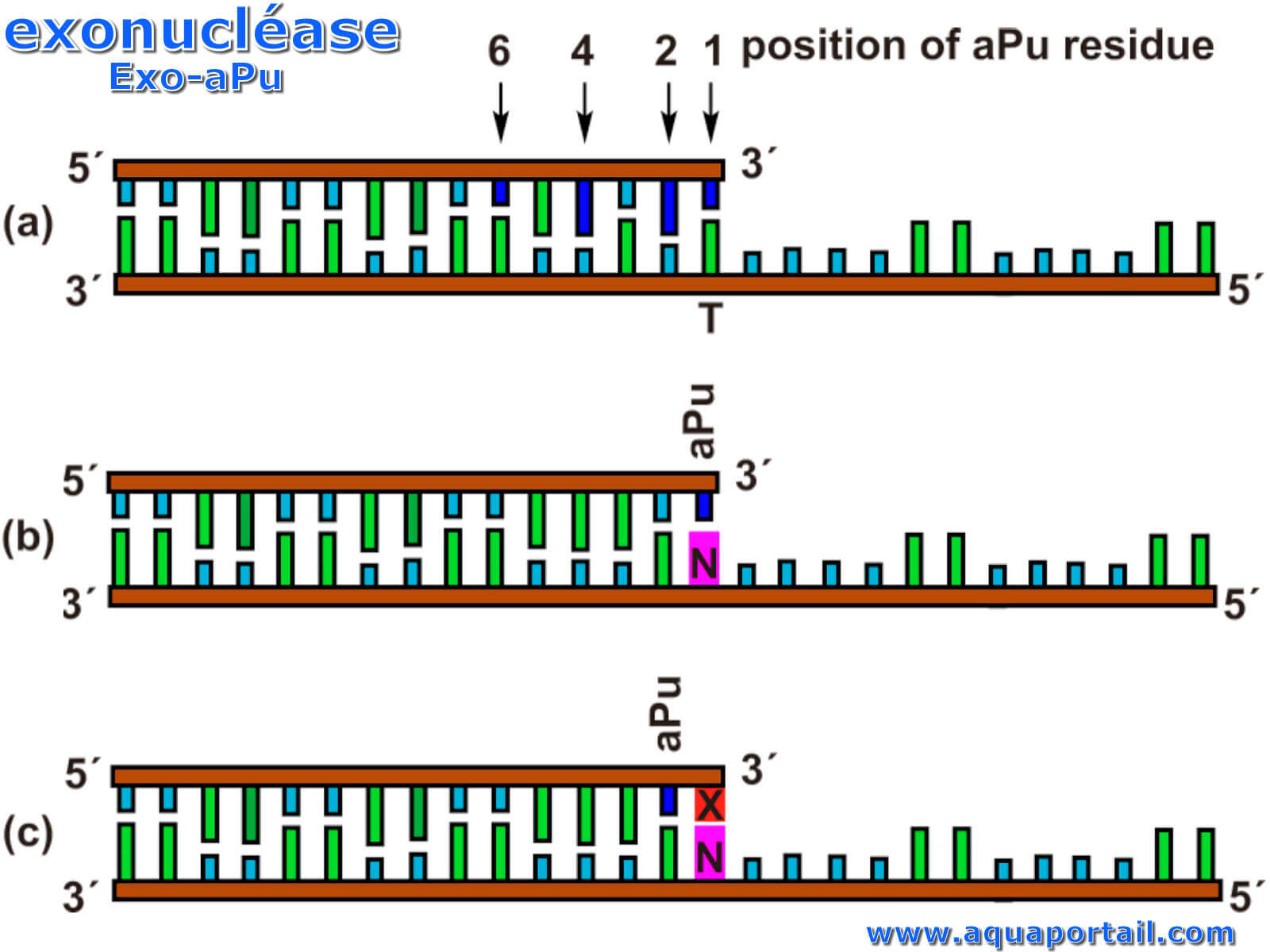
L'exonucléase distingue la partie qui est au-dehors de la nucléase. Les exonucléases sont des enzymes qui fonctionnent en coupant les nucléotides...
